Biopython - BioSQL-Modul
BioSQList ein generisches Datenbankschema, das hauptsächlich zum Speichern von Sequenzen und den zugehörigen Daten für alle RDBMS-Engines entwickelt wurde. Es ist so konzipiert, dass es die Daten aus allen gängigen Bioinformatik-Datenbanken wie GenBank, Swissport usw. enthält. Es kann auch zum Speichern von internen Daten verwendet werden.
BioSQL bietet derzeit ein spezifisches Schema für die folgenden Datenbanken -
- MySQL (biosqldb-mysql.sql)
- PostgreSQL (biosqldb-pg.sql)
- Oracle (biosqldb-ora / *. Sql)
- SQLite (biosqldb-sqlite.sql)
Es bietet auch minimale Unterstützung für Java-basierte HSQLDB- und Derby-Datenbanken.
BioPython bietet sehr einfache, einfache und erweiterte ORM-Funktionen für die Arbeit mit BioSQL-basierten Datenbanken. BioPython provides a module, BioSQL um die folgende Funktionalität zu tun -
- Erstellen / Entfernen einer BioSQL-Datenbank
- Stellen Sie eine Verbindung zu einer BioSQL-Datenbank her
- Analysieren Sie eine Sequenzdatenbank wie GenBank, Swisport, BLAST-Ergebnis, Entrez-Ergebnis usw. und laden Sie sie direkt in die BioSQL-Datenbank
- Rufen Sie die Sequenzdaten aus der BioSQL-Datenbank ab
- Rufen Sie Taxonomiedaten von NCBI BLAST ab und speichern Sie sie in der BioSQL-Datenbank
- Führen Sie eine SQL-Abfrage für die BioSQL-Datenbank aus
Übersicht über das BioSQL-Datenbankschema
Bevor wir uns eingehend mit BioSQL befassen, sollten wir uns mit den Grundlagen des BioSQL-Schemas vertraut machen. Das BioSQL-Schema bietet mehr als 25 Tabellen für Sequenzdaten, Sequenzmerkmale, Sequenzkategorie / Ontologie und Taxonomieinformationen. Einige der wichtigen Tabellen lauten wie folgt:
- biodatabase
- bioentry
- biosequence
- seqfeature
- taxon
- taxon_name
- antology
- term
- dxref
Erstellen einer BioSQL-Datenbank
In diesem Abschnitt erstellen wir eine BioSQL-Beispieldatenbank, biosql, unter Verwendung des vom BioSQL-Team bereitgestellten Schemas. Wir werden mit der SQLite-Datenbank arbeiten, da der Einstieg sehr einfach ist und keine komplexe Einrichtung aufweist.
Hier erstellen wir eine SQLite-basierte BioSQL-Datenbank mit den folgenden Schritten.
Step 1 - Laden Sie die SQLite-Datenbank-Engine herunter und installieren Sie sie.
Step 2 - Laden Sie das BioSQL-Projekt von der GitHub-URL herunter. https://github.com/biosql/biosql
Step 3 - Öffnen Sie eine Konsole, erstellen Sie mit mkdir ein Verzeichnis und geben Sie es ein.
cd /path/to/your/biopython/sample
mkdir sqlite-biosql
cd sqlite-biosqlStep 4 - Führen Sie den folgenden Befehl aus, um eine neue SQLite-Datenbank zu erstellen.
> sqlite3.exe mybiosql.db
SQLite version 3.25.2 2018-09-25 19:08:10
Enter ".help" for usage hints.
sqlite>Step 5 - Kopieren Sie die Datei biosqldb-sqlite.sql aus dem BioSQL-Projekt (/ sql / biosqldb-sqlite.sql`) und speichern Sie sie im aktuellen Verzeichnis.
Step 6 - Führen Sie den folgenden Befehl aus, um alle Tabellen zu erstellen.
sqlite> .read biosqldb-sqlite.sqlJetzt werden alle Tabellen in unserer neuen Datenbank erstellt.
Step 7 - Führen Sie den folgenden Befehl aus, um alle neuen Tabellen in unserer Datenbank anzuzeigen.
sqlite> .headers on
sqlite> .mode column
sqlite> .separator ROW "\n"
sqlite> SELECT name FROM sqlite_master WHERE type = 'table';
biodatabase
taxon
taxon_name
ontology
term
term_synonym
term_dbxref
term_relationship
term_relationship_term
term_path
bioentry
bioentry_relationship
bioentry_path
biosequence
dbxref
dbxref_qualifier_value
bioentry_dbxref
reference
bioentry_reference
comment
bioentry_qualifier_value
seqfeature
seqfeature_relationship
seqfeature_path
seqfeature_qualifier_value
seqfeature_dbxref
location
location_qualifier_value
sqlite>Die ersten drei Befehle sind Konfigurationsbefehle zum Konfigurieren von SQLite, um das Ergebnis formatiert anzuzeigen.
Step 8 - Kopieren Sie die vom BioPython-Team bereitgestellte GenBank-Beispieldatei ls_orchid.gbk https://raw.githubusercontent.com/biopython/biopython/master/Doc/examples/ls_orchid.gbk in das aktuelle Verzeichnis und speichern Sie es als orchid.gbk.
Step 9 - Erstellen Sie ein Python-Skript, load_orchid.py, mit dem folgenden Code und führen Sie es aus.
from Bio import SeqIO
from BioSQL import BioSeqDatabase
import os
server = BioSeqDatabase.open_database(driver = 'sqlite3', db = "orchid.db")
db = server.new_database("orchid")
count = db.load(SeqIO.parse("orchid.gbk", "gb"), True) server.commit()
server.close()Der obige Code analysiert den Datensatz in der Datei, konvertiert ihn in Python-Objekte und fügt ihn in die BioSQL-Datenbank ein. Wir werden den Code im späteren Abschnitt analysieren.
Schließlich haben wir eine neue BioSQL-Datenbank erstellt und einige Beispieldaten in diese geladen. Wir werden die wichtigen Tabellen im nächsten Kapitel diskutieren.
Einfaches ER-Diagramm
biodatabase Die Tabelle befindet sich oben in der Hierarchie und dient hauptsächlich dazu, einen Satz von Sequenzdaten in einer einzelnen Gruppe / virtuellen Datenbank zu organisieren. Every entry in the biodatabase refers to a separate database and it does not mingle with another database. Alle zugehörigen Tabellen in der BioSQL-Datenbank enthalten Verweise auf den Eintrag in die Biodatabase.
bioentryDie Tabelle enthält alle Details zu einer Sequenz mit Ausnahme der Sequenzdaten. Sequenzdaten eines bestimmtenbioentry wird gespeichert in biosequence Tabelle.
taxon und taxon_name sind Taxonomiedetails und jeder Eintrag verweist auf diese Tabelle, um seine Taxoninformationen anzugeben.
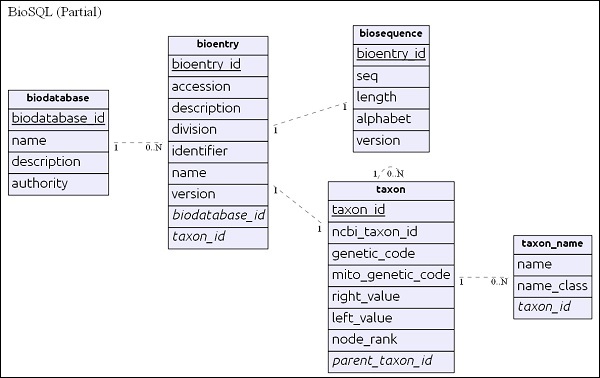
Nachdem wir das Schema verstanden haben, wollen wir uns im nächsten Abschnitt einige Fragen ansehen.
BioSQL-Abfragen
Lassen Sie uns einige SQL-Abfragen untersuchen, um besser zu verstehen, wie die Daten organisiert sind und die Tabellen miteinander in Beziehung stehen. Bevor Sie fortfahren, öffnen wir die Datenbank mit dem folgenden Befehl und legen einige Formatierungsbefehle fest.
> sqlite3 orchid.db
SQLite version 3.25.2 2018-09-25 19:08:10
Enter ".help" for usage hints.
sqlite> .header on
sqlite> .mode columns.header and .mode are formatting options to better visualize the data. Sie können die Abfrage auch mit einem beliebigen SQLite-Editor ausführen.
Listen Sie die im System verfügbare virtuelle Sequenzdatenbank wie folgt auf:
select
*
from
biodatabase;
*** Result ***
sqlite> .width 15 15 15 15
sqlite> select * from biodatabase;
biodatabase_id name authority description
--------------- --------------- --------------- ---------------
1 orchid
sqlite>Hier haben wir nur eine Datenbank, orchid.
Listen Sie die in der Datenbank verfügbaren Einträge (Top 3) auf orchid mit dem unten angegebenen Code
select
be.*,
bd.name
from
bioentry be
inner join
biodatabase bd
on bd.biodatabase_id = be.biodatabase_id
where
bd.name = 'orchid' Limit 1,
3;
*** Result ***
sqlite> .width 15 15 10 10 10 10 10 50 10 10
sqlite> select be.*, bd.name from bioentry be inner join biodatabase bd on
bd.biodatabase_id = be.biodatabase_id where bd.name = 'orchid' Limit 1,3;
bioentry_id biodatabase_id taxon_id name accession identifier division description version name
--------------- --------------- ---------- ---------- ---------- ---------- ----------
---------- ---------- ----------- ---------- --------- ---------- ----------
2 1 19 Z78532 Z78532 2765657 PLN
C.californicum 5.8S rRNA gene and ITS1 and ITS2 DN 1
orchid
3 1 20 Z78531 Z78531 2765656 PLN
C.fasciculatum 5.8S rRNA gene and ITS1 and ITS2 DN 1
orchid
4 1 21 Z78530 Z78530 2765655 PLN
C.margaritaceum 5.8S rRNA gene and ITS1 and ITS2 D 1
orchid
sqlite>Listen Sie die mit einem Eintrag verbundenen Sequenzdetails (Zugang - Z78530, Name - C. fasciculatum 5.8S rRNA-Gen und ITS1- und ITS2-DNA) mit dem angegebenen Code auf -
select
substr(cast(bs.seq as varchar), 0, 10) || '...' as seq,
bs.length,
be.accession,
be.description,
bd.name
from
biosequence bs
inner join
bioentry be
on be.bioentry_id = bs.bioentry_id
inner join
biodatabase bd
on bd.biodatabase_id = be.biodatabase_id
where
bd.name = 'orchid'
and be.accession = 'Z78532';
*** Result ***
sqlite> .width 15 5 10 50 10
sqlite> select substr(cast(bs.seq as varchar), 0, 10) || '...' as seq,
bs.length, be.accession, be.description, bd.name from biosequence bs inner
join bioentry be on be.bioentry_id = bs.bioentry_id inner join biodatabase bd
on bd.biodatabase_id = be.biodatabase_id where bd.name = 'orchid' and
be.accession = 'Z78532';
seq length accession description name
------------ ---------- ---------- ------------ ------------ ---------- ---------- -----------------
CGTAACAAG... 753 Z78532 C.californicum 5.8S rRNA gene and ITS1 and ITS2 DNA orchid
sqlite>Holen Sie sich die vollständige Sequenz, die einem Eintrag zugeordnet ist (Zugang - Z78530, Name - C. fasciculatum 5.8S rRNA-Gen und ITS1- und ITS2-DNA), unter Verwendung des folgenden Codes:
select
bs.seq
from
biosequence bs
inner join
bioentry be
on be.bioentry_id = bs.bioentry_id
inner join
biodatabase bd
on bd.biodatabase_id = be.biodatabase_id
where
bd.name = 'orchid'
and be.accession = 'Z78532';
*** Result ***
sqlite> .width 1000
sqlite> select bs.seq from biosequence bs inner join bioentry be on
be.bioentry_id = bs.bioentry_id inner join biodatabase bd on bd.biodatabase_id =
be.biodatabase_id where bd.name = 'orchid' and be.accession = 'Z78532';
seq
----------------------------------------------------------------------------------------
----------------------------
CGTAACAAGGTTTCCGTAGGTGAACCTGCGGAAGGATCATTGTTGAGACAACAGAATATATGATCGAGTGAATCT
GGAGGACCTGTGGTAACTCAGCTCGTCGTGGCACTGCTTTTGTCGTGACCCTGCTTTGTTGTTGGGCCTCC
TCAAGAGCTTTCATGGCAGGTTTGAACTTTAGTACGGTGCAGTTTGCGCCAAGTCATATAAAGCATCACTGATGAATGACATTATTGT
CAGAAAAAATCAGAGGGGCAGTATGCTACTGAGCATGCCAGTGAATTTTTATGACTCTCGCAACGGATATCTTGGCTC
TAACATCGATGAAGAACGCAG
sqlite>Liste Taxon mit Bio-Datenbank, Orchidee verbunden
select distinct
tn.name
from
biodatabase d
inner join
bioentry e
on e.biodatabase_id = d.biodatabase_id
inner join
taxon t
on t.taxon_id = e.taxon_id
inner join
taxon_name tn
on tn.taxon_id = t.taxon_id
where
d.name = 'orchid' limit 10;
*** Result ***
sqlite> select distinct tn.name from biodatabase d inner join bioentry e on
e.biodatabase_id = d.biodatabase_id inner join taxon t on t.taxon_id =
e.taxon_id inner join taxon_name tn on tn.taxon_id = t.taxon_id where d.name =
'orchid' limit 10;
name
------------------------------
Cypripedium irapeanum
Cypripedium californicum
Cypripedium fasciculatum
Cypripedium margaritaceum
Cypripedium lichiangense
Cypripedium yatabeanum
Cypripedium guttatum
Cypripedium acaule
pink lady's slipper
Cypripedium formosanum
sqlite>Laden Sie Daten in die BioSQL-Datenbank
In diesem Kapitel erfahren Sie, wie Sie Sequenzdaten in die BioSQL-Datenbank laden. Wir haben bereits den Code zum Laden von Daten in die Datenbank im vorherigen Abschnitt und der Code lautet wie folgt:
from Bio import SeqIO
from BioSQL import BioSeqDatabase
import os
server = BioSeqDatabase.open_database(driver = 'sqlite3', db = "orchid.db")
DBSCHEMA = "biosqldb-sqlite.sql"
SQL_FILE = os.path.join(os.getcwd(), DBSCHEMA)
server.load_database_sql(SQL_FILE)
server.commit()
db = server.new_database("orchid")
count = db.load(SeqIO.parse("orchid.gbk", "gb"), True) server.commit()
server.close()Wir werden uns jede Zeile des Codes und seinen Zweck genauer ansehen -
Line 1 - Lädt das SeqIO-Modul.
Line 2- Lädt das BioSeqDatabase-Modul. Dieses Modul bietet alle Funktionen für die Interaktion mit der BioSQL-Datenbank.
Line 3 - Lädt das OS-Modul.
Line 5- open_database öffnet die angegebene Datenbank (db) mit dem konfigurierten Treiber (Treiber) und gibt ein Handle an die BioSQL-Datenbank (Server) zurück. Biopython unterstützt SQLite-, MySQL-, Postgresql- und Oracle-Datenbanken.
Line 6-10- Die Methode load_database_sql lädt die SQL aus der externen Datei und führt sie aus. Die Commit-Methode schreibt die Transaktion fest. Wir können diesen Schritt überspringen, da wir die Datenbank bereits mit Schema erstellt haben.
Line 12 - new_database-Methoden erstellen eine neue virtuelle Datenbank, Orchidee und geben eine Handle-Datenbank zurück, um den Befehl für die Orchideendatenbank auszuführen.
Line 13- Die Methode load lädt die Sequenzeinträge (iterable SeqRecord) in die Orchideendatenbank. SqlIO.parse analysiert die GenBank-Datenbank und gibt alle darin enthaltenen Sequenzen als iterierbaren SeqRecord zurück. Der zweite Parameter (True) der Lademethode weist sie an, die Taxonomiedetails der Sequenzdaten von der NCBI-Explosionswebsite abzurufen, sofern diese nicht bereits im System verfügbar sind.
Line 14 - Commit schreibt die Transaktion fest.
Line 15 - close schließt die Datenbankverbindung und zerstört das Serverhandle.
Holen Sie sich die Sequenzdaten
Lassen Sie uns eine Sequenz mit der Kennung 2765658 aus der Orchideendatenbank wie folgt abrufen -
from BioSQL import BioSeqDatabase
server = BioSeqDatabase.open_database(driver = 'sqlite3', db = "orchid.db")
db = server["orchid"]
seq_record = db.lookup(gi = 2765658)
print(seq_record.id, seq_record.description[:50] + "...")
print("Sequence length %i," % len(seq_record.seq))Hier gibt Server ["Orchidee"] das Handle zurück, um Daten aus der virtuellen Datenbankorchid abzurufen. lookup Methode bietet eine Option zur Auswahl von Sequenzen basierend auf Kriterien und wir haben die Sequenz mit der Kennung 2765658 ausgewählt. lookupGibt die Sequenzinformationen als SeqRecordobject zurück. Da wir bereits wissen, wie man mit SeqRecord arbeitet, ist es einfach, Daten daraus zu erhalten.
Entfernen Sie eine Datenbank
Das Entfernen einer Datenbank ist so einfach wie das Aufrufen der Methode remove_database mit dem richtigen Datenbanknamen und das anschließende Festschreiben wie unten angegeben.
from BioSQL import BioSeqDatabase
server = BioSeqDatabase.open_database(driver = 'sqlite3', db = "orchid.db")
server.remove_database("orchids")
server.commit()