Biopython - Modulo BioSQL
BioSQLè uno schema di database generico progettato principalmente per memorizzare sequenze e dati correlati per tutti i motori RDBMS. È progettato in modo tale da contenere i dati di tutti i database bioinformatici più diffusi come GenBank, Swissport, ecc. Può essere utilizzato anche per archiviare dati interni.
BioSQL attualmente fornisce uno schema specifico per i database seguenti:
- MySQL (biosqldb-mysql.sql)
- PostgreSQL (biosqldb-pg.sql)
- Oracle (biosqldb-ora / *. Sql)
- SQLite (biosqldb-sqlite.sql)
Fornisce inoltre un supporto minimo per i database HSQLDB e Derby basati su Java.
BioPython fornisce funzionalità ORM molto semplici, facili e avanzate per lavorare con database basati su BioSQL. BioPython provides a module, BioSQL per eseguire le seguenti funzionalità:
- Crea / rimuovi un database BioSQL
- Connettiti a un database BioSQL
- Analizza un database di sequenze come GenBank, Swisport, BLAST result, Entrez result, ecc. E caricalo direttamente nel database BioSQL
- Recupera i dati della sequenza dal database BioSQL
- Recupera i dati della tassonomia da NCBI BLAST e memorizzali nel database BioSQL
- Esegui qualsiasi query SQL sul database BioSQL
Panoramica dello schema del database BioSQL
Prima di approfondire il BioSQL, comprendiamo le basi dello schema BioSQL. Lo schema BioSQL fornisce oltre 25 tabelle per contenere dati di sequenza, funzionalità di sequenza, categoria / ontologia di sequenza e informazioni sulla tassonomia. Alcune delle tabelle importanti sono le seguenti:
- biodatabase
- bioentry
- biosequence
- seqfeature
- taxon
- taxon_name
- antology
- term
- dxref
Creazione di un database BioSQL
In questa sezione, creiamo un database BioSQL di esempio, biosql utilizzando lo schema fornito dal team BioSQL. Lavoreremo con il database SQLite poiché è molto facile iniziare e non ha una configurazione complessa.
Qui creeremo un database BioSQL basato su SQLite utilizzando i passaggi seguenti.
Step 1 - Scarica il motore di database SQLite e installalo.
Step 2 - Scarica il progetto BioSQL dall'URL di GitHub. https://github.com/biosql/biosql
Step 3 - Apri una console e crea una directory usando mkdir ed entra in essa.
cd /path/to/your/biopython/sample
mkdir sqlite-biosql
cd sqlite-biosqlStep 4 - Esegui il comando seguente per creare un nuovo database SQLite.
> sqlite3.exe mybiosql.db
SQLite version 3.25.2 2018-09-25 19:08:10
Enter ".help" for usage hints.
sqlite>Step 5 - Copia il file biosqldb-sqlite.sql dal progetto BioSQL (/ sql / biosqldb-sqlite.sql`) e memorizzalo nella directory corrente.
Step 6 - Esegui il comando seguente per creare tutte le tabelle.
sqlite> .read biosqldb-sqlite.sqlOra, tutte le tabelle vengono create nel nostro nuovo database.
Step 7 - Esegui il comando seguente per vedere tutte le nuove tabelle nel nostro database.
sqlite> .headers on
sqlite> .mode column
sqlite> .separator ROW "\n"
sqlite> SELECT name FROM sqlite_master WHERE type = 'table';
biodatabase
taxon
taxon_name
ontology
term
term_synonym
term_dbxref
term_relationship
term_relationship_term
term_path
bioentry
bioentry_relationship
bioentry_path
biosequence
dbxref
dbxref_qualifier_value
bioentry_dbxref
reference
bioentry_reference
comment
bioentry_qualifier_value
seqfeature
seqfeature_relationship
seqfeature_path
seqfeature_qualifier_value
seqfeature_dbxref
location
location_qualifier_value
sqlite>I primi tre comandi sono comandi di configurazione per configurare SQLite per mostrare il risultato in modo formattato.
Step 8 - Copia il file GenBank di esempio, ls_orchid.gbk fornito dal team di BioPython https://raw.githubusercontent.com/biopython/biopython/master/Doc/examples/ls_orchid.gbk nella directory corrente e salvarlo come orchid.gbk.
Step 9 - Crea uno script python, load_orchid.py usando il codice seguente ed eseguilo.
from Bio import SeqIO
from BioSQL import BioSeqDatabase
import os
server = BioSeqDatabase.open_database(driver = 'sqlite3', db = "orchid.db")
db = server.new_database("orchid")
count = db.load(SeqIO.parse("orchid.gbk", "gb"), True) server.commit()
server.close()Il codice sopra analizza il record nel file e lo converte in oggetti Python e lo inserisce nel database BioSQL. Analizzeremo il codice nella sezione successiva.
Infine, abbiamo creato un nuovo database BioSQL e vi abbiamo caricato alcuni dati di esempio. Discuteremo le tabelle importanti nel prossimo capitolo.
Schema ER semplice
biodatabase table è in cima alla gerarchia e il suo scopo principale è organizzare una serie di dati di sequenza in un singolo gruppo / database virtuale. Every entry in the biodatabase refers to a separate database and it does not mingle with another database. Tutte le tabelle correlate nel database BioSQL hanno riferimenti alla voce del biodatabase.
bioentrytabella contiene tutti i dettagli su una sequenza tranne i dati della sequenza. dati di sequenza di un particolarebioentry verrà memorizzato in biosequence tavolo.
taxon e taxon_name sono dettagli della tassonomia e ogni voce fa riferimento a questa tabella per specificare le informazioni sul taxon.
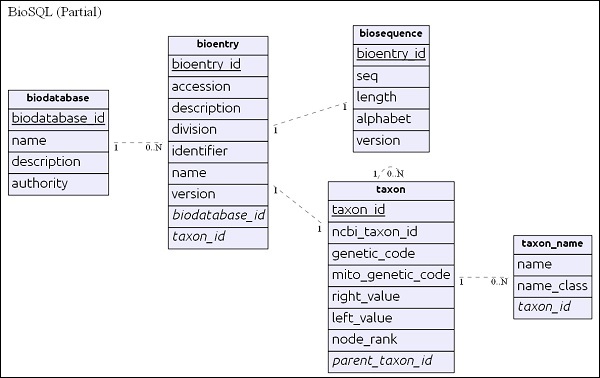
Dopo aver compreso lo schema, esaminiamo alcune query nella sezione successiva.
Query BioSQL
Approfondiamo alcune query SQL per capire meglio come sono organizzati i dati e le tabelle sono correlate tra loro. Prima di procedere, apriamo il database utilizzando il comando seguente e impostiamo alcuni comandi di formattazione:
> sqlite3 orchid.db
SQLite version 3.25.2 2018-09-25 19:08:10
Enter ".help" for usage hints.
sqlite> .header on
sqlite> .mode columns.header and .mode are formatting options to better visualize the data. È inoltre possibile utilizzare qualsiasi editor SQLite per eseguire la query.
Elenca il database delle sequenze virtuali disponibile nel sistema come indicato di seguito:
select
*
from
biodatabase;
*** Result ***
sqlite> .width 15 15 15 15
sqlite> select * from biodatabase;
biodatabase_id name authority description
--------------- --------------- --------------- ---------------
1 orchid
sqlite>Qui abbiamo un solo database, orchid.
Elenca le voci (prime 3) disponibili nel database orchid con il codice indicato di seguito
select
be.*,
bd.name
from
bioentry be
inner join
biodatabase bd
on bd.biodatabase_id = be.biodatabase_id
where
bd.name = 'orchid' Limit 1,
3;
*** Result ***
sqlite> .width 15 15 10 10 10 10 10 50 10 10
sqlite> select be.*, bd.name from bioentry be inner join biodatabase bd on
bd.biodatabase_id = be.biodatabase_id where bd.name = 'orchid' Limit 1,3;
bioentry_id biodatabase_id taxon_id name accession identifier division description version name
--------------- --------------- ---------- ---------- ---------- ---------- ----------
---------- ---------- ----------- ---------- --------- ---------- ----------
2 1 19 Z78532 Z78532 2765657 PLN
C.californicum 5.8S rRNA gene and ITS1 and ITS2 DN 1
orchid
3 1 20 Z78531 Z78531 2765656 PLN
C.fasciculatum 5.8S rRNA gene and ITS1 and ITS2 DN 1
orchid
4 1 21 Z78530 Z78530 2765655 PLN
C.margaritaceum 5.8S rRNA gene and ITS1 and ITS2 D 1
orchid
sqlite>Elenca i dettagli della sequenza associati a una voce (accession - Z78530, name - C. fasciculatum 5.8S rRNA gene and ITS1 and ITS2 DNA) with the given code -
select
substr(cast(bs.seq as varchar), 0, 10) || '...' as seq,
bs.length,
be.accession,
be.description,
bd.name
from
biosequence bs
inner join
bioentry be
on be.bioentry_id = bs.bioentry_id
inner join
biodatabase bd
on bd.biodatabase_id = be.biodatabase_id
where
bd.name = 'orchid'
and be.accession = 'Z78532';
*** Result ***
sqlite> .width 15 5 10 50 10
sqlite> select substr(cast(bs.seq as varchar), 0, 10) || '...' as seq,
bs.length, be.accession, be.description, bd.name from biosequence bs inner
join bioentry be on be.bioentry_id = bs.bioentry_id inner join biodatabase bd
on bd.biodatabase_id = be.biodatabase_id where bd.name = 'orchid' and
be.accession = 'Z78532';
seq length accession description name
------------ ---------- ---------- ------------ ------------ ---------- ---------- -----------------
CGTAACAAG... 753 Z78532 C.californicum 5.8S rRNA gene and ITS1 and ITS2 DNA orchid
sqlite>Ottieni la sequenza completa associata a una voce (accessione - Z78530, nome - gene rRNA C. fasciculatum 5.8S e DNA ITS1 e ITS2) utilizzando il codice seguente -
select
bs.seq
from
biosequence bs
inner join
bioentry be
on be.bioentry_id = bs.bioentry_id
inner join
biodatabase bd
on bd.biodatabase_id = be.biodatabase_id
where
bd.name = 'orchid'
and be.accession = 'Z78532';
*** Result ***
sqlite> .width 1000
sqlite> select bs.seq from biosequence bs inner join bioentry be on
be.bioentry_id = bs.bioentry_id inner join biodatabase bd on bd.biodatabase_id =
be.biodatabase_id where bd.name = 'orchid' and be.accession = 'Z78532';
seq
----------------------------------------------------------------------------------------
----------------------------
CGTAACAAGGTTTCCGTAGGTGAACCTGCGGAAGGATCATTGTTGAGACAACAGAATATATGATCGAGTGAATCT
GGAGGACCTGTGGTAACTCAGCTCGTCGTGGCACTGCTTTTGTCGTGACCCTGCTTTGTTGTTGGGCCTCC
TCAAGAGCTTTCATGGCAGGTTTGAACTTTAGTACGGTGCAGTTTGCGCCAAGTCATATAAAGCATCACTGATGAATGACATTATTGT
CAGAAAAAATCAGAGGGGCAGTATGCTACTGAGCATGCCAGTGAATTTTTATGACTCTCGCAACGGATATCTTGGCTC
TAACATCGATGAAGAACGCAG
sqlite>Elenca il taxon associato al database bio, orchidea
select distinct
tn.name
from
biodatabase d
inner join
bioentry e
on e.biodatabase_id = d.biodatabase_id
inner join
taxon t
on t.taxon_id = e.taxon_id
inner join
taxon_name tn
on tn.taxon_id = t.taxon_id
where
d.name = 'orchid' limit 10;
*** Result ***
sqlite> select distinct tn.name from biodatabase d inner join bioentry e on
e.biodatabase_id = d.biodatabase_id inner join taxon t on t.taxon_id =
e.taxon_id inner join taxon_name tn on tn.taxon_id = t.taxon_id where d.name =
'orchid' limit 10;
name
------------------------------
Cypripedium irapeanum
Cypripedium californicum
Cypripedium fasciculatum
Cypripedium margaritaceum
Cypripedium lichiangense
Cypripedium yatabeanum
Cypripedium guttatum
Cypripedium acaule
pink lady's slipper
Cypripedium formosanum
sqlite>Carica i dati nel database BioSQL
Impariamo come caricare i dati di sequenza nel database BioSQL in questo capitolo. Abbiamo già il codice per caricare i dati nel database nella sezione precedente e il codice è il seguente:
from Bio import SeqIO
from BioSQL import BioSeqDatabase
import os
server = BioSeqDatabase.open_database(driver = 'sqlite3', db = "orchid.db")
DBSCHEMA = "biosqldb-sqlite.sql"
SQL_FILE = os.path.join(os.getcwd(), DBSCHEMA)
server.load_database_sql(SQL_FILE)
server.commit()
db = server.new_database("orchid")
count = db.load(SeqIO.parse("orchid.gbk", "gb"), True) server.commit()
server.close()Daremo uno sguardo più approfondito a ogni riga del codice e al suo scopo:
Line 1 - Carica il modulo SeqIO.
Line 2- Carica il modulo BioSeqDatabase. Questo modulo fornisce tutte le funzionalità per interagire con il database BioSQL.
Line 3 - Carica il modulo del sistema operativo.
Line 5- open_database apre il database specificato (db) con il driver configurato (driver) e restituisce un handle al database BioSQL (server). Biopython supporta i database sqlite, mysql, postgresql e Oracle.
Line 6-10- Il metodo load_database_sql carica sql dal file esterno e lo esegue. Il metodo commit esegue il commit della transazione. Possiamo saltare questo passaggio perché abbiamo già creato il database con lo schema.
Line 12 - i metodi new_database creano un nuovo database virtuale, orchid e restituiscono un handle db per eseguire il comando sul database orchid.
Line 13- Il metodo load carica le voci della sequenza (SeqRecord iterabile) nel database dell'orchidea. SqlIO.parse analizza il database GenBank e restituisce tutte le sequenze in esso contenute come SeqRecord iterabile. Il secondo parametro (True) del metodo di caricamento indica di recuperare i dettagli della tassonomia dei dati della sequenza dal sito Web NCBI Blast, se non è già disponibile nel sistema.
Line 14 - commit esegue il commit della transazione.
Line 15 - close chiude la connessione al database e distrugge l'handle del server.
Recupera i dati della sequenza
Andiamo a prendere una sequenza con identificatore, 2765658 dal database orchidea come di seguito -
from BioSQL import BioSeqDatabase
server = BioSeqDatabase.open_database(driver = 'sqlite3', db = "orchid.db")
db = server["orchid"]
seq_record = db.lookup(gi = 2765658)
print(seq_record.id, seq_record.description[:50] + "...")
print("Sequence length %i," % len(seq_record.seq))Qui, il server ["orchid"] restituisce l'handle per recuperare i dati dal database virtualeorchid. lookup fornisce un'opzione per selezionare le sequenze in base a criteri e abbiamo selezionato la sequenza con identificatore, 2765658. lookuprestituisce le informazioni sulla sequenza come SeqRecordobject. Poiché sappiamo già come lavorare con SeqRecord`, è facile ottenere dati da esso.
Rimuovi un database
La rimozione di un database è semplice come chiamare il metodo remove_database con il nome del database corretto e quindi eseguirne il commit come specificato di seguito -
from BioSQL import BioSeqDatabase
server = BioSeqDatabase.open_database(driver = 'sqlite3', db = "orchid.db")
server.remove_database("orchids")
server.commit()