Biopython - Plottaggio
Questo capitolo spiega come tracciare sequenze. Prima di passare a questo argomento, comprendiamo le basi della stampa.
Tracciare
Matplotlib è una libreria di plottaggio Python che produce figure di qualità in una varietà di formati. Possiamo creare diversi tipi di grafici come grafico a linee, istogrammi, grafico a barre, grafico a torta, grafico a dispersione, ecc.
pyLab is a module that belongs to the matplotlib which combines the numerical module numpy with the graphical plotting module pyplot.Biopython utilizza il modulo pylab per tracciare sequenze. Per fare ciò, dobbiamo importare il codice seguente:
import pylabPrima di importare, dobbiamo installare il pacchetto matplotlib utilizzando il comando pip con il comando indicato di seguito -
pip install matplotlibFile di input di esempio
Crea un file di esempio denominato plot.fasta nella tua directory Biopython e aggiungi le seguenti modifiche:
>seq0 FQTWEEFSRAAEKLYLADPMKVRVVLKYRHVDGNLCIKVTDDLVCLVYRTDQAQDVKKIEKF
>seq1 KYRTWEEFTRAAEKLYQADPMKVRVVLKYRHCDGNLCIKVTDDVVCLLYRTDQAQDVKKIEKFHSQLMRLME
>seq2 EEYQTWEEFARAAEKLYLTDPMKVRVVLKYRHCDGNLCMKVTDDAVCLQYKTDQAQDVKKVEKLHGK
>seq3 MYQVWEEFSRAVEKLYLTDPMKVRVVLKYRHCDGNLCIKVTDNSVCLQYKTDQAQDV
>seq4 EEFSRAVEKLYLTDPMKVRVVLKYRHCDGNLCIKVTDNSVVSYEMRLFGVQKDNFALEHSLL
>seq5 SWEEFAKAAEVLYLEDPMKCRMCTKYRHVDHKLVVKLTDNHTVLKYVTDMAQDVKKIEKLTTLLMR
>seq6 FTNWEEFAKAAERLHSANPEKCRFVTKYNHTKGELVLKLTDDVVCLQYSTNQLQDVKKLEKLSSTLLRSI
>seq7 SWEEFVERSVQLFRGDPNATRYVMKYRHCEGKLVLKVTDDRECLKFKTDQAQDAKKMEKLNNIFF
>seq8 SWDEFVDRSVQLFRADPESTRYVMKYRHCDGKLVLKVTDNKECLKFKTDQAQEAKKMEKLNNIFFTLM
>seq9 KNWEDFEIAAENMYMANPQNCRYTMKYVHSKGHILLKMSDNVKCVQYRAENMPDLKK
>seq10 FDSWDEFVSKSVELFRNHPDTTRYVVKYRHCEGKLVLKVTDNHECLKFKTDQAQDAKKMEKGrafico a linee
Ora, creiamo un semplice grafico a linee per il file fasta sopra.
Step 1 - Importa il modulo SeqIO per leggere il file fasta.
>>> from Bio import SeqIOStep 2 - Analizza il file di input.
>>> records = [len(rec) for rec in SeqIO.parse("plot.fasta", "fasta")]
>>> len(records)
11
>>> max(records)
72
>>> min(records)
57Step 3 - Importiamo il modulo pylab.
>>> import pylabStep 4 - Configurare il grafico a linee assegnando le etichette degli assi x e y.
>>> pylab.xlabel("sequence length")
Text(0.5, 0, 'sequence length')
>>> pylab.ylabel("count")
Text(0, 0.5, 'count')
>>>Step 5 - Configurare il grafico a linee impostando la visualizzazione della griglia.
>>> pylab.grid()Step 6 - Disegna un semplice grafico a linee chiamando il metodo di trama e fornendo i record come input.
>>> pylab.plot(records)
[<matplotlib.lines.Line2D object at 0x10b6869d 0>]Step 7 - Infine salva il grafico utilizzando il comando seguente.
>>> pylab.savefig("lines.png")Risultato
Dopo aver eseguito il comando precedente, potresti vedere la seguente immagine salvata nella tua directory Biopython.
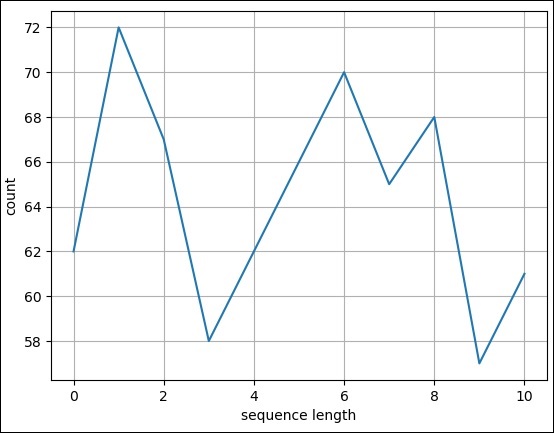
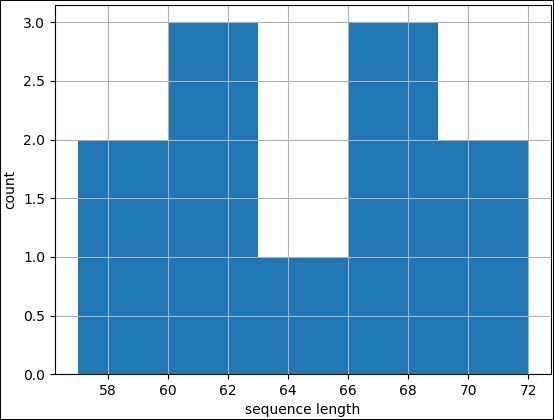
Grafico istogramma
Un istogramma viene utilizzato per i dati continui, in cui i contenitori rappresentano intervalli di dati. L'istogramma del disegno è lo stesso del grafico a linee tranne pylab.plot. Invece, chiama il metodo hist del modulo pylab con i record e un po 'di valore custum per bins (5). La codifica completa è la seguente:
Step 1 - Importa il modulo SeqIO per leggere il file fasta.
>>> from Bio import SeqIOStep 2 - Analizza il file di input.
>>> records = [len(rec) for rec in SeqIO.parse("plot.fasta", "fasta")]
>>> len(records)
11
>>> max(records)
72
>>> min(records)
57Step 3 - Importiamo il modulo pylab.
>>> import pylabStep 4 - Configurare il grafico a linee assegnando le etichette degli assi x e y.
>>> pylab.xlabel("sequence length")
Text(0.5, 0, 'sequence length')
>>> pylab.ylabel("count")
Text(0, 0.5, 'count')
>>>Step 5 - Configurare il grafico a linee impostando la visualizzazione della griglia.
>>> pylab.grid()Step 6 - Disegna un semplice grafico a linee chiamando il metodo di trama e fornendo i record come input.
>>> pylab.hist(records,bins=5)
(array([2., 3., 1., 3., 2.]), array([57., 60., 63., 66., 69., 72.]), <a list
of 5 Patch objects>)
>>>Step 7 - Infine salva il grafico utilizzando il comando seguente.
>>> pylab.savefig("hist.png")Risultato
Dopo aver eseguito il comando precedente, potresti vedere la seguente immagine salvata nella tua directory Biopython.
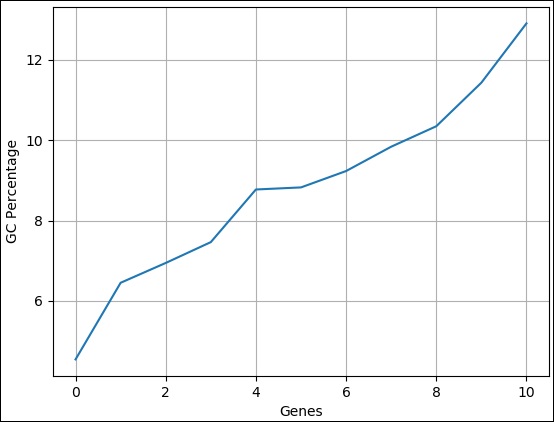
Percentuale GC in sequenza
La percentuale GC è uno dei dati analitici comunemente utilizzati per confrontare sequenze diverse. Possiamo fare un semplice grafico a linee usando GC Percentage di un insieme di sequenze e confrontarlo immediatamente. Qui, possiamo semplicemente cambiare i dati dalla lunghezza della sequenza alla percentuale GC. Di seguito viene fornita la codifica completa:
Step 1 - Importa il modulo SeqIO per leggere il file fasta.
>>> from Bio import SeqIOStep 2 - Analizza il file di input.
>>> from Bio.SeqUtils import GC
>>> gc = sorted(GC(rec.seq) for rec in SeqIO.parse("plot.fasta", "fasta"))Step 3 - Importiamo il modulo pylab.
>>> import pylabStep 4 - Configurare il grafico a linee assegnando le etichette degli assi x e y.
>>> pylab.xlabel("Genes")
Text(0.5, 0, 'Genes')
>>> pylab.ylabel("GC Percentage")
Text(0, 0.5, 'GC Percentage')
>>>Step 5 - Configurare il grafico a linee impostando la visualizzazione della griglia.
>>> pylab.grid()Step 6 - Disegna un semplice grafico a linee chiamando il metodo di trama e fornendo i record come input.
>>> pylab.plot(gc)
[<matplotlib.lines.Line2D object at 0x10b6869d 0>]Step 7 - Infine salva il grafico utilizzando il comando seguente.
>>> pylab.savefig("gc.png")Risultato
Dopo aver eseguito il comando precedente, potresti vedere la seguente immagine salvata nella tua directory Biopython.