Microsoft Cognitive Toolkit - Szybki przewodnik
W tym rozdziale dowiemy się, czym jest CNTK, jakie są jego funkcje, jaka jest różnica między wersją 1.0 i 2.0 oraz najważniejsze informacje dotyczące wersji 2.7.
Co to jest Microsoft Cognitive Toolkit (CNTK)?
Microsoft Cognitive Toolkit (CNTK), wcześniej znany jako Computational Network Toolkit, to darmowy, łatwy w użyciu, komercyjny zestaw narzędzi o otwartym kodzie źródłowym, który pozwala nam trenować algorytmy uczenia głębokiego, aby uczyć się jak ludzki mózg. Umożliwia nam tworzenie popularnych systemów uczenia głębokiego, takich jakfeed-forward neural network time series prediction systems and Convolutional neural network (CNN) image classifiers.
Aby uzyskać optymalną wydajność, jego funkcje ramowe są napisane w C ++. Chociaż możemy wywołać jego funkcję za pomocą C ++, ale najczęściej używanym podejściem do tego samego jest użycie programu w języku Python.
Funkcje CNTK
Poniżej przedstawiono niektóre funkcje i możliwości oferowane w najnowszej wersji Microsoft CNTK:
Wbudowane komponenty
CNTK ma wysoce zoptymalizowane wbudowane komponenty, które mogą obsługiwać wielowymiarowe, gęste lub rzadkie dane z Pythona, C ++ lub BrainScript.
Z uwagą możemy wdrożyć CNN, FNN, RNN, Batch Normalization i Sequence-to-Sequence.
Zapewnia nam funkcjonalność dodawania nowych zdefiniowanych przez użytkownika podstawowych komponentów do GPU z Pythona.
Zapewnia również automatyczne dostrajanie hiperparametrów.
Możemy wdrożyć uczenie się ze wzmocnieniem, generatywne sieci adwersyjne (GAN), uczenie nadzorowane i nienadzorowane.
W przypadku ogromnych zbiorów danych CNTK ma wbudowane zoptymalizowane czytniki.
Efektywne wykorzystanie zasobów
CNTK zapewnia nam równoległość z wysoką dokładnością na wielu GPU / maszynach za pośrednictwem 1-bitowego SGD.
Aby zmieścić największe modele w pamięci GPU, zapewnia współdzielenie pamięci i inne wbudowane metody.
Łatwo wyrażaj własne sieci
CNTK ma pełne interfejsy API do definiowania własnej sieci, osób uczących się, czytelników, szkoleń i oceny w językach Python, C ++ i BrainScript.
Używając CNTK, możemy łatwo oceniać modele za pomocą Pythona, C ++, C # lub BrainScript.
Zapewnia zarówno interfejsy API wysokiego, jak i niskiego poziomu.
Na podstawie naszych danych może automatycznie kształtować wnioskowanie.
Posiada w pełni zoptymalizowane symboliczne pętle rekurencyjnych sieci neuronowych (RNN).
Mierzenie wydajności modelu
CNTK zapewnia różne komponenty do pomiaru wydajności budowanych sieci neuronowych.
Generuje dane dziennika z Twojego modelu i powiązanego optymalizatora, których możemy użyć do monitorowania procesu uczenia.
Wersja 1.0 vs wersja 2.0
Poniższa tabela porównuje wersje CNTK 1.0 i 2.0:
| Wersja 1.0.0 | Wersja 2.0.0 |
|---|---|
| Został wydany w 2016 roku. | Jest to znacząca przeróbka wersji 1.0 i została wydana w czerwcu 2017 roku. |
| Używał zastrzeżonego języka skryptowego o nazwie BrainScript. | Jego funkcje ramowe można wywołać za pomocą C ++, Python. Nasze moduły możemy łatwo załadować w języku C # lub Javie. BrainScript jest również obsługiwany przez wersję 2.0. |
| Działa w systemach Windows i Linux, ale nie bezpośrednio w systemie Mac OS. | Działa również w systemach Windows (Win 8.1, Win 10, Server 2012 R2 i nowsze) i Linux, ale nie bezpośrednio w systemie Mac OS. |
Ważne informacje o wersji 2.7
Version 2.7jest ostatnią główną wydaną wersją Microsoft Cognitive Toolkit. Posiada pełne wsparcie dla ONNX 1.4.1. Poniżej znajduje się kilka ważnych informacji dotyczących ostatniej wydanej wersji CNTK.
Pełne wsparcie dla ONNX 1.4.1.
Obsługa CUDA 10 dla systemów Windows i Linux.
Obsługuje zaawansowaną pętlę rekurencyjnych sieci neuronowych (RNN) w eksporcie ONNX.
Może eksportować więcej niż modele 2 GB w formacie ONNX.
Obsługuje FP16 w działaniu szkoleniowym języka skryptowego BrainScript.
Tutaj zrozumiemy o instalacji CNTK w systemie Windows i Linux. Ponadto rozdział wyjaśnia instalację pakietu CNTK, kroki instalacji Anacondy, pliki CNTK, strukturę katalogów i organizację biblioteki CNTK.
Wymagania wstępne
Aby zainstalować CNTK, musimy mieć zainstalowany Python na naszych komputerach. Możesz przejść do linkuhttps://www.python.org/downloads/i wybierz najnowszą wersję dla swojego systemu operacyjnego, tj. Windows i Linux / Unix. Aby zapoznać się z podstawowym samouczkiem dotyczącym języka Python, możesz skorzystać z łączahttps://www.tutorialspoint.com/python3/index.htm.

CNTK jest obsługiwany zarówno w systemie Windows, jak i Linux, więc omówimy oba z nich.
Instalowanie w systemie Windows
Aby uruchomić CNTK w systemie Windows, będziemy używać rozszerzenia Anaconda versionPythona. Wiemy, że Anaconda jest redystrybucją Pythona. Zawiera dodatkowe pakiety, takie jakScipy iScikit-learn które są używane przez CNTK do wykonywania różnych użytecznych obliczeń.
Więc najpierw zobaczmy kroki, aby zainstalować Anaconda na twoim komputerze -
Step 1−Najpierw pobierz pliki instalacyjne z publicznej witryny internetowej https://www.anaconda.com/distribution/.
Step 2 - Po pobraniu plików instalacyjnych rozpocznij instalację i postępuj zgodnie z instrukcjami z łącza https://docs.anaconda.com/anaconda/install/.
Step 3- Po zainstalowaniu Anaconda zainstaluje również inne narzędzia, które automatycznie dołączą wszystkie pliki wykonywalne Anaconda do zmiennej PATH komputera. Możemy zarządzać naszym środowiskiem Python z tego znaku zachęty, możemy instalować pakiety i uruchamiać skrypty Pythona.
Instalowanie pakietu CNTK
Po zakończeniu instalacji Anacondy możesz użyć najczęstszego sposobu instalacji pakietu CNTK za pomocą pliku wykonywalnego pip, używając następującego polecenia -
pip install cntkIstnieje wiele innych metod instalacji Cognitive Toolkit na komputerze. Firma Microsoft ma zgrabny zestaw dokumentacji, która szczegółowo wyjaśnia inne metody instalacji. Proszę skorzystać z linkuhttps://docs.microsoft.com/en-us/cognitive-toolkit/Setup-CNTK-on-your-machine.
Instalowanie w systemie Linux
Instalacja CNTK w systemie Linux różni się nieco od instalacji w systemie Windows. Tutaj, dla Linuksa zamierzamy użyć Anacondy do zainstalowania CNTK, ale zamiast graficznego instalatora dla Anacondy będziemy używać instalatora opartego na terminalu w Linuksie. Chociaż instalator będzie działał z prawie wszystkimi dystrybucjami Linuksa, ograniczyliśmy opis do Ubuntu.
Więc najpierw zobaczmy kroki, aby zainstalować Anaconda na twoim komputerze -
Kroki, aby zainstalować Anaconda
Step 1- Przed zainstalowaniem Anacondy upewnij się, że system jest w pełni aktualny. Aby to sprawdzić, najpierw wykonaj następujące dwa polecenia w terminalu -
sudo apt update
sudo apt upgradeStep 2 - Po zaktualizowaniu komputera pobierz adres URL z publicznej witryny internetowej https://www.anaconda.com/distribution/ najnowsze pliki instalacyjne Anaconda.
Step 3 - Po skopiowaniu adresu URL otwórz okno terminala i wykonaj następujące polecenie -
wget -0 anaconda-installer.sh url SHAPE \* MERGEFORMAT
y
f
x
| }Zastąp url symbol zastępczy z adresem URL skopiowanym z witryny internetowej Anaconda.
Step 4 - Następnie za pomocą następującego polecenia możemy zainstalować Anacondę -
sh ./anaconda-installer.shPowyższe polecenie zostanie domyślnie zainstalowane Anaconda3 wewnątrz naszego katalogu domowego.
Instalowanie pakietu CNTK
Po zakończeniu instalacji Anacondy możesz użyć najczęstszego sposobu instalacji pakietu CNTK za pomocą pliku wykonywalnego pip, używając następującego polecenia -
pip install cntkBadanie plików i struktury katalogów CNTK
Po zainstalowaniu CNTK jako pakietu Pythona możemy sprawdzić jego strukturę plików i katalogów. Jest oC:\Users\

Weryfikacja instalacji CNTK
Po zainstalowaniu CNTK jako pakietu Pythona należy sprawdzić, czy CNTK został zainstalowany poprawnie. W powłoce poleceń Anaconda uruchom interpreter Pythona, wpisującipython. Następnie zaimportuj CNTK wpisując następujące polecenie.
import cntk as cPo zaimportowaniu sprawdź jego wersję za pomocą następującego polecenia -
print(c.__version__)Tłumacz odpowie z zainstalowaną wersją CNTK. Jeśli nie odpowiada, wystąpi problem z instalacją.
Organizacja biblioteczna CNTK
CNTK, pakiet Pythona z technicznego punktu widzenia, jest podzielony na 13 pod-pakietów wysokiego poziomu i 8 mniejszych pod-pakietów. Poniższa tabela zawiera 10 najczęściej używanych pakietów:
| Sr.No | Nazwa i opis pakietu |
|---|---|
| 1 | cntk.io Zawiera funkcje do odczytu danych. Na przykład: next_minibatch () |
| 2 | cntk.layers Zawiera funkcje wysokiego poziomu do tworzenia sieci neuronowych. Na przykład: Gęsty () |
| 3 | cntk.learners Zawiera funkcje do treningu. Na przykład: sgd () |
| 4 | cntk.losses Zawiera funkcje do pomiaru błędu treningu. Na przykład: squared_error () |
| 5 | cntk.metrics Zawiera funkcje do pomiaru błędu modelu. Na przykład: classificatoin_error |
| 6 | cntk.ops Zawiera niskopoziomowe funkcje do tworzenia sieci neuronowych. Na przykład: tanh () |
| 7 | cntk.random Zawiera funkcje do generowania liczb losowych. Na przykład: normal () |
| 8 | cntk.train Zawiera funkcje treningowe. Na przykład: train_minibatch () |
| 9 | cntk.initializer Zawiera inicjatory parametrów modelu. Na przykład: normal () i uniform () |
| 10 | cntk.variables Zawiera konstrukcje niskiego poziomu. Na przykład: Parameter () and Variable () |
Microsoft Cognitive Toolkit oferuje dwie różne wersje kompilacji, a mianowicie tylko CPU i tylko GPU.
Wersja kompilacji tylko dla procesora
Wersja kompilacji CNTK tylko z procesorem korzysta ze zoptymalizowanej wersji Intel MKLML, gdzie MKLML jest podzbiorem MKL (Math Kernel Library) i wydana z Intel MKL-DNN jako zakończona wersja Intel MKL dla MKL-DNN.
Wersja kompilacji tylko dla GPU
Z drugiej strony wersja CNTK przeznaczona tylko do GPU wykorzystuje wysoce zoptymalizowane biblioteki NVIDIA, takie jak CUB i cuDNN. Obsługuje rozproszone szkolenie na wielu procesorach graficznych i wielu maszynach. Aby jeszcze bardziej przyspieszyć rozproszone szkolenie w CNTK, wersja kompilacji GPU zawiera również -
Opracowany przez MSR 1-bitowy kwantowany SGD.
Algorytmy równoległego uczenia Block-Momum SGD.
Włączanie GPU z CNTK w systemie Windows
W poprzedniej sekcji widzieliśmy, jak zainstalować podstawową wersję CNTK do użytku z procesorem. Omówmy teraz, jak możemy zainstalować CNTK do użytku z GPU. Ale zanim się w to zagłębisz, najpierw powinieneś mieć obsługiwaną kartę graficzną.
Obecnie CNTK obsługuje kartę graficzną NVIDIA z obsługą co najmniej CUDA 3.0. Aby się upewnić, możesz sprawdzić pod adresemhttps://developer.nvidia.com/cuda-gpus czy twój GPU obsługuje CUDA.
Zobaczmy więc, jak włączyć GPU z CNTK w systemie operacyjnym Windows -
Step 1 - W zależności od używanej karty graficznej, najpierw musisz mieć najnowsze sterowniki GeForce lub Quadro dla swojej karty graficznej.
Step 2 - Po pobraniu sterowników należy zainstalować zestaw narzędzi CUDA w wersji 9.0 dla Windows ze strony internetowej NVIDIA https://developer.nvidia.com/cuda-90-download-archive?target_os=Windows&target_arch=x86_64. Po zainstalowaniu uruchom instalator i postępuj zgodnie z instrukcjami.
Step 3 - Następnie musisz zainstalować pliki binarne cuDNN ze strony internetowej NVIDIA https://developer.nvidia.com/rdp/form/cudnn-download-survey. W wersji CUDA 9.0 cuDNN 7.4.1 działa dobrze. Zasadniczo cuDNN to warstwa na wierzchu CUDA, używana przez CNTK.
Step 4 - Po pobraniu plików binarnych cuDNN należy rozpakować plik zip do folderu głównego instalacji zestawu narzędzi CUDA.
Step 5- To ostatni krok, który umożliwi użycie GPU w CNTK. Wykonaj następujące polecenie w wierszu polecenia Anaconda w systemie operacyjnym Windows -
pip install cntk-gpuWłączanie GPU z CNTK w systemie Linux
Zobaczmy, jak możemy włączyć GPU z CNTK w systemie operacyjnym Linux -
Pobieranie zestawu narzędzi CUDA
Najpierw musisz zainstalować zestaw narzędzi CUDA z witryny NVIDIA https://developer.nvidia.com/cuda-90-download-archive?target_os=Linux&target_arch=x86_64&target_distro=Ubuntu&target_version=1604&target_type = runfilelocal .
Uruchomienie instalatora
Teraz, gdy masz już pliki binarne na dysku, uruchom instalator, otwierając terminal i wykonując następujące polecenie oraz instrukcję na ekranie -
sh cuda_9.0.176_384.81_linux-runZmodyfikuj skrypt profilu Bash
Po zainstalowaniu zestawu narzędzi CUDA na komputerze z systemem Linux należy zmodyfikować skrypt profilu BASH. W tym celu najpierw otwórz plik $ HOME / .bashrc w edytorze tekstu. Teraz na końcu skryptu dołącz następujące wiersze -
export PATH=/usr/local/cuda-9.0/bin${PATH:+:${PATH}} export LD_LIBRARY_PATH=/usr/local/cuda-9.0/lib64\ ${LD_LIBRARY_PATH:+:${LD_LIBRARY_PATH}}
InstallingInstalowanie bibliotek cuDNN
W końcu musimy zainstalować pliki binarne cuDNN. Można go pobrać ze strony internetowej NVIDIAhttps://developer.nvidia.com/rdp/form/cudnn-download-survey. W wersji CUDA 9.0 cuDNN 7.4.1 działa dobrze. Zasadniczo cuDNN to warstwa na wierzchu CUDA, używana przez CNTK.
Po pobraniu wersji dla systemu Linux wyodrębnij ją do pliku /usr/local/cuda-9.0 folder za pomocą następującego polecenia -
tar xvzf -C /usr/local/cuda-9.0/ cudnn-9.0-linux-x64-v7.4.1.5.tgzZmień ścieżkę do nazwy pliku zgodnie z wymaganiami.
W tym rozdziale dowiemy się szczegółowo o sekwencjach w CNTK i ich klasyfikacji.
Tensory
Koncepcja, nad którą pracuje CNTK, to tensor. Zasadniczo wejścia i wyjścia CNTK, a także parametry są zorganizowane jakotensors, który jest często uważany za uogólnioną macierz. Każdy tensor marank -
Tensor rzędu 0 jest skalarem.
Tensor rzędu 1 jest wektorem.
Tensor rzędu 2 to amatrix.
Tutaj te różne wymiary są nazywane axes.
Osie statyczne i osie dynamiczne
Jak sama nazwa wskazuje, statyczne osie mają tę samą długość przez cały okres eksploatacji sieci. Z drugiej strony długość dynamicznych osi może się różnić w zależności od instancji. W rzeczywistości ich długość zazwyczaj nie jest znana przed przedstawieniem każdej minibatchu.
Osie dynamiczne są jak osie statyczne, ponieważ definiują również znaczące zgrupowanie liczb zawartych w tensorze.
Przykład
Aby było jaśniej, zobaczmy, jak mała partia krótkich klipów wideo jest reprezentowana w CNTK. Załóżmy, że wszystkie klipy wideo mają rozdzielczość 640 * 480. Ponadto klipy są nagrywane w kolorze, który jest zwykle kodowany trzema kanałami. Oznacza to ponadto, że nasz minibatch ma następujące -
3 osie statyczne o długości odpowiednio 640, 480 i 3.
Dwie dynamiczne osie; długość filmu i osie minibatchu.
Oznacza to, że jeśli minibatch zawiera 16 filmów, z których każdy ma 240 klatek, zostanie przedstawiony jako 16*240*3*640*480 tensory.
Praca z sekwencjami w CNTK
Zrozummy sekwencje w CNTK, najpierw poznając Sieć Pamięci Długoterminowej.
Sieć pamięci długoterminowej (LSTM)

Sieci pamięci długoterminowej (LSTM) zostały wprowadzone przez Hochreiter & Schmidhuber. Rozwiązał problem uzyskania podstawowej powtarzającej się warstwy, aby pamiętać rzeczy przez długi czas. Architekturę LSTM przedstawiono powyżej na schemacie. Jak widzimy, ma neurony wejściowe, komórki pamięci i neurony wyjściowe. Aby zwalczyć problem znikającego gradientu, sieci pamięci długoterminowej używają jawnej komórki pamięci (przechowuje poprzednie wartości) i następujących bramek -
Forget gate- Jak sama nazwa wskazuje, mówi komórce pamięci, aby zapomniała poprzednich wartości. Komórka pamięci przechowuje wartości do momentu, gdy bramka, tj. „Zapomnij bramę”, powie jej, aby je zapomnieć.
Input gate - Jak sama nazwa wskazuje, dodaje nowe rzeczy do komórki.
Output gate - Jak sama nazwa wskazuje, bramka wyjściowa decyduje, kiedy przejść wzdłuż wektorów z komórki do następnego stanu ukrytego.
Praca z sekwencjami w CNTK jest bardzo łatwa. Zobaczmy to na poniższym przykładzie -
import sys
import os
from cntk import Trainer, Axis
from cntk.io import MinibatchSource, CTFDeserializer, StreamDef, StreamDefs,\
INFINITELY_REPEAT
from cntk.learners import sgd, learning_parameter_schedule_per_sample
from cntk import input_variable, cross_entropy_with_softmax, \
classification_error, sequence
from cntk.logging import ProgressPrinter
from cntk.layers import Sequential, Embedding, Recurrence, LSTM, Dense
def create_reader(path, is_training, input_dim, label_dim):
return MinibatchSource(CTFDeserializer(path, StreamDefs(
features=StreamDef(field='x', shape=input_dim, is_sparse=True),
labels=StreamDef(field='y', shape=label_dim, is_sparse=False)
)), randomize=is_training,
max_sweeps=INFINITELY_REPEAT if is_training else 1)
def LSTM_sequence_classifier_net(input, num_output_classes, embedding_dim,
LSTM_dim, cell_dim):
lstm_classifier = Sequential([Embedding(embedding_dim),
Recurrence(LSTM(LSTM_dim, cell_dim)),
sequence.last,
Dense(num_output_classes)])
return lstm_classifier(input)
def train_sequence_classifier():
input_dim = 2000
cell_dim = 25
hidden_dim = 25
embedding_dim = 50
num_output_classes = 5
features = sequence.input_variable(shape=input_dim, is_sparse=True)
label = input_variable(num_output_classes)
classifier_output = LSTM_sequence_classifier_net(
features, num_output_classes, embedding_dim, hidden_dim, cell_dim)
ce = cross_entropy_with_softmax(classifier_output, label)
pe = classification_error(classifier_output, label)
rel_path = ("../../../Tests/EndToEndTests/Text/" +
"SequenceClassification/Data/Train.ctf")
path = os.path.join(os.path.dirname(os.path.abspath(__file__)), rel_path)
reader = create_reader(path, True, input_dim, num_output_classes)
input_map = {
features: reader.streams.features,
label: reader.streams.labels
}
lr_per_sample = learning_parameter_schedule_per_sample(0.0005)
progress_printer = ProgressPrinter(0)
trainer = Trainer(classifier_output, (ce, pe),
sgd(classifier_output.parameters, lr=lr_per_sample),progress_printer)
minibatch_size = 200
for i in range(255):
mb = reader.next_minibatch(minibatch_size, input_map=input_map)
trainer.train_minibatch(mb)
evaluation_average = float(trainer.previous_minibatch_evaluation_average)
loss_average = float(trainer.previous_minibatch_loss_average)
return evaluation_average, loss_average
if __name__ == '__main__':
error, _ = train_sequence_classifier()
print(" error: %f" % error)average since average since examples
loss last metric last
------------------------------------------------------
1.61 1.61 0.886 0.886 44
1.61 1.6 0.714 0.629 133
1.6 1.59 0.56 0.448 316
1.57 1.55 0.479 0.41 682
1.53 1.5 0.464 0.449 1379
1.46 1.4 0.453 0.441 2813
1.37 1.28 0.45 0.447 5679
1.3 1.23 0.448 0.447 11365
error: 0.333333Szczegółowe wyjaśnienie powyższego programu zostanie omówione w następnych sekcjach, zwłaszcza gdy będziemy budować rekurencyjne sieci neuronowe.
Ten rozdział dotyczy budowy modelu regresji logistycznej w CNTK.
Podstawy modelu regresji logistycznej
Regresja logistyczna, jedna z najprostszych technik ML, jest techniką przeznaczoną szczególnie do klasyfikacji binarnej. Innymi słowy, aby utworzyć model predykcyjny w sytuacjach, w których wartość zmiennej do prognozowania może być jedną z zaledwie dwóch wartości jakościowych. Jednym z najprostszych przykładów regresji logistycznej jest przewidywanie, czy dana osoba jest mężczyzną czy kobietą, na podstawie jej wieku, głosu, włosów i tak dalej.
Przykład
Zrozummy pojęcie regresji logistycznej matematycznie na innym przykładzie -
Załóżmy, że chcemy przewidzieć zdolność kredytową wniosku o pożyczkę; 0 oznacza odrzucenie, a 1 oznacza zatwierdzenie w oparciu o wnioskodawcędebt , income i credit rating. Reprezentujemy zadłużenie na X1, dochód na X2 i rating kredytowy na X3.
W regresji logistycznej określamy wartość wagi, reprezentowaną przez w, dla każdej funkcji i pojedynczej wartości odchylenia, reprezentowanej przez b.
Teraz przypuśćmy,
X1 = 3.0
X2 = -2.0
X3 = 1.0Załóżmy, że określimy wagę i odchylenie w następujący sposób -
W1 = 0.65, W2 = 1.75, W3 = 2.05 and b = 0.33Teraz, aby przewidzieć klasę, musimy zastosować następującą formułę -
Z = (X1*W1)+(X2*W2)+(X3+W3)+b
i.e. Z = (3.0)*(0.65) + (-2.0)*(1.75) + (1.0)*(2.05) + 0.33
= 0.83Następnie musimy obliczyć P = 1.0/(1.0 + exp(-Z)). Tutaj funkcja exp () jest liczbą Eulera.
P = 1.0/(1.0 + exp(-0.83)
= 0.6963Wartość P można interpretować jako prawdopodobieństwo, że klasa jest równa 1. Jeżeli P <0,5, predykcja to klasa = 0, w przeciwnym razie prognoza (P> = 0,5) jest równa klasie = 1.
Aby określić wartości wagi i odchylenia, musimy uzyskać zestaw danych uczących ze znanymi wartościami predyktorów wejściowych i znanymi prawidłowymi wartościami etykiet klas. Następnie możemy użyć algorytmu, ogólnie Gradient Descent, aby znaleźć wartości wagi i odchylenia.
Przykład implementacji modelu LR
W tym modelu LR będziemy używać następującego zestawu danych -
1.0, 2.0, 0
3.0, 4.0, 0
5.0, 2.0, 0
6.0, 3.0, 0
8.0, 1.0, 0
9.0, 2.0, 0
1.0, 4.0, 1
2.0, 5.0, 1
4.0, 6.0, 1
6.0, 5.0, 1
7.0, 3.0, 1
8.0, 5.0, 1Aby rozpocząć implementację tego modelu LR w CNTK, musimy najpierw zaimportować następujące pakiety -
import numpy as np
import cntk as CProgram ma strukturę main () w następujący sposób -
def main():
print("Using CNTK version = " + str(C.__version__) + "\n")Teraz musimy załadować dane treningowe do pamięci w następujący sposób -
data_file = ".\\dataLRmodel.txt"
print("Loading data from " + data_file + "\n")
features_mat = np.loadtxt(data_file, dtype=np.float32, delimiter=",", skiprows=0, usecols=[0,1])
labels_mat = np.loadtxt(data_file, dtype=np.float32, delimiter=",", skiprows=0, usecols=[2], ndmin=2)Teraz będziemy tworzyć program szkoleniowy, który tworzy model regresji logistycznej, który jest zgodny z danymi uczącymi -
features_dim = 2
labels_dim = 1
X = C.ops.input_variable(features_dim, np.float32)
y = C.input_variable(labels_dim, np.float32)
W = C.parameter(shape=(features_dim, 1)) # trainable cntk.Parameter
b = C.parameter(shape=(labels_dim))
z = C.times(X, W) + b
p = 1.0 / (1.0 + C.exp(-z))
model = pTeraz musimy stworzyć Lernera i trenera w następujący sposób -
ce_error = C.binary_cross_entropy(model, y) # CE a bit more principled for LR
fixed_lr = 0.010
learner = C.sgd(model.parameters, fixed_lr)
trainer = C.Trainer(model, (ce_error), [learner])
max_iterations = 4000Szkolenie LR Model
Kiedy już stworzyliśmy model LR, następnie czas przystąpić do szkolenia -
np.random.seed(4)
N = len(features_mat)
for i in range(0, max_iterations):
row = np.random.choice(N,1) # pick a random row from training items
trainer.train_minibatch({ X: features_mat[row], y: labels_mat[row] })
if i % 1000 == 0 and i > 0:
mcee = trainer.previous_minibatch_loss_average
print(str(i) + " Cross-entropy error on curr item = %0.4f " % mcee)Teraz za pomocą poniższego kodu możemy wydrukować wagi i odchylenie modelu -
np.set_printoptions(precision=4, suppress=True)
print("Model weights: ")
print(W.value)
print("Model bias:")
print(b.value)
print("")
if __name__ == "__main__":
main()Trenowanie modelu regresji logistycznej - pełny przykład
import numpy as np
import cntk as C
def main():
print("Using CNTK version = " + str(C.__version__) + "\n")
data_file = ".\\dataLRmodel.txt" # provide the name and the location of data file
print("Loading data from " + data_file + "\n")
features_mat = np.loadtxt(data_file, dtype=np.float32, delimiter=",", skiprows=0, usecols=[0,1])
labels_mat = np.loadtxt(data_file, dtype=np.float32, delimiter=",", skiprows=0, usecols=[2], ndmin=2)
features_dim = 2
labels_dim = 1
X = C.ops.input_variable(features_dim, np.float32)
y = C.input_variable(labels_dim, np.float32)
W = C.parameter(shape=(features_dim, 1)) # trainable cntk.Parameter
b = C.parameter(shape=(labels_dim))
z = C.times(X, W) + b
p = 1.0 / (1.0 + C.exp(-z))
model = p
ce_error = C.binary_cross_entropy(model, y) # CE a bit more principled for LR
fixed_lr = 0.010
learner = C.sgd(model.parameters, fixed_lr)
trainer = C.Trainer(model, (ce_error), [learner])
max_iterations = 4000
np.random.seed(4)
N = len(features_mat)
for i in range(0, max_iterations):
row = np.random.choice(N,1) # pick a random row from training items
trainer.train_minibatch({ X: features_mat[row], y: labels_mat[row] })
if i % 1000 == 0 and i > 0:
mcee = trainer.previous_minibatch_loss_average
print(str(i) + " Cross-entropy error on curr item = %0.4f " % mcee)
np.set_printoptions(precision=4, suppress=True)
print("Model weights: ")
print(W.value)
print("Model bias:")
print(b.value)
if __name__ == "__main__":
main()Wynik
Using CNTK version = 2.7
1000 cross entropy error on curr item = 0.1941
2000 cross entropy error on curr item = 0.1746
3000 cross entropy error on curr item = 0.0563
Model weights:
[-0.2049]
[0.9666]]
Model bias:
[-2.2846]Prognozowanie przy użyciu wyszkolonego modelu LR
Po przeszkoleniu modelu LR możemy go użyć do prognozowania w następujący sposób -
Przede wszystkim nasz program oceny importuje pakiet numpy i ładuje dane szkoleniowe do macierzy funkcji i macierzy etykiet klas w taki sam sposób, jak program szkoleniowy, który implementujemy powyżej -
import numpy as np
def main():
data_file = ".\\dataLRmodel.txt" # provide the name and the location of data file
features_mat = np.loadtxt(data_file, dtype=np.float32, delimiter=",",
skiprows=0, usecols=(0,1))
labels_mat = np.loadtxt(data_file, dtype=np.float32, delimiter=",",
skiprows=0, usecols=[2], ndmin=2)Następnie nadszedł czas, aby ustawić wartości wag i odchylenia, które zostały określone przez nasz program treningowy -
print("Setting weights and bias values \n")
weights = np.array([0.0925, 1.1722], dtype=np.float32)
bias = np.array([-4.5400], dtype=np.float32)
N = len(features_mat)
features_dim = 2Następnie nasz program oceny obliczy prawdopodobieństwo regresji logistycznej, przechodząc przez każdy element szkolenia w następujący sposób -
print("item pred_prob pred_label act_label result")
for i in range(0, N): # each item
x = features_mat[i]
z = 0.0
for j in range(0, features_dim):
z += x[j] * weights[j]
z += bias[0]
pred_prob = 1.0 / (1.0 + np.exp(-z))
pred_label = 0 if pred_prob < 0.5 else 1
act_label = labels_mat[i]
pred_str = ‘correct’ if np.absolute(pred_label - act_label) < 1.0e-5 \
else ‘WRONG’
print("%2d %0.4f %0.0f %0.0f %s" % \ (i, pred_prob, pred_label, act_label, pred_str))Teraz pokażemy, jak prognozować -
x = np.array([9.5, 4.5], dtype=np.float32)
print("\nPredicting class for age, education = ")
print(x)
z = 0.0
for j in range(0, features_dim):
z += x[j] * weights[j]
z += bias[0]
p = 1.0 / (1.0 + np.exp(-z))
print("Predicted p = " + str(p))
if p < 0.5: print("Predicted class = 0")
else: print("Predicted class = 1")Kompletny program oceny prognoz
import numpy as np
def main():
data_file = ".\\dataLRmodel.txt" # provide the name and the location of data file
features_mat = np.loadtxt(data_file, dtype=np.float32, delimiter=",",
skiprows=0, usecols=(0,1))
labels_mat = np.loadtxt(data_file, dtype=np.float32, delimiter=",",
skiprows=0, usecols=[2], ndmin=2)
print("Setting weights and bias values \n")
weights = np.array([0.0925, 1.1722], dtype=np.float32)
bias = np.array([-4.5400], dtype=np.float32)
N = len(features_mat)
features_dim = 2
print("item pred_prob pred_label act_label result")
for i in range(0, N): # each item
x = features_mat[i]
z = 0.0
for j in range(0, features_dim):
z += x[j] * weights[j]
z += bias[0]
pred_prob = 1.0 / (1.0 + np.exp(-z))
pred_label = 0 if pred_prob < 0.5 else 1
act_label = labels_mat[i]
pred_str = ‘correct’ if np.absolute(pred_label - act_label) < 1.0e-5 \
else ‘WRONG’
print("%2d %0.4f %0.0f %0.0f %s" % \ (i, pred_prob, pred_label, act_label, pred_str))
x = np.array([9.5, 4.5], dtype=np.float32)
print("\nPredicting class for age, education = ")
print(x)
z = 0.0
for j in range(0, features_dim):
z += x[j] * weights[j]
z += bias[0]
p = 1.0 / (1.0 + np.exp(-z))
print("Predicted p = " + str(p))
if p < 0.5: print("Predicted class = 0")
else: print("Predicted class = 1")
if __name__ == "__main__":
main()Wynik
Ustawianie wag i wartości odchylenia.
Item pred_prob pred_label act_label result
0 0.3640 0 0 correct
1 0.7254 1 0 WRONG
2 0.2019 0 0 correct
3 0.3562 0 0 correct
4 0.0493 0 0 correct
5 0.1005 0 0 correct
6 0.7892 1 1 correct
7 0.8564 1 1 correct
8 0.9654 1 1 correct
9 0.7587 1 1 correct
10 0.3040 0 1 WRONG
11 0.7129 1 1 correct
Predicting class for age, education =
[9.5 4.5]
Predicting p = 0.526487952
Predicting class = 1W tym rozdziale omówiono koncepcje sieci neuronowych w odniesieniu do CNTK.
Jak wiemy, do stworzenia sieci neuronowej wykorzystuje się kilka warstw neuronów. Powstaje jednak pytanie, w jaki sposób w CNTK możemy modelować warstwy NN? Można to zrobić za pomocą funkcji warstw zdefiniowanych w module warstw.
Funkcja warstwy
W rzeczywistości w CNTK praca z warstwami ma wyraźny charakter programowania funkcjonalnego. Funkcja warstwy wygląda jak zwykła funkcja i tworzy funkcję matematyczną z zestawem wstępnie zdefiniowanych parametrów. Zobaczmy, jak możemy stworzyć najbardziej podstawowy typ warstwy, Gęsty, za pomocą funkcji warstwy.
Przykład
Wykonując podstawowe czynności, możemy stworzyć najbardziej podstawowy typ warstwy -
Step 1 - Najpierw musimy zaimportować funkcję gęstej warstwy z pakietu warstw CNTK.
from cntk.layers import DenseStep 2 - Następnie z pakietu głównego CNTK musimy zaimportować funkcję input_variable.
from cntk import input_variableStep 3- Teraz musimy utworzyć nową zmienną wejściową za pomocą funkcji input_variable. Musimy również podać jego rozmiar.
feature = input_variable(100)Step 4 - Nareszcie stworzymy nową warstwę za pomocą funkcji Dense wraz z podaniem odpowiedniej liczby neuronów.
layer = Dense(40)(feature)Teraz możemy wywołać skonfigurowaną funkcję gęstej warstwy, aby podłączyć gęstą warstwę do wejścia.
Kompletny przykład wdrożenia
from cntk.layers import Dense
from cntk import input_variable
feature= input_variable(100)
layer = Dense(40)(feature)Dostosowywanie warstw
Jak widzieliśmy, CNTK zapewnia nam całkiem niezły zestaw ustawień domyślnych do budowania NN. Oparte naactivationfunkcji i innych wybranych przez nas ustawień, zachowanie i wydajność NN są inne. Jest to kolejny bardzo przydatny algorytm bazujący. Dlatego dobrze jest zrozumieć, co możemy skonfigurować.
Kroki, aby skonfigurować gęstą warstwę
Każda warstwa w NN ma swoje unikalne opcje konfiguracji, a kiedy mówimy o warstwie gęstej, mamy następujące ważne ustawienia do zdefiniowania -
shape - Jak sama nazwa wskazuje, definiuje wyjściowy kształt warstwy, który dodatkowo określa liczbę neuronów w tej warstwie.
activation - Definiuje funkcję aktywacji tej warstwy, dzięki czemu może przekształcić dane wejściowe.
init- Określa funkcję inicjalizacyjną tej warstwy. Zainicjalizuje parametry warstwy, gdy zaczniemy trenować NN.
Zobaczmy kroki, za pomocą których możemy skonfigurować plik Dense warstwa -
Step1 - Najpierw musimy zaimportować plik Dense funkcja warstwy z pakietu warstw CNTK.
from cntk.layers import DenseStep2 - Następnie z pakietu CNTK ops musimy zaimportować plik sigmoid operator. Będzie używany do konfiguracji jako funkcja aktywacji.
from cntk.ops import sigmoidStep3 - Teraz z pakietu inicjalizującego musimy zaimportować plik glorot_uniform inicjator.
from cntk.initializer import glorot_uniformStep4 - Nareszcie utworzymy nową warstwę używając funkcji Dense wraz z podaniem liczby neuronów jako pierwszego argumentu. Podaj równieżsigmoid operator jako activation funkcja i glorot_uniform jako init funkcja dla warstwy.
layer = Dense(50, activation = sigmoid, init = glorot_uniform)Kompletny przykład wdrożenia -
from cntk.layers import Dense
from cntk.ops import sigmoid
from cntk.initializer import glorot_uniform
layer = Dense(50, activation = sigmoid, init = glorot_uniform)Optymalizacja parametrów
Do tej pory widzieliśmy, jak tworzyć strukturę NN i konfigurować różne ustawienia. Tutaj zobaczymy, jak możemy zoptymalizować parametry sieci NN. Za pomocą połączenia dwóch składników, a mianowicielearners i trainers, możemy zoptymalizować parametry sieci NN.
składnik trenera
Pierwszym komponentem używanym do optymalizacji parametrów sieci NN jest trainerskładnik. Zasadniczo realizuje proces wstecznej propagacji. Jeśli mówimy o jego działaniu, przekazuje dane przez NN w celu uzyskania prognozy.
Następnie używa innego komponentu zwanego uczniem w celu uzyskania nowych wartości parametrów w sieci NN. Po uzyskaniu nowych wartości stosuje te nowe wartości i powtarza proces aż do spełnienia kryterium wyjścia.
składnik ucznia
Drugim składnikiem używanym do optymalizacji parametrów sieci NN jest learner składnik, który jest w zasadzie odpowiedzialny za wykonanie algorytmu zejścia gradientowego.
Osoby uczące się zawarte w bibliotece CNTK
Poniżej znajduje się lista niektórych interesujących uczniów zawartych w bibliotece CNTK -
Stochastic Gradient Descent (SGD) - Ten uczeń reprezentuje podstawowe stochastyczne zejście gradientu, bez żadnych dodatków.
Momentum Stochastic Gradient Descent (MomentumSGD) - Dzięki SGD ten uczeń wykorzystuje rozmach, aby przezwyciężyć problem lokalnych maksimów.
RMSProp - Ten uczeń, aby kontrolować tempo schodzenia, używa malejących wskaźników uczenia się.
Adam - Ten uczeń, aby zmniejszyć tempo opadania w czasie, wykorzystuje zanikający pęd.
Adagrad - Ten uczeń, zarówno w przypadku często, jak i rzadko występujących cech, stosuje różne wskaźniki uczenia się.
CNTK - Tworzenie pierwszej sieci neuronowej
W tym rozdziale zajmiemy się tworzeniem sieci neuronowej w CNTK.
Zbuduj strukturę sieci
Aby zastosować koncepcje CNTK do zbudowania naszego pierwszego NN, będziemy używać NN do klasyfikowania gatunków kwiatów tęczówki na podstawie właściwości fizycznych szerokości i długości działek oraz szerokości i długości płatków. Zbiór danych, którego będziemy używać, zbiór danych tęczówki, który opisuje właściwości fizyczne różnych odmian kwiatów tęczówki -
- Długość działki
- Szerokość działki
- Długość płatka
- Szerokość płatka
- Klasa tj. Iris setosa lub iris versicolor lub iris virginica
Tutaj będziemy budować zwykły NN zwany wyprzedzającym NN. Zobaczmy kroki implementacji, aby zbudować strukturę NN -
Step 1 - Najpierw zaimportujemy niezbędne komponenty, takie jak nasze typy warstw, funkcje aktywacji i funkcję, która pozwala nam zdefiniować zmienną wejściową dla naszego NN, z biblioteki CNTK.
from cntk import default_options, input_variable
from cntk.layers import Dense, Sequential
from cntk.ops import log_softmax, reluStep 2- Następnie stworzymy nasz model za pomocą funkcji sekwencyjnej. Po utworzeniu będziemy go karmić wybranymi warstwami. Tutaj utworzymy dwie odrębne warstwy w naszym NN; jeden z czterema neuronami, a drugi z trzema neuronami.
model = Sequential([Dense(4, activation=relu), Dense(3, activation=log_sogtmax)])Step 3- W końcu, aby skompilować NN, połączymy sieć ze zmienną wejściową. Posiada warstwę wejściową z czterema neuronami i warstwę wyjściową z trzema neuronami.
feature= input_variable(4)
z = model(feature)Stosowanie funkcji aktywacji
Istnieje wiele funkcji aktywacji do wyboru, a wybór odpowiedniej funkcji aktywacji z pewnością wpłynie na to, jak dobrze będzie działał nasz model głębokiego uczenia.
Na warstwie wyjściowej
Wybór activation funkcja w warstwie wyjściowej będzie zależeć od rodzaju problemu, który zamierzamy rozwiązać za pomocą naszego modelu.
W przypadku problemu regresji powinniśmy użyć linear activation function na warstwie wyjściowej.
W przypadku problemu z klasyfikacją binarną powinniśmy użyć pliku sigmoid activation function na warstwie wyjściowej.
W przypadku problemu klasyfikacji wieloklasowej powinniśmy użyć softmax activation function na warstwie wyjściowej.
Tutaj zbudujemy model do przewidywania jednej z trzech klas. Oznacza to, że musimy użyćsoftmax activation function w warstwie wyjściowej.
Na ukrytej warstwie
Wybór activation funkcja w warstwie ukrytej wymaga pewnych eksperymentów w celu monitorowania wydajności, aby zobaczyć, która funkcja aktywacji działa dobrze.
W przypadku problemu klasyfikacyjnego musimy przewidzieć prawdopodobieństwo, że próbka należy do określonej klasy. Dlatego potrzebujemyactivation functionto daje nam wartości probabilistyczne. Aby osiągnąć ten cel,sigmoid activation function może nam pomóc.
Jednym z głównych problemów związanych z funkcją sigmoidy jest problem znikającego gradientu. Aby przezwyciężyć taki problem, możemy skorzystaćReLU activation function który ukrywa wszystkie wartości ujemne do zera i działa jako filtr przechodzący dla wartości dodatnich.
Wybór funkcji straty
Kiedy już mamy strukturę naszego modelu NN, musimy ją zoptymalizować. Do optymalizacji potrzebujemy plikuloss function. w odróżnieniuactivation functions, mamy do wyboru bardzo mniej funkcji strat. Jednak wybór funkcji straty będzie zależał od rodzaju problemu, który zamierzamy rozwiązać za pomocą naszego modelu.
Na przykład w przypadku problemu klasyfikacji powinniśmy użyć funkcji straty, która może zmierzyć różnicę między przewidywaną klasą a klasą rzeczywistą.
funkcja straty
W przypadku problemu klasyfikacji rozwiążemy za pomocą naszego modelu NN, categorical cross entropyfunkcja straty jest najlepszym kandydatem. W CNTK jest zaimplementowany jakocross_entropy_with_softmax z których można importować cntk.losses pakiet w następujący sposób -
label= input_variable(3)
loss = cross_entropy_with_softmax(z, label)Metryka
Mając strukturę naszego modelu NN i funkcję straty do zastosowania, mamy wszystkie składniki, aby rozpocząć tworzenie przepisu na optymalizację naszego modelu uczenia głębokiego. Zanim jednak zagłębimy się w tę kwestię, powinniśmy poznać metryki.
cntk.metricsCNTK ma pakiet o nazwie cntk.metricsz którego możemy zaimportować metryki, których będziemy używać. Podczas budowania modelu klasyfikacji będziemy używaćclassification_error matric, który wygeneruje liczbę z przedziału od 0 do 1. Liczba od 0 do 1 wskazuje procent poprawnie przewidywanych próbek -
Najpierw musimy zaimportować metrykę z cntk.metrics pakiet -
from cntk.metrics import classification_error
error_rate = classification_error(z, label)Powyższa funkcja w rzeczywistości wymaga danych wyjściowych NN i oczekiwanej etykiety jako danych wejściowych.
CNTK - szkolenie sieci neuronowej
Tutaj zrozumiemy, jak trenować sieć neuronową w CNTK.
Trenowanie modelu w CNTK
W poprzedniej sekcji zdefiniowaliśmy wszystkie komponenty modelu uczenia głębokiego. Teraz czas go wyszkolić. Jak omówiliśmy wcześniej, możemy wytrenować model NN w CNTK przy użyciu kombinacjilearner i trainer.
Wybór osoby uczącej się i przygotowanie szkolenia
W tej sekcji będziemy definiować learner. CNTK zapewnia kilkalearnersdo wybrania z. W naszym modelu, zdefiniowanym w poprzednich sekcjach, będziemy używaćStochastic Gradient Descent (SGD) learner.
Aby wytrenować sieć neuronową, skonfigurujmy learner i trainer za pomocą następujących kroków -
Step 1 - Najpierw musimy importować sgd funkcja z cntk.lerners pakiet.
from cntk.learners import sgdStep 2 - Następnie musimy zaimportować Trainer funkcja z cntk.trainpakiet .trainer.
from cntk.train.trainer import TrainerStep 3 - Teraz musimy stworzyć plik learner. Można go utworzyć przez wywołaniesgd funkcji wraz z podaniem parametrów modelu i wartości współczynnika uczenia się.
learner = sgd(z.parametrs, 0.01)Step 4 - W końcu musimy zainicjować trainer. Należy podać sieć, kombinacjęloss i metric razem z learner.
trainer = Trainer(z, (loss, error_rate), [learner])Szybkość uczenia się, która steruje szybkością optymalizacji, powinna wynosić od 0,1 do 0,001.
Wybór osoby uczącej się i przygotowanie szkolenia - pełny przykład
from cntk.learners import sgd
from cntk.train.trainer import Trainer
learner = sgd(z.parametrs, 0.01)
trainer = Trainer(z, (loss, error_rate), [learner])Dostarczanie danych do trenera
Po wybraniu i skonfigurowaniu trenera czas na załadowanie zestawu danych. Uratowaliśmyiris zbiór danych jako plik.CSV plik i będziemy używać pakietu do zarządzania danymi o nazwie pandas aby załadować zbiór danych.
Kroki ładowania zestawu danych z pliku .CSV
Step 1 - Najpierw musimy zaimportować plik pandas pakiet.
from import pandas as pdStep 2 - Teraz musimy wywołać funkcję o nazwie read_csv funkcję załadowania pliku .csv z dysku.
df_source = pd.read_csv(‘iris.csv’, names = [‘sepal_length’, ‘sepal_width’,
‘petal_length’, ‘petal_width’, index_col=False)Po załadowaniu zbioru danych musimy podzielić go na zestaw funkcji i etykietę.
Kroki, aby podzielić zbiór danych na funkcje i etykietę
Step 1- Najpierw musimy wybrać wszystkie wiersze i pierwsze cztery kolumny ze zbioru danych. Można to zrobić za pomocąiloc funkcjonować.
x = df_source.iloc[:, :4].valuesStep 2- Następnie musimy wybrać kolumnę gatunku ze zbioru danych tęczówki. Będziemy używać właściwości values, aby uzyskać dostęp do elementu bazowegonumpy szyk.
x = df_source[‘species’].valuesKroki, aby zakodować kolumnę gatunków do liczbowej reprezentacji wektorowej
Jak omówiliśmy wcześniej, nasz model jest oparty na klasyfikacji, wymaga numerycznych wartości wejściowych. Dlatego tutaj musimy zakodować kolumnę gatunków do numerycznej reprezentacji wektorowej. Zobaczmy kroki, aby to zrobić -
Step 1- Najpierw musimy utworzyć wyrażenie listy, które będzie iterować po wszystkich elementach tablicy. Następnie wyszukaj każdą wartość w słowniku label_mapping.
label_mapping = {‘Iris-Setosa’ : 0, ‘Iris-Versicolor’ : 1, ‘Iris-Virginica’ : 2}Step 2- Następnie przekonwertuj tę przekonwertowaną wartość liczbową na jeden zakodowany wektor. Będziemy używaćone_hot działają w następujący sposób -
def one_hot(index, length):
result = np.zeros(length)
result[index] = 1
return resultStep 3 - W końcu musimy przekształcić tę przekonwertowaną listę w plik numpy szyk.
y = np.array([one_hot(label_mapping[v], 3) for v in y])Kroki umożliwiające wykrycie nadmiernego dopasowania
Sytuacja, w której model pamięta próbki, ale nie może wydedukować reguł z próbek uczących, jest nadmiernym dopasowaniem. Wykonując następujące kroki, możemy wykryć nadmierne dopasowanie w naszym modelu -
Step 1 - Najpierw z sklearn pakiet, zaimportuj plik train_test_split funkcja z model_selection moduł.
from sklearn.model_selection import train_test_splitStep 2 - Następnie musimy wywołać funkcję train_test_split z cechami x i etykietami y w następujący sposób -
x_train, x_test, y_train, y_test = train_test_split(X, y, test_size=0-2,
stratify=y)Określiliśmy test_size na 0,2, aby odłożyć na bok 20% wszystkich danych.
label_mapping = {‘Iris-Setosa’ : 0, ‘Iris-Versicolor’ : 1, ‘Iris-Virginica’ : 2}Kroki, aby przesłać zestaw szkoleniowy i zestaw walidacyjny do naszego modelu
Step 1 - Aby wytrenować nasz model, najpierw będziemy wywoływać train_minibatchmetoda. Następnie daj mu słownik, który odwzorowuje dane wejściowe na zmienną wejściową, której użyliśmy do zdefiniowania NN i związanej z nią funkcji straty.
trainer.train_minibatch({ features: X_train, label: y_train})Step 2 - Następnie zadzwoń train_minibatch używając następującej pętli for -
for _epoch in range(10):
trainer.train_minbatch ({ feature: X_train, label: y_train})
print(‘Loss: {}, Acc: {}’.format(
trainer.previous_minibatch_loss_average,
trainer.previous_minibatch_evaluation_average))Wprowadzanie danych do trenera - kompletny przykład
from import pandas as pd
df_source = pd.read_csv(‘iris.csv’, names = [‘sepal_length’, ‘sepal_width’, ‘petal_length’, ‘petal_width’, index_col=False)
x = df_source.iloc[:, :4].values
x = df_source[‘species’].values
label_mapping = {‘Iris-Setosa’ : 0, ‘Iris-Versicolor’ : 1, ‘Iris-Virginica’ : 2}
def one_hot(index, length):
result = np.zeros(length)
result[index] = 1
return result
y = np.array([one_hot(label_mapping[v], 3) for v in y])
from sklearn.model_selection import train_test_split
x_train, x_test, y_train, y_test = train_test_split(X, y, test_size=0-2, stratify=y)
label_mapping = {‘Iris-Setosa’ : 0, ‘Iris-Versicolor’ : 1, ‘Iris-Virginica’ : 2}
trainer.train_minibatch({ features: X_train, label: y_train})
for _epoch in range(10):
trainer.train_minbatch ({ feature: X_train, label: y_train})
print(‘Loss: {}, Acc: {}’.format(
trainer.previous_minibatch_loss_average,
trainer.previous_minibatch_evaluation_average))Pomiar wydajności NN
Aby zoptymalizować nasz model NN, ilekroć przekazujemy dane przez trenera, mierzy on wydajność modelu za pomocą metryki, którą skonfigurowaliśmy dla trenera. Taki pomiar wydajności modelu NN podczas treningu odbywa się na danych uczących. Ale z drugiej strony, do pełnej analizy wydajności modelu, potrzebujemy również danych testowych.
Tak więc, aby zmierzyć wydajność modelu przy użyciu danych testowych, możemy wywołać metodę test_minibatch metoda na trainer w następujący sposób -
trainer.test_minibatch({ features: X_test, label: y_test})Przewidywanie z NN
Po wytrenowaniu modelu uczenia głębokiego najważniejszą rzeczą jest zrobienie prognoz na jego podstawie. Aby dokonać prognozy na podstawie wyuczonego powyżej NN, możemy postępować zgodnie z podanymi krokami:
Step 1 - Najpierw musimy wybrać losową pozycję z zestawu testowego za pomocą następującej funkcji -
np.random.choiceStep 2 - Następnie musimy wybrać przykładowe dane ze zbioru testowego za pomocą sample_index.
Step 3 - Teraz, aby przekonwertować wyjście numeryczne na NN na rzeczywistą etykietę, utwórz odwrócone mapowanie.
Step 4 - Teraz użyj wybranego sampledane. Dokonaj prognozy, wywołując NN z jako funkcję.
Step 5- Teraz, kiedy otrzymasz przewidywane wyjście, weź indeks neuronu, który ma najwyższą wartość, jako wartość przewidywaną. Można to zrobić za pomocąnp.argmax funkcja z numpy pakiet.
Step 6 - Na koniec przekonwertuj wartość indeksu na prawdziwą etykietę za pomocą inverted_mapping.
Prognozowanie z NN - pełny przykład
sample_index = np.random.choice(X_test.shape[0])
sample = X_test[sample_index]
inverted_mapping = {
1:’Iris-setosa’,
2:’Iris-versicolor’,
3:’Iris-virginica’
}
prediction = z(sample)
predicted_label = inverted_mapping[np.argmax(prediction)]
print(predicted_label)Wynik
Po przeszkoleniu powyższego modelu uczenia głębokiego i uruchomieniu go uzyskasz następujący wynik -
Iris-versicolorCNTK - w pamięci i duże zbiory danych
W tym rozdziale dowiemy się, jak pracować z zapisanymi w pamięci i dużymi zbiorami danych w CNTK.
Trening z małymi zestawami danych w pamięci
Kiedy mówimy o podawaniu danych do trenera CNTK, może być wiele sposobów, ale będzie to zależeć od rozmiaru zbioru danych i formatu danych. Zestawy danych mogą być małymi w pamięci lub dużymi zbiorami danych.
W tej sekcji będziemy pracować z zestawami danych w pamięci. W tym celu użyjemy następujących dwóch frameworków -
- Numpy
- Pandas
Korzystanie z tablic Numpy
Tutaj będziemy pracować z losowo wygenerowanym zbiorem danych w CNTK. W tym przykładzie zamierzamy symulować dane dotyczące problemu klasyfikacji binarnej. Załóżmy, że mamy zestaw obserwacji z 4 cechami i chcemy przewidzieć dwie możliwe etykiety za pomocą naszego modelu uczenia głębokiego.
Przykład implementacji
W tym celu najpierw musimy wygenerować zestaw etykiet zawierający jedną gorącą wektorową reprezentację etykiet, które chcemy przewidzieć. Można to zrobić za pomocą następujących kroków -
Step 1 - Zaimportuj numpy pakiet w następujący sposób -
import numpy as np
num_samples = 20000Step 2 - Następnie wygeneruj mapowanie etykiety za pomocą np.eye działają w następujący sposób -
label_mapping = np.eye(2)Step 3 - Teraz używając np.random.choice funkcji, zbierz 20000 losowych próbek w następujący sposób -
y = label_mapping[np.random.choice(2,num_samples)].astype(np.float32)Step 4 - Teraz w końcu, używając funkcji np.random.random, wygeneruj tablicę losowych wartości zmiennoprzecinkowych w następujący sposób -
x = np.random.random(size=(num_samples, 4)).astype(np.float32)Po wygenerowaniu tablicy losowych wartości zmiennoprzecinkowych musimy przekonwertować je na 32-bitowe liczby zmiennoprzecinkowe, aby można było dopasować je do formatu oczekiwanego przez CNTK. Aby to zrobić, wykonaj poniższe czynności -
Step 5 - Zaimportuj funkcje warstwy gęstej i sekwencyjnej z modułu cntk.layers w następujący sposób -
from cntk.layers import Dense, SequentialStep 6- Teraz musimy zaimportować funkcję aktywacji dla warstw w sieci. Zaimportujmy pliksigmoid jako funkcja aktywacji -
from cntk import input_variable, default_options
from cntk.ops import sigmoidStep 7- Teraz musimy zaimportować funkcję strat, aby nauczyć sieć. Importujmybinary_cross_entropy jako funkcja straty -
from cntk.losses import binary_cross_entropyStep 8- Następnie musimy zdefiniować domyślne opcje sieci. Tutaj zapewnimysigmoidfunkcja aktywacji jako ustawienie domyślne. Utwórz również model za pomocą funkcji warstw sekwencyjnych w następujący sposób -
with default_options(activation=sigmoid):
model = Sequential([Dense(6),Dense(2)])Step 9 - Następnie zainicjalizuj plik input_variable z 4 funkcjami wejściowymi służącymi jako wejście dla sieci.
features = input_variable(4)Step 10 - Teraz, aby go ukończyć, musimy podłączyć zmienne funkcji do NN.
z = model(features)Więc teraz mamy NN, z pomocą następujących kroków, wytrenujmy go przy użyciu zestawu danych w pamięci -
Step 11 - Aby wytrenować ten NN, najpierw musimy zaimportować uczestnika z cntk.learnersmoduł. Sprowadzimysgd uczeń w następujący sposób -
from cntk.learners import sgdStep 12 - Wraz z tym importem ProgressPrinter od cntk.logging moduł również.
from cntk.logging import ProgressPrinter
progress_writer = ProgressPrinter(0)Step 13 - Następnie zdefiniuj nową zmienną wejściową dla etykiet w następujący sposób -
labels = input_variable(2)Step 14 - Aby wytrenować model NN, następnie musimy zdefiniować stratę za pomocą binary_cross_entropyfunkcjonować. Podaj również model z i zmienną etykiety.
loss = binary_cross_entropy(z, labels)Step 15 - Następnie zainicjuj plik sgd uczeń w następujący sposób -
learner = sgd(z.parameters, lr=0.1)Step 16- W końcu wywołaj metodę pociągu na funkcji straty. Podaj również dane wejściowe, pliksgd uczeń i progress_printer.−
training_summary=loss.train((x,y),parameter_learners=[learner],callbacks=[progress_writer])Kompletny przykład wdrożenia
import numpy as np
num_samples = 20000
label_mapping = np.eye(2)
y = label_mapping[np.random.choice(2,num_samples)].astype(np.float32)
x = np.random.random(size=(num_samples, 4)).astype(np.float32)
from cntk.layers import Dense, Sequential
from cntk import input_variable, default_options
from cntk.ops import sigmoid
from cntk.losses import binary_cross_entropy
with default_options(activation=sigmoid):
model = Sequential([Dense(6),Dense(2)])
features = input_variable(4)
z = model(features)
from cntk.learners import sgd
from cntk.logging import ProgressPrinter
progress_writer = ProgressPrinter(0)
labels = input_variable(2)
loss = binary_cross_entropy(z, labels)
learner = sgd(z.parameters, lr=0.1)
training_summary=loss.train((x,y),parameter_learners=[learner],callbacks=[progress_writer])Wynik
Build info:
Built time: *** ** **** 21:40:10
Last modified date: *** *** ** 21:08:46 2019
Build type: Release
Build target: CPU-only
With ASGD: yes
Math lib: mkl
Build Branch: HEAD
Build SHA1:ae9c9c7c5f9e6072cc9c94c254f816dbdc1c5be6 (modified)
MPI distribution: Microsoft MPI
MPI version: 7.0.12437.6
-------------------------------------------------------------------
average since average since examples
loss last metric last
------------------------------------------------------
Learning rate per minibatch: 0.1
1.52 1.52 0 0 32
1.51 1.51 0 0 96
1.48 1.46 0 0 224
1.45 1.42 0 0 480
1.42 1.4 0 0 992
1.41 1.39 0 0 2016
1.4 1.39 0 0 4064
1.39 1.39 0 0 8160
1.39 1.39 0 0 16352Korzystanie z Pandas DataFrames
Tablice Numpy są bardzo ograniczone pod względem tego, co mogą zawierać i są jednym z najbardziej podstawowych sposobów przechowywania danych. Na przykład pojedyncza tablica n-wymiarowa może zawierać dane jednego typu. Ale z drugiej strony w wielu rzeczywistych przypadkach potrzebujemy biblioteki, która może obsłużyć więcej niż jeden typ danych w jednym zbiorze danych.
Jedna z bibliotek Pythona o nazwie Pandas ułatwia pracę z tego rodzaju zbiorami danych. Wprowadza koncepcję DataFrame (DF) i pozwala nam ładować zestawy danych z dysku przechowywane w różnych formatach jako DF. Na przykład możemy czytać DF zapisane jako CSV, JSON, Excel itp.
Więcej szczegółów na temat biblioteki Python Pandas można znaleźć pod adresem https://www.tutorialspoint.com/python_pandas/index.htm.
Przykład implementacji
W tym przykładzie posłużymy się przykładem klasyfikacji trzech możliwych gatunków kwiatów tęczówki na podstawie czterech właściwości. Stworzyliśmy ten model głębokiego uczenia się również w poprzednich sekcjach. Model wygląda następująco -
from cntk.layers import Dense, Sequential
from cntk import input_variable, default_options
from cntk.ops import sigmoid, log_softmax
from cntk.losses import binary_cross_entropy
model = Sequential([
Dense(4, activation=sigmoid),
Dense(3, activation=log_softmax)
])
features = input_variable(4)
z = model(features)Powyższy model zawiera jedną ukrytą warstwę i warstwę wyjściową z trzema neuronami, aby dopasować liczbę klas, które możemy przewidzieć.
Następnie użyjemy train metoda i lossfunkcję uczenia sieci. W tym celu najpierw musimy załadować i wstępnie przetworzyć zestaw danych tęczówki, tak aby pasował do oczekiwanego układu i formatu danych dla sieci NN. Można to zrobić za pomocą następujących kroków -
Step 1 - Zaimportuj numpy i Pandas pakiet w następujący sposób -
import numpy as np
import pandas as pdStep 2 - Następnie użyj read_csv funkcja ładowania zbioru danych do pamięci -
df_source = pd.read_csv(‘iris.csv’, names = [‘sepal_length’, ‘sepal_width’,
‘petal_length’, ‘petal_width’, ‘species’], index_col=False)Step 3 - Teraz musimy stworzyć słownik, który będzie mapował etykiety w zbiorze danych wraz z ich reprezentacją numeryczną.
label_mapping = {‘Iris-Setosa’ : 0, ‘Iris-Versicolor’ : 1, ‘Iris-Virginica’ : 2}Step 4 - Teraz używając iloc indeksator w DataFrame, wybierz pierwsze cztery kolumny w następujący sposób -
x = df_source.iloc[:, :4].valuesStep 5−Następnie musimy wybrać kolumny gatunków jako etykiety zbioru danych. Można to zrobić w następujący sposób -
y = df_source[‘species’].valuesStep 6 - Teraz musimy zmapować etykiety w zbiorze danych, co można zrobić za pomocą label_mapping. Użyj równieżone_hot kodowanie, aby przekonwertować je na jedno gorące tablice kodowania.
y = np.array([one_hot(label_mapping[v], 3) for v in y])Step 7 - Następnie, aby użyć funkcji i zmapowanych etykiet z CNTK, musimy przekonwertować je oba na elementy zmiennoprzecinkowe -
x= x.astype(np.float32)
y= y.astype(np.float32)Jak wiemy, etykiety są przechowywane w zbiorze danych jako ciągi i CNTK nie może pracować z tymi ciągami. Z tego powodu potrzebuje zakodowanych na gorąco wektorów reprezentujących etykiety. W tym celu możemy zdefiniować funkcję powiedzone_hot w następujący sposób -
def one_hot(index, length):
result = np.zeros(length)
result[index] = index
return resultTeraz mamy tablicę numpy w odpowiednim formacie, z pomocą poniższych kroków możemy ich użyć do wytrenowania naszego modelu -
Step 8- Najpierw musimy zaimportować funkcję strat, aby wytrenować sieć. Importujmybinary_cross_entropy_with_softmax jako funkcja straty -
from cntk.losses import binary_cross_entropy_with_softmaxStep 9 - Aby wytrenować ten NN, musimy także zaimportować uczestnika z cntk.learnersmoduł. Sprowadzimysgd uczeń w następujący sposób -
from cntk.learners import sgdStep 10 - Wraz z tym importem ProgressPrinter od cntk.logging moduł również.
from cntk.logging import ProgressPrinter
progress_writer = ProgressPrinter(0)Step 11 - Następnie zdefiniuj nową zmienną wejściową dla etykiet w następujący sposób -
labels = input_variable(3)Step 12 - Aby wytrenować model NN, następnie musimy zdefiniować stratę za pomocą binary_cross_entropy_with_softmaxfunkcjonować. Podaj również model z i zmienną etykiety.
loss = binary_cross_entropy_with_softmax (z, labels)Step 13 - Następnie zainicjuj plik sgd uczeń w następujący sposób -
learner = sgd(z.parameters, 0.1)Step 14- W końcu wywołaj metodę pociągu na funkcji straty. Podaj również dane wejściowe, pliksgd uczeń i progress_printer.
training_summary=loss.train((x,y),parameter_learners=[learner],callbacks=
[progress_writer],minibatch_size=16,max_epochs=5)Kompletny przykład wdrożenia
from cntk.layers import Dense, Sequential
from cntk import input_variable, default_options
from cntk.ops import sigmoid, log_softmax
from cntk.losses import binary_cross_entropy
model = Sequential([
Dense(4, activation=sigmoid),
Dense(3, activation=log_softmax)
])
features = input_variable(4)
z = model(features)
import numpy as np
import pandas as pd
df_source = pd.read_csv(‘iris.csv’, names = [‘sepal_length’, ‘sepal_width’, ‘petal_length’, ‘petal_width’, ‘species’], index_col=False)
label_mapping = {‘Iris-Setosa’ : 0, ‘Iris-Versicolor’ : 1, ‘Iris-Virginica’ : 2}
x = df_source.iloc[:, :4].values
y = df_source[‘species’].values
y = np.array([one_hot(label_mapping[v], 3) for v in y])
x= x.astype(np.float32)
y= y.astype(np.float32)
def one_hot(index, length):
result = np.zeros(length)
result[index] = index
return result
from cntk.losses import binary_cross_entropy_with_softmax
from cntk.learners import sgd
from cntk.logging import ProgressPrinter
progress_writer = ProgressPrinter(0)
labels = input_variable(3)
loss = binary_cross_entropy_with_softmax (z, labels)
learner = sgd(z.parameters, 0.1)
training_summary=loss.train((x,y),parameter_learners=[learner],callbacks=[progress_writer],minibatch_size=16,max_epochs=5)Wynik
Build info:
Built time: *** ** **** 21:40:10
Last modified date: *** *** ** 21:08:46 2019
Build type: Release
Build target: CPU-only
With ASGD: yes
Math lib: mkl
Build Branch: HEAD
Build SHA1:ae9c9c7c5f9e6072cc9c94c254f816dbdc1c5be6 (modified)
MPI distribution: Microsoft MPI
MPI version: 7.0.12437.6
-------------------------------------------------------------------
average since average since examples
loss last metric last
------------------------------------------------------
Learning rate per minibatch: 0.1
1.1 1.1 0 0 16
0.835 0.704 0 0 32
1.993 1.11 0 0 48
1.14 1.14 0 0 112
[………]Szkolenie z dużymi zbiorami danych
W poprzedniej sekcji pracowaliśmy z małymi zestawami danych w pamięci przy użyciu Numpy i pand, ale nie wszystkie zestawy danych są tak małe. W szczególności zbiory danych zawierające obrazy, filmy, próbki dźwięków są duże.MinibatchSourceto komponent, który może ładować dane w fragmentach, dostarczany przez CNTK do pracy z tak dużymi zbiorami danych. Niektóre funkcjeMinibatchSource komponenty są następujące -
MinibatchSource może zapobiec nadmiernemu dopasowaniu NN przez automatyczne losowanie próbek odczytywanych ze źródła danych.
Posiada wbudowany potok transformacji, który może służyć do rozszerzania danych.
Ładuje dane w wątku w tle niezależnym od procesu uczenia.
W kolejnych sekcjach zamierzamy zbadać, jak używać źródła minibatch z danymi o braku pamięci do pracy z dużymi zestawami danych. Zbadamy również, jak możemy go użyć do karmienia w celu szkolenia NN.
Tworzenie instancji MinibatchSource
W poprzedniej sekcji użyliśmy przykładu kwiatu tęczówki i pracowaliśmy z małym zbiorem danych w pamięci przy użyciu Pandas DataFrames. Tutaj zastąpimy kod korzystający z danych z pandy DF naMinibatchSource. Najpierw musimy utworzyć wystąpienieMinibatchSource za pomocą następujących kroków -
Przykład implementacji
Step 1 - Najpierw z cntk.io moduł importuje komponenty dla minibatchsource w następujący sposób -
from cntk.io import StreamDef, StreamDefs, MinibatchSource, CTFDeserializer,
INFINITY_REPEATStep 2 - Teraz używając StreamDef class, utwórz definicję strumienia dla etykiet.
labels_stream = StreamDef(field=’labels’, shape=3, is_sparse=False)Step 3 - Następnie utwórz, aby odczytać funkcje złożone z pliku wejściowego, utwórz kolejną instancję StreamDef następująco.
feature_stream = StreamDef(field=’features’, shape=4, is_sparse=False)Step 4 - Teraz musimy to zapewnić iris.ctf plik jako dane wejściowe i zainicjuj plik deserializer w następujący sposób -
deserializer = CTFDeserializer(‘iris.ctf’, StreamDefs(labels=
label_stream, features=features_stream)Step 5 - Nareszcie musimy stworzyć instancję minisourceBatch używając deserializer w następujący sposób -
Minibatch_source = MinibatchSource(deserializer, randomize=True)Tworzenie instancji MinibatchSource - pełny przykład implementacji
from cntk.io import StreamDef, StreamDefs, MinibatchSource, CTFDeserializer, INFINITY_REPEAT
labels_stream = StreamDef(field=’labels’, shape=3, is_sparse=False)
feature_stream = StreamDef(field=’features’, shape=4, is_sparse=False)
deserializer = CTFDeserializer(‘iris.ctf’, StreamDefs(labels=label_stream, features=features_stream)
Minibatch_source = MinibatchSource(deserializer, randomize=True)Tworzenie pliku MCTF
Jak widzieliście powyżej, pobieramy dane z pliku „iris.ctf”. Ma format pliku o nazwie CNTK Text Format (CTF). Utworzenie pliku CTF jest obowiązkowe, aby uzyskać dane dlaMinibatchSourceprzykład, który stworzyliśmy powyżej. Zobaczmy, jak możemy utworzyć plik CTF.
Przykład implementacji
Step 1 - Najpierw musimy zaimportować pandy i paczki numpy w następujący sposób -
import pandas as pd
import numpy as npStep 2- Następnie musimy załadować do pamięci nasz plik z danymi, czyli iris.csv. Następnie umieść go wdf_source zmienna.
df_source = pd.read_csv(‘iris.csv’, names = [‘sepal_length’, ‘sepal_width’, ‘petal_length’, ‘petal_width’, ‘species’], index_col=False)Step 3 - Teraz używając ilocindeksator jako funkcje, weź zawartość pierwszych czterech kolumn. Wykorzystaj również dane z kolumny gatunków w następujący sposób -
features = df_source.iloc[: , :4].values
labels = df_source[‘species’].valuesStep 4- Następnie musimy utworzyć mapowanie między nazwą etykiety a jej reprezentacją numeryczną. Można to zrobić, tworząclabel_mapping w następujący sposób -
label_mapping = {‘Iris-Setosa’ : 0, ‘Iris-Versicolor’ : 1, ‘Iris-Virginica’ : 2}Step 5 - Teraz przekonwertuj etykiety na zestaw jeden-gorących zakodowanych wektorów w następujący sposób -
labels = [one_hot(label_mapping[v], 3) for v in labels]Teraz, tak jak poprzednio, utwórz funkcję narzędzia o nazwie one_hotzakodować etykiety. Można to zrobić w następujący sposób -
def one_hot(index, length):
result = np.zeros(length)
result[index] = 1
return resultPonieważ załadowaliśmy i wstępnie przetworzyliśmy dane, czas zapisać je na dysku w formacie pliku CTF. Możemy to zrobić za pomocą następującego kodu Pythona -
With open(‘iris.ctf’, ‘w’) as output_file:
for index in range(0, feature.shape[0]):
feature_values = ‘ ‘.join([str(x) for x in np.nditer(features[index])])
label_values = ‘ ‘.join([str(x) for x in np.nditer(labels[index])])
output_file.write(‘features {} | labels {} \n’.format(feature_values, label_values))Tworzenie pliku MCTF - przykład pełnego wdrożenia
import pandas as pd
import numpy as np
df_source = pd.read_csv(‘iris.csv’, names = [‘sepal_length’, ‘sepal_width’, ‘petal_length’, ‘petal_width’, ‘species’], index_col=False)
features = df_source.iloc[: , :4].values
labels = df_source[‘species’].values
label_mapping = {‘Iris-Setosa’ : 0, ‘Iris-Versicolor’ : 1, ‘Iris-Virginica’ : 2}
labels = [one_hot(label_mapping[v], 3) for v in labels]
def one_hot(index, length):
result = np.zeros(length)
result[index] = 1
return result
With open(‘iris.ctf’, ‘w’) as output_file:
for index in range(0, feature.shape[0]):
feature_values = ‘ ‘.join([str(x) for x in np.nditer(features[index])])
label_values = ‘ ‘.join([str(x) for x in np.nditer(labels[index])])
output_file.write(‘features {} | labels {} \n’.format(feature_values, label_values))Dostarczanie danych
Po utworzeniu MinibatchSource,na przykład, musimy go wyszkolić. Możemy użyć tej samej logiki treningowej, która była używana podczas pracy z małymi zestawami danych w pamięci. Tutaj użyjemyMinibatchSource przykład jako dane wejściowe dla metody pociągu w funkcji straty w następujący sposób -
Przykład implementacji
Step 1 - Aby zarejestrować wynik sesji szkoleniowej, najpierw zaimportuj ProgressPrinter z cntk.logging moduł w następujący sposób -
from cntk.logging import ProgressPrinterStep 2 - Następnie, aby skonfigurować sesję treningową, zaimportuj plik trainer i training_session od cntk.train moduł w następujący sposób -
from cntk.train import Trainer,Step 3 - Teraz musimy zdefiniować jakiś zestaw stałych, takich jak minibatch_size, samples_per_epoch i num_epochs w następujący sposób -
minbatch_size = 16
samples_per_epoch = 150
num_epochs = 30Step 4 - Następnie, aby wiedzieć, jak CNTK odczytuje dane podczas uczenia, musimy zdefiniować mapowanie między zmienną wejściową dla sieci a strumieniami w źródle minibatch.
input_map = {
features: minibatch.source.streams.features,
labels: minibatch.source.streams.features
}Step 5 - Następnie, aby zarejestrować dane wyjściowe procesu uczenia, zainicjuj plik progress_printer zmienna z nowym ProgressPrinter przykład w następujący sposób -
progress_writer = ProgressPrinter(0)Step 6 - W końcu musimy wywołać metodę pociągu na stracie w następujący sposób -
train_history = loss.train(minibatch_source,
parameter_learners=[learner],
model_inputs_to_streams=input_map,
callbacks=[progress_writer],
epoch_size=samples_per_epoch,
max_epochs=num_epochs)Dostarczanie danych - kompletny przykład wdrożenia
from cntk.logging import ProgressPrinter
from cntk.train import Trainer, training_session
minbatch_size = 16
samples_per_epoch = 150
num_epochs = 30
input_map = {
features: minibatch.source.streams.features,
labels: minibatch.source.streams.features
}
progress_writer = ProgressPrinter(0)
train_history = loss.train(minibatch_source,
parameter_learners=[learner],
model_inputs_to_streams=input_map,
callbacks=[progress_writer],
epoch_size=samples_per_epoch,
max_epochs=num_epochs)Wynik
-------------------------------------------------------------------
average since average since examples
loss last metric last
------------------------------------------------------
Learning rate per minibatch: 0.1
1.21 1.21 0 0 32
1.15 0.12 0 0 96
[………]CNTK - pomiar wydajności
W tym rozdziale wyjaśniono, jak mierzyć wydajność modelu w CNKT.
Strategia walidacji wydajności modelu
Po zbudowaniu modelu ML trenowaliśmy go przy użyciu zestawu próbek danych. Dzięki temu szkoleniu nasz model ML uczy się i wyprowadza kilka ogólnych zasad. Wydajność modelu ML ma znaczenie, gdy wprowadzamy do modelu nowe próbki, tj. Inne próbki niż dostarczone w czasie treningu. W takim przypadku model zachowuje się inaczej. Może być gorzej w zrobieniu dobrych prognoz na podstawie tych nowych próbek.
Ale model musi działać dobrze również dla nowych próbek, ponieważ w środowisku produkcyjnym otrzymamy inne dane wejściowe niż dane przykładowe, które wykorzystaliśmy do celów szkoleniowych. Z tego powodu powinniśmy zweryfikować model ML, używając zestawu próbek innych niż próbki, których używaliśmy do celów szkoleniowych. Tutaj omówimy dwie różne techniki tworzenia zbioru danych do walidacji NN.
Zbiór danych wstrzymanych
Jest to jedna z najłatwiejszych metod tworzenia zestawu danych do walidacji NN. Jak sama nazwa wskazuje, w tej metodzie będziemy wstrzymywać jeden zestaw próbek przed uczeniem (powiedzmy 20%) i używać go do testowania wydajności naszego modelu ML. Poniższy diagram przedstawia stosunek między próbkami uczącymi i walidacyjnymi -

Model zbioru danych typu Hold-out zapewnia, że mamy wystarczającą ilość danych do wytrenowania naszego modelu ML, a jednocześnie będziemy mieć rozsądną liczbę próbek, aby uzyskać dobry pomiar wydajności modelu.
Aby uwzględnić je w zestawie uczącym i zbiorze testowym, dobrą praktyką jest wybranie losowych próbek z głównego zbioru danych. Zapewnia równomierne rozłożenie między treningiem a zestawem testowym.
Poniżej znajduje się przykład, w którym tworzymy własny zbiór danych wstrzymanych przy użyciu train_test_split funkcja z scikit-learn biblioteka.
Przykład
from sklearn.datasets import load_iris
iris = load_iris()
X = iris.data
y = iris.target
from sklearn.model_selection import train_test_split
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2, random_state=1)
# Here above test_size = 0.2 represents that we provided 20% of the data as test data.
from sklearn.neighbors import KNeighborsClassifier
from sklearn import metrics
classifier_knn = KNeighborsClassifier(n_neighbors=3)
classifier_knn.fit(X_train, y_train)
y_pred = classifier_knn.predict(X_test)
# Providing sample data and the model will make prediction out of that data
sample = [[5, 5, 3, 2], [2, 4, 3, 5]]
preds = classifier_knn.predict(sample)
pred_species = [iris.target_names[p] for p in preds] print("Predictions:", pred_species)Wynik
Predictions: ['versicolor', 'virginica']Korzystając z CNTK, musimy losowo zmieniać kolejność naszego zestawu danych za każdym razem, gdy trenujemy nasz model, ponieważ -
Na algorytmy głębokiego uczenia się duży wpływ mają generatory liczb losowych.
Kolejność, w jakiej dostarczamy próbki do NN podczas treningu, ma duży wpływ na jego wydajność.
Główną wadą stosowania techniki wstrzymywania zbioru danych jest to, że jest niewiarygodna, ponieważ czasami uzyskujemy bardzo dobre wyniki, ale czasami otrzymujemy złe wyniki.
K-krotna weryfikacja krzyżowa
Aby uczynić nasz model ML bardziej niezawodnym, istnieje technika zwana walidacją krzyżową K-fold. W naturze technika walidacji krzyżowej K-krotnej jest taka sama jak poprzednia technika, ale powtarza ją kilka razy - zwykle około 5 do 10 razy. Poniższy diagram przedstawia jego koncepcję -

Praca z walidacją krzyżową K-fold
Działanie walidacji krzyżowej K-fold można zrozumieć za pomocą następujących kroków:
Step 1- Podobnie jak w przypadku techniki Hand-out dataset, w technice walidacji krzyżowej K-fold, najpierw musimy podzielić zbiór danych na zbiór uczący i testowy. Idealnie, stosunek ten wynosi 80-20, czyli 80% zbioru uczącego i 20% zbioru testowego.
Step 2 - Następnie musimy wytrenować nasz model za pomocą zestawu uczącego.
Step 3- Na koniec będziemy używać zestawu testowego do pomiaru wydajności naszego modelu. Jedyną różnicą między techniką zbioru danych Hold-out a techniką walidacji k-cross jest to, że powyższy proces jest powtarzany zwykle od 5 do 10 razy, a na koniec obliczana jest średnia ze wszystkich wskaźników wydajności. Ta średnia byłaby ostatecznym miernikiem wydajności.
Zobaczmy przykład z małym zestawem danych -
Przykład
from numpy import array
from sklearn.model_selection import KFold
data = array([0.1, 0.2, 0.3, 0.4, 0.5, 0.6, 0.7, 0.8, 0.9, 1.0])
kfold = KFold(5, True, 1)
for train, test in kfold.split(data):
print('train: %s, test: %s' % (data[train],(data[test]))Wynik
train: [0.1 0.2 0.4 0.5 0.6 0.7 0.8 0.9], test: [0.3 1. ]
train: [0.1 0.2 0.3 0.4 0.6 0.8 0.9 1. ], test: [0.5 0.7]
train: [0.2 0.3 0.5 0.6 0.7 0.8 0.9 1. ], test: [0.1 0.4]
train: [0.1 0.3 0.4 0.5 0.6 0.7 0.9 1. ], test: [0.2 0.8]
train: [0.1 0.2 0.3 0.4 0.5 0.7 0.8 1. ], test: [0.6 0.9]Jak widać, dzięki zastosowaniu bardziej realistycznego scenariusza szkolenia i testów, technika walidacji krzyżowej k-krotnej daje nam znacznie bardziej stabilny pomiar wydajności, ale z drugiej strony, walidacja modeli uczenia głębokiego zajmuje dużo czasu.
CNTK nie obsługuje walidacji k-cross, dlatego musimy napisać własny skrypt, aby to zrobić.
Wykrywanie niedopasowania i nadmiernego dopasowania
Niezależnie od tego, czy używamy zestawu danych Hand-out, czy techniki k-krotnej walidacji krzyżowej, odkryjemy, że dane wyjściowe dla metryk będą różne dla zestawu danych używanego do uczenia i zestawu danych używanego do walidacji.
Wykrywanie nadmiernego dopasowania
Zjawisko zwane overfittingiem to sytuacja, w której nasz model ML wyjątkowo dobrze modeluje dane treningowe, ale nie działa dobrze na danych testowych, tj. Nie był w stanie przewidzieć danych testowych.
Dzieje się tak, gdy model ML uczy się określonego wzorca i szumu z danych uczących w takim stopniu, że wpływa to negatywnie na zdolność tego modelu do uogólniania z danych szkoleniowych na nowe, tj. Niewidoczne dane. Tutaj szum jest nieistotną informacją lub losowością w zbiorze danych.
Oto dwa sposoby, za pomocą których możemy wykryć, czy nasz model jest przepasowany lub nie -
Model overfit będzie działał dobrze na tych samych próbkach, których używaliśmy do treningu, ale będzie działał bardzo źle na nowych próbkach, tj. Próbkach innych niż treningowe.
Model jest nadmiernie dopasowany podczas walidacji, jeśli metryka w zestawie testowym jest niższa niż ta sama metryka, której używamy w naszym zbiorze uczącym.
Wykrywanie niedopasowania
Inną sytuacją, która może wystąpić w naszym ML, jest niedopasowanie. Jest to sytuacja, w której nasz model ML nie modelował dobrze danych szkoleniowych i nie przewiduje przydatnych wyników. Kiedy zaczniemy trenować pierwszą epokę, nasz model będzie niedopasowany, ale będzie mniej dopasowany w miarę postępów w treningu.
Jednym ze sposobów wykrycia, czy nasz model jest niedopasowany, czy nie, jest przyjrzenie się metrykom zestawu uczącego i zestawu testowego. Nasz model będzie niedopasowany, jeśli metryka na zbiorze testowym jest wyższa niż metryka na zbiorze uczącym.
CNTK - klasyfikacja sieci neuronowych
W tym rozdziale zajmiemy się klasyfikacją sieci neuronowych za pomocą CNTK.
Wprowadzenie
Klasyfikację można zdefiniować jako proces przewidywania kategorialnych etykiet wyjściowych lub odpowiedzi dla danych wejściowych. Skategoryzowane dane wyjściowe, które będą oparte na tym, czego model nauczył się w fazie szkolenia, mogą mieć postać, na przykład „Czarny” lub „Biały”, „Spam” lub „Brak spamu”.
Z drugiej strony, matematycznie, jest to zadanie przybliżenia funkcji mapującej, powiedzmy f ze zmiennych wejściowych powiedz X do zmiennych wyjściowych powiedz Y.
Klasycznym przykładem problemu klasyfikacyjnego może być wykrywanie spamu w wiadomościach e-mail. Oczywiste jest, że mogą istnieć tylko dwie kategorie wyników, „spam” i „brak spamu”.
Aby zaimplementować taką klasyfikację, musimy najpierw przeprowadzić szkolenie klasyfikatora, w którym wiadomości e-mail „spam” i „bez spamu” byłyby używane jako dane szkoleniowe. Po pomyślnym przeszkoleniu klasyfikator może zostać użyty do wykrycia nieznanej wiadomości e-mail.
Tutaj utworzymy 4-5-3 NN przy użyciu zestawu danych kwiatu tęczówki, który ma następujące parametry -
Węzły 4-wejściowe (po jednym dla każdej wartości predyktora).
5-ukrytych węzłów przetwarzania.
Węzły 3-wyjściowe (ponieważ w zbiorze danych tęczówki istnieją trzy możliwe gatunki).
Ładowanie zbioru danych
Będziemy korzystać z zestawu danych o kwiatach irysa, na podstawie którego będziemy chcieli sklasyfikować gatunki kwiatów irysa na podstawie fizycznych właściwości szerokości i długości działek oraz szerokości i długości płatków. Zbiór danych opisuje właściwości fizyczne różnych odmian kwiatów tęczówki -
Długość działki
Szerokość działki
Długość płatka
Szerokość płatka
Klasa tj. Iris setosa lub iris versicolor lub iris virginica
Mamy iris.CSVplik, którego używaliśmy również wcześniej w poprzednich rozdziałach. Można go załadować za pomocąPandasbiblioteka. Ale zanim go użyjemy lub załadujemy do naszego klasyfikatora, musimy przygotować pliki szkoleniowe i testowe, aby można go było łatwo używać z CNTK.
Przygotowywanie plików szkoleniowych i testowych
Zbiór danych Iris jest jednym z najpopularniejszych zbiorów danych dla projektów ML. Zawiera 150 pozycji danych, a surowe dane wyglądają następująco -
5.1 3.5 1.4 0.2 setosa
4.9 3.0 1.4 0.2 setosa
…
7.0 3.2 4.7 1.4 versicolor
6.4 3.2 4.5 1.5 versicolor
…
6.3 3.3 6.0 2.5 virginica
5.8 2.7 5.1 1.9 virginicaJak powiedziano wcześniej, pierwsze cztery wartości w każdej linii opisują właściwości fizyczne różnych odmian, tj. Długość działki, szerokość działki, długość płatka, szerokość płatka tęczówki.
Ale powinniśmy przekonwertować dane do formatu, który może być łatwo użyty przez CNTK, a tym formatem jest plik .ctf (utworzyliśmy również jeden iris.ctf w poprzedniej sekcji). Będzie wyglądać następująco -
|attribs 5.1 3.5 1.4 0.2|species 1 0 0
|attribs 4.9 3.0 1.4 0.2|species 1 0 0
…
|attribs 7.0 3.2 4.7 1.4|species 0 1 0
|attribs 6.4 3.2 4.5 1.5|species 0 1 0
…
|attribs 6.3 3.3 6.0 2.5|species 0 0 1
|attribs 5.8 2.7 5.1 1.9|species 0 0 1W powyższych danych znacznik | atrybuty oznacza początek wartości cechy, a | gatunek oznacza wartości etykiety klasy. Możemy również użyć dowolnych innych nazw tagów, nawet jeśli możemy dodać identyfikator przedmiotu. Na przykład spójrz na następujące dane -
|ID 001 |attribs 5.1 3.5 1.4 0.2|species 1 0 0 |#setosa
|ID 002 |attribs 4.9 3.0 1.4 0.2|species 1 0 0 |#setosa
…
|ID 051 |attribs 7.0 3.2 4.7 1.4|species 0 1 0 |#versicolor
|ID 052 |attribs 6.4 3.2 4.5 1.5|species 0 1 0 |#versicolor
…W zbiorze danych tęczówki znajduje się łącznie 150 pozycji danych i dla tego przykładu będziemy używać reguły zbioru danych 80–20 wstrzymanych, tj. 80% (120 elementów) elementów danych do celów szkoleniowych i pozostałe 20% (30 elementów) elementów danych do testowania cel, powód.
Konstruowanie modelu klasyfikacyjnego
Najpierw musimy przetworzyć pliki danych w formacie CNTK i do tego użyjemy funkcji pomocniczej o nazwie create_reader w następujący sposób -
def create_reader(path, input_dim, output_dim, rnd_order, sweeps):
x_strm = C.io.StreamDef(field='attribs', shape=input_dim, is_sparse=False)
y_strm = C.io.StreamDef(field='species', shape=output_dim, is_sparse=False)
streams = C.io.StreamDefs(x_src=x_strm, y_src=y_strm)
deserial = C.io.CTFDeserializer(path, streams)
mb_src = C.io.MinibatchSource(deserial, randomize=rnd_order, max_sweeps=sweeps)
return mb_srcTeraz musimy ustawić argumenty architektury dla naszego NN, a także podać lokalizację plików danych. Można to zrobić za pomocą następującego kodu Pythona -
def main():
print("Using CNTK version = " + str(C.__version__) + "\n")
input_dim = 4
hidden_dim = 5
output_dim = 3
train_file = ".\\...\\" #provide the name of the training file(120 data items)
test_file = ".\\...\\" #provide the name of the test file(30 data items)Teraz przy pomocy następującego wiersza kodu nasz program utworzy nieprzeszkolony NN -
X = C.ops.input_variable(input_dim, np.float32)
Y = C.ops.input_variable(output_dim, np.float32)
with C.layers.default_options(init=C.initializer.uniform(scale=0.01, seed=1)):
hLayer = C.layers.Dense(hidden_dim, activation=C.ops.tanh, name='hidLayer')(X)
oLayer = C.layers.Dense(output_dim, activation=None, name='outLayer')(hLayer)
nnet = oLayer
model = C.ops.softmax(nnet)Teraz, gdy stworzyliśmy podwójny nietrenowany model, musimy skonfigurować obiekt algorytmu ucznia, a następnie użyć go do utworzenia obiektu szkoleniowego Trainer. Będziemy używać uczącego się SGD icross_entropy_with_softmax funkcja straty -
tr_loss = C.cross_entropy_with_softmax(nnet, Y)
tr_clas = C.classification_error(nnet, Y)
max_iter = 2000
batch_size = 10
learn_rate = 0.01
learner = C.sgd(nnet.parameters, learn_rate)
trainer = C.Trainer(nnet, (tr_loss, tr_clas), [learner])Zakoduj algorytm uczenia się w następujący sposób -
max_iter = 2000
batch_size = 10
lr_schedule = C.learning_parameter_schedule_per_sample([(1000, 0.05), (1, 0.01)])
mom_sch = C.momentum_schedule([(100, 0.99), (0, 0.95)], batch_size)
learner = C.fsadagrad(nnet.parameters, lr=lr_schedule, momentum=mom_sch)
trainer = C.Trainer(nnet, (tr_loss, tr_clas), [learner])Teraz, kiedy skończyliśmy pracę z obiektem Trainer, musimy stworzyć funkcję czytnika, aby odczytać dane szkoleniowe
rdr = create_reader(train_file, input_dim, output_dim, rnd_order=True, sweeps=C.io.INFINITELY_REPEAT)
iris_input_map = { X : rdr.streams.x_src, Y : rdr.streams.y_src }Teraz czas na szkolenie naszego modelu NN−
for i in range(0, max_iter):
curr_batch = rdr.next_minibatch(batch_size, input_map=iris_input_map) trainer.train_minibatch(curr_batch)
if i % 500 == 0:
mcee = trainer.previous_minibatch_loss_average
macc = (1.0 - trainer.previous_minibatch_evaluation_average) * 100
print("batch %4d: mean loss = %0.4f, accuracy = %0.2f%% " \ % (i, mcee, macc))Kiedy już skończyliśmy z uczeniem, oceńmy model przy użyciu elementów danych testowych -
print("\nEvaluating test data \n")
rdr = create_reader(test_file, input_dim, output_dim, rnd_order=False, sweeps=1)
iris_input_map = { X : rdr.streams.x_src, Y : rdr.streams.y_src }
num_test = 30
all_test = rdr.next_minibatch(num_test, input_map=iris_input_map) acc = (1.0 - trainer.test_minibatch(all_test)) * 100
print("Classification accuracy = %0.2f%%" % acc)Po ocenie dokładności naszego wytrenowanego modelu NN będziemy go używać do prognozowania niewidocznych danych -
np.set_printoptions(precision = 1, suppress=True)
unknown = np.array([[6.4, 3.2, 4.5, 1.5]], dtype=np.float32)
print("\nPredicting Iris species for input features: ")
print(unknown[0]) pred_prob = model.eval(unknown)
np.set_printoptions(precision = 4, suppress=True)
print("Prediction probabilities are: ")
print(pred_prob[0])Kompletny model klasyfikacji
Import numpy as np
Import cntk as C
def create_reader(path, input_dim, output_dim, rnd_order, sweeps):
x_strm = C.io.StreamDef(field='attribs', shape=input_dim, is_sparse=False)
y_strm = C.io.StreamDef(field='species', shape=output_dim, is_sparse=False)
streams = C.io.StreamDefs(x_src=x_strm, y_src=y_strm)
deserial = C.io.CTFDeserializer(path, streams)
mb_src = C.io.MinibatchSource(deserial, randomize=rnd_order, max_sweeps=sweeps)
return mb_src
def main():
print("Using CNTK version = " + str(C.__version__) + "\n")
input_dim = 4
hidden_dim = 5
output_dim = 3
train_file = ".\\...\\" #provide the name of the training file(120 data items)
test_file = ".\\...\\" #provide the name of the test file(30 data items)
X = C.ops.input_variable(input_dim, np.float32)
Y = C.ops.input_variable(output_dim, np.float32)
with C.layers.default_options(init=C.initializer.uniform(scale=0.01, seed=1)):
hLayer = C.layers.Dense(hidden_dim, activation=C.ops.tanh, name='hidLayer')(X)
oLayer = C.layers.Dense(output_dim, activation=None, name='outLayer')(hLayer)
nnet = oLayer
model = C.ops.softmax(nnet)
tr_loss = C.cross_entropy_with_softmax(nnet, Y)
tr_clas = C.classification_error(nnet, Y)
max_iter = 2000
batch_size = 10
learn_rate = 0.01
learner = C.sgd(nnet.parameters, learn_rate)
trainer = C.Trainer(nnet, (tr_loss, tr_clas), [learner])
max_iter = 2000
batch_size = 10
lr_schedule = C.learning_parameter_schedule_per_sample([(1000, 0.05), (1, 0.01)])
mom_sch = C.momentum_schedule([(100, 0.99), (0, 0.95)], batch_size)
learner = C.fsadagrad(nnet.parameters, lr=lr_schedule, momentum=mom_sch)
trainer = C.Trainer(nnet, (tr_loss, tr_clas), [learner])
rdr = create_reader(train_file, input_dim, output_dim, rnd_order=True, sweeps=C.io.INFINITELY_REPEAT)
iris_input_map = { X : rdr.streams.x_src, Y : rdr.streams.y_src }
for i in range(0, max_iter):
curr_batch = rdr.next_minibatch(batch_size, input_map=iris_input_map) trainer.train_minibatch(curr_batch)
if i % 500 == 0:
mcee = trainer.previous_minibatch_loss_average
macc = (1.0 - trainer.previous_minibatch_evaluation_average) * 100
print("batch %4d: mean loss = %0.4f, accuracy = %0.2f%% " \ % (i, mcee, macc))
print("\nEvaluating test data \n")
rdr = create_reader(test_file, input_dim, output_dim, rnd_order=False, sweeps=1)
iris_input_map = { X : rdr.streams.x_src, Y : rdr.streams.y_src }
num_test = 30
all_test = rdr.next_minibatch(num_test, input_map=iris_input_map) acc = (1.0 - trainer.test_minibatch(all_test)) * 100
print("Classification accuracy = %0.2f%%" % acc)
np.set_printoptions(precision = 1, suppress=True)
unknown = np.array([[7.0, 3.2, 4.7, 1.4]], dtype=np.float32)
print("\nPredicting species for input features: ")
print(unknown[0])
pred_prob = model.eval(unknown)
np.set_printoptions(precision = 4, suppress=True)
print("Prediction probabilities: ")
print(pred_prob[0])
if __name__== ”__main__”:
main()Wynik
Using CNTK version = 2.7
batch 0: mean loss = 1.0986, mean accuracy = 40.00%
batch 500: mean loss = 0.6677, mean accuracy = 80.00%
batch 1000: mean loss = 0.5332, mean accuracy = 70.00%
batch 1500: mean loss = 0.2408, mean accuracy = 100.00%
Evaluating test data
Classification accuracy = 94.58%
Predicting species for input features:
[7.0 3.2 4.7 1.4]
Prediction probabilities:
[0.0847 0.736 0.113]Zapisywanie wytrenowanego modelu
Ten zestaw danych Iris zawiera tylko 150 elementów danych, dlatego wytrenowanie modelu klasyfikatora NN zajmie tylko kilka sekund, ale uczenie na dużym zestawie danych zawierającym sto lub tysiące elementów danych może zająć godziny lub nawet dni.
Możemy zapisać nasz model, aby nie musieć zachowywać go od zera. Z pomocą śledzenia kodu Pythona możemy zapisać nasz wyszkolony NN -
nn_classifier = “.\\neuralclassifier.model” #provide the name of the file
model.save(nn_classifier, format=C.ModelFormat.CNTKv2)Poniżej przedstawiono argumenty save() funkcja używana powyżej -
Nazwa pliku jest pierwszym argumentem save()funkcjonować. Można go również zapisywać wraz ze ścieżką do pliku.
Kolejnym parametrem jest format parametr, który ma wartość domyślną C.ModelFormat.CNTKv2.
Ładowanie wytrenowanego modelu
Po zapisaniu wytrenowanego modelu bardzo łatwo jest go załadować. Musimy tylko użyćload ()funkcjonować. Sprawdźmy to w poniższym przykładzie -
import numpy as np
import cntk as C
model = C.ops.functions.Function.load(“.\\neuralclassifier.model”)
np.set_printoptions(precision = 1, suppress=True)
unknown = np.array([[7.0, 3.2, 4.7, 1.4]], dtype=np.float32)
print("\nPredicting species for input features: ")
print(unknown[0])
pred_prob = model.eval(unknown)
np.set_printoptions(precision = 4, suppress=True)
print("Prediction probabilities: ")
print(pred_prob[0])Zaletą zapisanego modelu jest to, że po załadowaniu zapisanego modelu można go używać dokładnie tak, jakby model został właśnie wytrenowany.
CNTK - klasyfikacja binarna sieci neuronowej
Zrozummy, czym jest binarna klasyfikacja sieci neuronowych za pomocą CNTK w tym rozdziale.
Klasyfikacja binarna przy użyciu NN jest jak klasyfikacja wieloklasowa, jedyną rzeczą jest to, że istnieją tylko dwa węzły wyjściowe zamiast trzech lub więcej. Tutaj przeprowadzimy klasyfikację binarną za pomocą sieci neuronowej przy użyciu dwóch technik, a mianowicie techniki jedno- i dwuwęzłowej. Technika z jednym węzłem jest bardziej powszechna niż technika z dwoma węzłami.
Ładowanie zbioru danych
Aby obie te techniki wdrożyć przy użyciu NN, będziemy używać zbioru danych banknotów. Zestaw danych można pobrać z repozytorium UCI Machine Learning Repository, które jest dostępne pod adresemhttps://archive.ics.uci.edu/ml/datasets/banknote+authentication.
W naszym przykładzie użyjemy 50 autentycznych elementów danych z klasą fałszerstwa = 0 i pierwszych 50 fałszywych elementów z klasą fałszerstwa = 1.
Przygotowywanie plików szkoleniowych i testowych
Pełny zbiór danych zawiera 1372 pozycje danych. Surowy zbiór danych wygląda następująco -
3.6216, 8.6661, -2.8076, -0.44699, 0
4.5459, 8.1674, -2.4586, -1.4621, 0
…
-1.3971, 3.3191, -1.3927, -1.9948, 1
0.39012, -0.14279, -0.031994, 0.35084, 1Teraz najpierw musimy przekonwertować te surowe dane na dwuwęzłowy format CNTK, który wyglądałby następująco -
|stats 3.62160000 8.66610000 -2.80730000 -0.44699000 |forgery 0 1 |# authentic
|stats 4.54590000 8.16740000 -2.45860000 -1.46210000 |forgery 0 1 |# authentic
. . .
|stats -1.39710000 3.31910000 -1.39270000 -1.99480000 |forgery 1 0 |# fake
|stats 0.39012000 -0.14279000 -0.03199400 0.35084000 |forgery 1 0 |# fakeMożesz użyć następującego programu w języku Python do tworzenia danych w formacie CNTK z danych surowych -
fin = open(".\\...", "r") #provide the location of saved dataset text file.
for line in fin:
line = line.strip()
tokens = line.split(",")
if tokens[4] == "0":
print("|stats %12.8f %12.8f %12.8f %12.8f |forgery 0 1 |# authentic" % \
(float(tokens[0]), float(tokens[1]), float(tokens[2]), float(tokens[3])) )
else:
print("|stats %12.8f %12.8f %12.8f %12.8f |forgery 1 0 |# fake" % \
(float(tokens[0]), float(tokens[1]), float(tokens[2]), float(tokens[3])) )
fin.close()Dwuwęzłowy binarny model klasyfikacji
Istnieje bardzo niewielka różnica między klasyfikacją dwuwęzłową a klasyfikacją wieloklasową. Tutaj najpierw musimy przetworzyć pliki danych w formacie CNTK i do tego użyjemy funkcji pomocniczej o nazwiecreate_reader w następujący sposób -
def create_reader(path, input_dim, output_dim, rnd_order, sweeps):
x_strm = C.io.StreamDef(field='stats', shape=input_dim, is_sparse=False)
y_strm = C.io.StreamDef(field='forgery', shape=output_dim, is_sparse=False)
streams = C.io.StreamDefs(x_src=x_strm, y_src=y_strm)
deserial = C.io.CTFDeserializer(path, streams)
mb_src = C.io.MinibatchSource(deserial, randomize=rnd_order, max_sweeps=sweeps)
return mb_srcTeraz musimy ustawić argumenty architektury dla naszego NN, a także podać lokalizację plików danych. Można to zrobić za pomocą następującego kodu Pythona -
def main():
print("Using CNTK version = " + str(C.__version__) + "\n")
input_dim = 4
hidden_dim = 10
output_dim = 2
train_file = ".\\...\\" #provide the name of the training file
test_file = ".\\...\\" #provide the name of the test fileTeraz przy pomocy następującego wiersza kodu nasz program utworzy nieprzeszkolony NN -
X = C.ops.input_variable(input_dim, np.float32)
Y = C.ops.input_variable(output_dim, np.float32)
with C.layers.default_options(init=C.initializer.uniform(scale=0.01, seed=1)):
hLayer = C.layers.Dense(hidden_dim, activation=C.ops.tanh, name='hidLayer')(X)
oLayer = C.layers.Dense(output_dim, activation=None, name='outLayer')(hLayer)
nnet = oLayer
model = C.ops.softmax(nnet)Teraz, gdy stworzyliśmy podwójny nietrenowany model, musimy skonfigurować obiekt algorytmu ucznia, a następnie użyć go do utworzenia obiektu szkoleniowego Trainer. Będziemy używać funkcji ucznia SGD i cross_entropy_with_softmax -
tr_loss = C.cross_entropy_with_softmax(nnet, Y)
tr_clas = C.classification_error(nnet, Y)
max_iter = 500
batch_size = 10
learn_rate = 0.01
learner = C.sgd(nnet.parameters, learn_rate)
trainer = C.Trainer(nnet, (tr_loss, tr_clas), [learner])Teraz, gdy skończyliśmy pracę z obiektem Trainer, musimy utworzyć funkcję czytnika, aby odczytać dane szkoleniowe -
rdr = create_reader(train_file, input_dim, output_dim, rnd_order=True, sweeps=C.io.INFINITELY_REPEAT)
banknote_input_map = { X : rdr.streams.x_src, Y : rdr.streams.y_src }Nadszedł czas, aby wyszkolić nasz model NN -
for i in range(0, max_iter):
curr_batch = rdr.next_minibatch(batch_size, input_map=iris_input_map) trainer.train_minibatch(curr_batch)
if i % 500 == 0:
mcee = trainer.previous_minibatch_loss_average
macc = (1.0 - trainer.previous_minibatch_evaluation_average) * 100
print("batch %4d: mean loss = %0.4f, accuracy = %0.2f%% " \ % (i, mcee, macc))Po zakończeniu uczenia oceńmy model przy użyciu elementów danych testowych -
print("\nEvaluating test data \n")
rdr = create_reader(test_file, input_dim, output_dim, rnd_order=False, sweeps=1)
banknote_input_map = { X : rdr.streams.x_src, Y : rdr.streams.y_src }
num_test = 20
all_test = rdr.next_minibatch(num_test, input_map=iris_input_map) acc = (1.0 - trainer.test_minibatch(all_test)) * 100
print("Classification accuracy = %0.2f%%" % acc)Po ocenie dokładności naszego wytrenowanego modelu NN będziemy go używać do prognozowania niewidocznych danych -
np.set_printoptions(precision = 1, suppress=True)
unknown = np.array([[0.6, 1.9, -3.3, -0.3]], dtype=np.float32)
print("\nPredicting Banknote authenticity for input features: ")
print(unknown[0])
pred_prob = model.eval(unknown)
np.set_printoptions(precision = 4, suppress=True)
print("Prediction probabilities are: ")
print(pred_prob[0])
if pred_prob[0,0] < pred_prob[0,1]:
print(“Prediction: authentic”)
else:
print(“Prediction: fake”)Kompletny model klasyfikacji dwuwęzłowej
def create_reader(path, input_dim, output_dim, rnd_order, sweeps):
x_strm = C.io.StreamDef(field='stats', shape=input_dim, is_sparse=False)
y_strm = C.io.StreamDef(field='forgery', shape=output_dim, is_sparse=False)
streams = C.io.StreamDefs(x_src=x_strm, y_src=y_strm)
deserial = C.io.CTFDeserializer(path, streams)
mb_src = C.io.MinibatchSource(deserial, randomize=rnd_order, max_sweeps=sweeps)
return mb_src
def main():
print("Using CNTK version = " + str(C.__version__) + "\n")
input_dim = 4
hidden_dim = 10
output_dim = 2
train_file = ".\\...\\" #provide the name of the training file
test_file = ".\\...\\" #provide the name of the test file
X = C.ops.input_variable(input_dim, np.float32)
Y = C.ops.input_variable(output_dim, np.float32)
withC.layers.default_options(init=C.initializer.uniform(scale=0.01, seed=1)):
hLayer = C.layers.Dense(hidden_dim, activation=C.ops.tanh, name='hidLayer')(X)
oLayer = C.layers.Dense(output_dim, activation=None, name='outLayer')(hLayer)
nnet = oLayer
model = C.ops.softmax(nnet)
tr_loss = C.cross_entropy_with_softmax(nnet, Y)
tr_clas = C.classification_error(nnet, Y)
max_iter = 500
batch_size = 10
learn_rate = 0.01
learner = C.sgd(nnet.parameters, learn_rate)
trainer = C.Trainer(nnet, (tr_loss, tr_clas), [learner])
rdr = create_reader(train_file, input_dim, output_dim, rnd_order=True, sweeps=C.io.INFINITELY_REPEAT)
banknote_input_map = { X : rdr.streams.x_src, Y : rdr.streams.y_src }
for i in range(0, max_iter):
curr_batch = rdr.next_minibatch(batch_size, input_map=iris_input_map) trainer.train_minibatch(curr_batch)
if i % 500 == 0:
mcee = trainer.previous_minibatch_loss_average
macc = (1.0 - trainer.previous_minibatch_evaluation_average) * 100
print("batch %4d: mean loss = %0.4f, accuracy = %0.2f%% " \ % (i, mcee, macc))
print("\nEvaluating test data \n")
rdr = create_reader(test_file, input_dim, output_dim, rnd_order=False, sweeps=1)
banknote_input_map = { X : rdr.streams.x_src, Y : rdr.streams.y_src }
num_test = 20
all_test = rdr.next_minibatch(num_test, input_map=iris_input_map) acc = (1.0 - trainer.test_minibatch(all_test)) * 100
print("Classification accuracy = %0.2f%%" % acc)
np.set_printoptions(precision = 1, suppress=True)
unknown = np.array([[0.6, 1.9, -3.3, -0.3]], dtype=np.float32)
print("\nPredicting Banknote authenticity for input features: ")
print(unknown[0])
pred_prob = model.eval(unknown)
np.set_printoptions(precision = 4, suppress=True)
print("Prediction probabilities are: ")
print(pred_prob[0])
if pred_prob[0,0] < pred_prob[0,1]:
print(“Prediction: authentic”)
else:
print(“Prediction: fake”)
if __name__== ”__main__”:
main()Wynik
Using CNTK version = 2.7
batch 0: mean loss = 0.6928, accuracy = 80.00%
batch 50: mean loss = 0.6877, accuracy = 70.00%
batch 100: mean loss = 0.6432, accuracy = 80.00%
batch 150: mean loss = 0.4978, accuracy = 80.00%
batch 200: mean loss = 0.4551, accuracy = 90.00%
batch 250: mean loss = 0.3755, accuracy = 90.00%
batch 300: mean loss = 0.2295, accuracy = 100.00%
batch 350: mean loss = 0.1542, accuracy = 100.00%
batch 400: mean loss = 0.1581, accuracy = 100.00%
batch 450: mean loss = 0.1499, accuracy = 100.00%
Evaluating test data
Classification accuracy = 84.58%
Predicting banknote authenticity for input features:
[0.6 1.9 -3.3 -0.3]
Prediction probabilities are:
[0.7847 0.2536]
Prediction: fakeModel klasyfikacji binarnej z jednym węzłem
Program implementacji jest prawie taki sam, jak powyżej dla klasyfikacji dwuwęzłowej. Główną zmianą jest to, że przy zastosowaniu techniki klasyfikacji dwuwęzłowej.
Możemy użyć wbudowanej funkcji CNTK Classification_error (), ale w przypadku klasyfikacji jednowęzłowej CNTK nie obsługuje funkcji Classification_error (). Dlatego musimy zaimplementować funkcję zdefiniowaną przez program w następujący sposób -
def class_acc(mb, x_var, y_var, model):
num_correct = 0; num_wrong = 0
x_mat = mb[x_var].asarray()
y_mat = mb[y_var].asarray()
for i in range(mb[x_var].shape[0]):
p = model.eval(x_mat[i]
y = y_mat[i]
if p[0,0] < 0.5 and y[0,0] == 0.0 or p[0,0] >= 0.5 and y[0,0] == 1.0:
num_correct += 1
else:
num_wrong += 1
return (num_correct * 100.0)/(num_correct + num_wrong)Dzięki tej zmianie zobaczmy pełny przykład klasyfikacji z jednym węzłem -
Kompletny jednowęzłowy model klasyfikacji
import numpy as np
import cntk as C
def create_reader(path, input_dim, output_dim, rnd_order, sweeps):
x_strm = C.io.StreamDef(field='stats', shape=input_dim, is_sparse=False)
y_strm = C.io.StreamDef(field='forgery', shape=output_dim, is_sparse=False)
streams = C.io.StreamDefs(x_src=x_strm, y_src=y_strm)
deserial = C.io.CTFDeserializer(path, streams)
mb_src = C.io.MinibatchSource(deserial, randomize=rnd_order, max_sweeps=sweeps)
return mb_src
def class_acc(mb, x_var, y_var, model):
num_correct = 0; num_wrong = 0
x_mat = mb[x_var].asarray()
y_mat = mb[y_var].asarray()
for i in range(mb[x_var].shape[0]):
p = model.eval(x_mat[i]
y = y_mat[i]
if p[0,0] < 0.5 and y[0,0] == 0.0 or p[0,0] >= 0.5 and y[0,0] == 1.0:
num_correct += 1
else:
num_wrong += 1
return (num_correct * 100.0)/(num_correct + num_wrong)
def main():
print("Using CNTK version = " + str(C.__version__) + "\n")
input_dim = 4
hidden_dim = 10
output_dim = 1
train_file = ".\\...\\" #provide the name of the training file
test_file = ".\\...\\" #provide the name of the test file
X = C.ops.input_variable(input_dim, np.float32)
Y = C.ops.input_variable(output_dim, np.float32)
with C.layers.default_options(init=C.initializer.uniform(scale=0.01, seed=1)):
hLayer = C.layers.Dense(hidden_dim, activation=C.ops.tanh, name='hidLayer')(X)
oLayer = C.layers.Dense(output_dim, activation=None, name='outLayer')(hLayer)
model = oLayer
tr_loss = C.cross_entropy_with_softmax(model, Y)
max_iter = 1000
batch_size = 10
learn_rate = 0.01
learner = C.sgd(model.parameters, learn_rate)
trainer = C.Trainer(model, (tr_loss), [learner])
rdr = create_reader(train_file, input_dim, output_dim, rnd_order=True, sweeps=C.io.INFINITELY_REPEAT)
banknote_input_map = {X : rdr.streams.x_src, Y : rdr.streams.y_src }
for i in range(0, max_iter):
curr_batch = rdr.next_minibatch(batch_size, input_map=iris_input_map) trainer.train_minibatch(curr_batch)
if i % 100 == 0:
mcee=trainer.previous_minibatch_loss_average
ca = class_acc(curr_batch, X,Y, model)
print("batch %4d: mean loss = %0.4f, accuracy = %0.2f%% " \ % (i, mcee, ca))
print("\nEvaluating test data \n")
rdr = create_reader(test_file, input_dim, output_dim, rnd_order=False, sweeps=1)
banknote_input_map = { X : rdr.streams.x_src, Y : rdr.streams.y_src }
num_test = 20
all_test = rdr.next_minibatch(num_test, input_map=iris_input_map)
acc = class_acc(all_test, X,Y, model)
print("Classification accuracy = %0.2f%%" % acc)
np.set_printoptions(precision = 1, suppress=True)
unknown = np.array([[0.6, 1.9, -3.3, -0.3]], dtype=np.float32)
print("\nPredicting Banknote authenticity for input features: ")
print(unknown[0])
pred_prob = model.eval({X:unknown})
print("Prediction probability: ")
print(“%0.4f” % pred_prob[0,0])
if pred_prob[0,0] < 0.5:
print(“Prediction: authentic”)
else:
print(“Prediction: fake”)
if __name__== ”__main__”:
main()Wynik
Using CNTK version = 2.7
batch 0: mean loss = 0.6936, accuracy = 10.00%
batch 100: mean loss = 0.6882, accuracy = 70.00%
batch 200: mean loss = 0.6597, accuracy = 50.00%
batch 300: mean loss = 0.5298, accuracy = 70.00%
batch 400: mean loss = 0.4090, accuracy = 100.00%
batch 500: mean loss = 0.3790, accuracy = 90.00%
batch 600: mean loss = 0.1852, accuracy = 100.00%
batch 700: mean loss = 0.1135, accuracy = 100.00%
batch 800: mean loss = 0.1285, accuracy = 100.00%
batch 900: mean loss = 0.1054, accuracy = 100.00%
Evaluating test data
Classification accuracy = 84.00%
Predicting banknote authenticity for input features:
[0.6 1.9 -3.3 -0.3]
Prediction probability:
0.8846
Prediction: fakeCNTK - regresja sieci neuronowej
Ten rozdział pomoże ci zrozumieć regresję sieci neuronowej w odniesieniu do CNTK.
Wprowadzenie
Jak wiemy, aby przewidzieć wartość liczbową na podstawie jednej lub więcej zmiennych predykcyjnych, używamy regresji. Weźmy przykład przewidywania mediany wartości domu powiedzmy w jednym ze 100 miast. Aby to zrobić, posiadamy dane, które obejmują:
Statystyka przestępczości dla każdego miasta.
Wiek domów w każdym mieście.
Miara odległości od każdego miasta do doskonałej lokalizacji.
Stosunek liczby uczniów do liczby nauczycieli w każdym mieście.
Rasowe statystyki demograficzne dla każdego miasta.
Średnia wartość domu w każdym mieście.
Na podstawie tych pięciu predyktorów chcielibyśmy przewidzieć medianę wartości domu. W tym celu możemy stworzyć model regresji liniowej wzdłuż linii -
Y = a0+a1(crime)+a2(house-age)+(a3)(distance)+(a4)(ratio)+(a5)(racial)W powyższym równaniu -
Y jest przewidywaną wartością mediany
a0 jest stałą i
a1 do a5 wszystkie są stałymi związanymi z pięcioma predyktorami omówionymi powyżej.
Mamy również alternatywne podejście do korzystania z sieci neuronowej. Stworzy dokładniejszy model prognozowania.
Tutaj utworzymy model regresji sieci neuronowej przy użyciu CNTK.
Ładowanie zbioru danych
Aby zaimplementować regresję sieci neuronowej za pomocą CNTK, użyjemy zestawu danych wartości domu w Bostonie. Zestaw danych można pobrać z repozytorium UCI Machine Learning Repository, które jest dostępne pod adresemhttps://archive.ics.uci.edu/ml/machine-learning-databases/housing/. Ten zbiór danych zawiera łącznie 14 zmiennych i 506 instancji.
Ale w naszym programie wdrożeniowym użyjemy sześciu z 14 zmiennych i 100 instancji. Na 6, 5 jako predyktory i jeden jako wartość do przewidzenia. Ze 100 instancji będziemy używać 80 do celów szkoleniowych i 20 do celów testowych. Wartość, którą chcemy przewidzieć, to mediana ceny domu w mieście. Zobaczmy pięć predyktorów, których będziemy używać -
Crime per capita in the town - Spodziewalibyśmy się, że z tym predyktorem zostaną skojarzone mniejsze wartości.
Proportion of owner - lokale zamieszkane zbudowane przed 1940 rokiem - Spodziewalibyśmy się, że z tym predyktorem będą powiązane mniejsze wartości, ponieważ większa wartość oznacza starszy dom.
Weighed distance of the town to five Boston employment centers.
Area school pupil-to-teacher ratio.
An indirect metric of the proportion of black residents in the town.
Przygotowywanie plików szkoleniowych i testowych
Tak jak wcześniej, najpierw musimy przekonwertować surowe dane do formatu CNTK. Zamierzamy użyć pierwszych 80 elementów danych do celów szkoleniowych, więc rozdzielany tabulatorami format CNTK jest następujący -
|predictors 1.612820 96.90 3.76 21.00 248.31 |medval 13.50
|predictors 0.064170 68.20 3.36 19.20 396.90 |medval 18.90
|predictors 0.097440 61.40 3.38 19.20 377.56 |medval 20.00
. . .Kolejnych 20 pozycji, również przekonwertowanych do formatu CNTK, posłuży do testów.
Konstruowanie modelu regresji
Najpierw musimy przetworzyć pliki danych w formacie CNTK iw tym celu użyjemy funkcji pomocniczej o nazwie create_reader w następujący sposób -
def create_reader(path, input_dim, output_dim, rnd_order, sweeps):
x_strm = C.io.StreamDef(field='predictors', shape=input_dim, is_sparse=False)
y_strm = C.io.StreamDef(field='medval', shape=output_dim, is_sparse=False)
streams = C.io.StreamDefs(x_src=x_strm, y_src=y_strm)
deserial = C.io.CTFDeserializer(path, streams)
mb_src = C.io.MinibatchSource(deserial, randomize=rnd_order, max_sweeps=sweeps)
return mb_srcNastępnie musimy utworzyć funkcję pomocniczą, która akceptuje obiekt mini-wsadowy CNTK i oblicza niestandardową metrykę dokładności.
def mb_accuracy(mb, x_var, y_var, model, delta):
num_correct = 0
num_wrong = 0
x_mat = mb[x_var].asarray()
y_mat = mb[y_var].asarray()
for i in range(mb[x_var].shape[0]):
v = model.eval(x_mat[i])
y = y_mat[i]
if np.abs(v[0,0] – y[0,0]) < delta:
num_correct += 1
else:
num_wrong += 1
return (num_correct * 100.0)/(num_correct + num_wrong)Teraz musimy ustawić argumenty architektury dla naszego NN, a także podać lokalizację plików danych. Można to zrobić za pomocą następującego kodu Pythona -
def main():
print("Using CNTK version = " + str(C.__version__) + "\n")
input_dim = 5
hidden_dim = 20
output_dim = 1
train_file = ".\\...\\" #provide the name of the training file(80 data items)
test_file = ".\\...\\" #provide the name of the test file(20 data items)Teraz przy pomocy następującego wiersza kodu nasz program utworzy nieprzeszkolony NN -
X = C.ops.input_variable(input_dim, np.float32)
Y = C.ops.input_variable(output_dim, np.float32)
with C.layers.default_options(init=C.initializer.uniform(scale=0.01, seed=1)):
hLayer = C.layers.Dense(hidden_dim, activation=C.ops.tanh, name='hidLayer')(X)
oLayer = C.layers.Dense(output_dim, activation=None, name='outLayer')(hLayer)
model = C.ops.alias(oLayer)Teraz, kiedy już stworzyliśmy podwójny nietrenowany model, musimy skonfigurować obiekt algorytmu ucznia. Będziemy używać uczącego się SGD isquared_error funkcja straty -
tr_loss = C.squared_error(model, Y)
max_iter = 3000
batch_size = 5
base_learn_rate = 0.02
sch=C.learning_parameter_schedule([base_learn_rate, base_learn_rate/2], minibatch_size=batch_size, epoch_size=int((max_iter*batch_size)/2))
learner = C.sgd(model.parameters, sch)
trainer = C.Trainer(model, (tr_loss), [learner])Teraz, gdy skończymy z obiektem algorytmu uczenia się, musimy utworzyć funkcję czytnika, aby odczytać dane szkoleniowe -
rdr = create_reader(train_file, input_dim, output_dim, rnd_order=True, sweeps=C.io.INFINITELY_REPEAT)
boston_input_map = { X : rdr.streams.x_src, Y : rdr.streams.y_src }Nadszedł czas, aby wyszkolić nasz model NN -
for i in range(0, max_iter):
curr_batch = rdr.next_minibatch(batch_size, input_map=boston_input_map) trainer.train_minibatch(curr_batch)
if i % int(max_iter/10) == 0:
mcee = trainer.previous_minibatch_loss_average
acc = mb_accuracy(curr_batch, X, Y, model, delta=3.00)
print("batch %4d: mean squared error = %8.4f, accuracy = %5.2f%% " \ % (i, mcee, acc))Po zakończeniu uczenia oceńmy model przy użyciu elementów danych testowych -
print("\nEvaluating test data \n")
rdr = create_reader(test_file, input_dim, output_dim, rnd_order=False, sweeps=1)
boston_input_map = { X : rdr.streams.x_src, Y : rdr.streams.y_src }
num_test = 20
all_test = rdr.next_minibatch(num_test, input_map=boston_input_map)
acc = mb_accuracy(all_test, X, Y, model, delta=3.00)
print("Prediction accuracy = %0.2f%%" % acc)Po ocenie dokładności naszego wytrenowanego modelu NN będziemy go używać do prognozowania niewidocznych danych -
np.set_printoptions(precision = 2, suppress=True)
unknown = np.array([[0.09, 50.00, 4.5, 17.00, 350.00], dtype=np.float32)
print("\nPredicting median home value for feature/predictor values: ")
print(unknown[0])
pred_prob = model.eval({X: unknown)
print("\nPredicted value is: ")
print(“$%0.2f (x1000)” %pred_value[0,0])Kompletny model regresji
import numpy as np
import cntk as C
def create_reader(path, input_dim, output_dim, rnd_order, sweeps):
x_strm = C.io.StreamDef(field='predictors', shape=input_dim, is_sparse=False)
y_strm = C.io.StreamDef(field='medval', shape=output_dim, is_sparse=False)
streams = C.io.StreamDefs(x_src=x_strm, y_src=y_strm)
deserial = C.io.CTFDeserializer(path, streams)
mb_src = C.io.MinibatchSource(deserial, randomize=rnd_order, max_sweeps=sweeps)
return mb_src
def mb_accuracy(mb, x_var, y_var, model, delta):
num_correct = 0
num_wrong = 0
x_mat = mb[x_var].asarray()
y_mat = mb[y_var].asarray()
for i in range(mb[x_var].shape[0]):
v = model.eval(x_mat[i])
y = y_mat[i]
if np.abs(v[0,0] – y[0,0]) < delta:
num_correct += 1
else:
num_wrong += 1
return (num_correct * 100.0)/(num_correct + num_wrong)
def main():
print("Using CNTK version = " + str(C.__version__) + "\n")
input_dim = 5
hidden_dim = 20
output_dim = 1
train_file = ".\\...\\" #provide the name of the training file(80 data items)
test_file = ".\\...\\" #provide the name of the test file(20 data items)
X = C.ops.input_variable(input_dim, np.float32)
Y = C.ops.input_variable(output_dim, np.float32)
with C.layers.default_options(init=C.initializer.uniform(scale=0.01, seed=1)):
hLayer = C.layers.Dense(hidden_dim, activation=C.ops.tanh, name='hidLayer')(X)
oLayer = C.layers.Dense(output_dim, activation=None, name='outLayer')(hLayer)
model = C.ops.alias(oLayer)
tr_loss = C.squared_error(model, Y)
max_iter = 3000
batch_size = 5
base_learn_rate = 0.02
sch = C.learning_parameter_schedule([base_learn_rate, base_learn_rate/2], minibatch_size=batch_size, epoch_size=int((max_iter*batch_size)/2))
learner = C.sgd(model.parameters, sch)
trainer = C.Trainer(model, (tr_loss), [learner])
rdr = create_reader(train_file, input_dim, output_dim, rnd_order=True, sweeps=C.io.INFINITELY_REPEAT)
boston_input_map = { X : rdr.streams.x_src, Y : rdr.streams.y_src }
for i in range(0, max_iter):
curr_batch = rdr.next_minibatch(batch_size, input_map=boston_input_map) trainer.train_minibatch(curr_batch)
if i % int(max_iter/10) == 0:
mcee = trainer.previous_minibatch_loss_average
acc = mb_accuracy(curr_batch, X, Y, model, delta=3.00)
print("batch %4d: mean squared error = %8.4f, accuracy = %5.2f%% " \ % (i, mcee, acc))
print("\nEvaluating test data \n")
rdr = create_reader(test_file, input_dim, output_dim, rnd_order=False, sweeps=1)
boston_input_map = { X : rdr.streams.x_src, Y : rdr.streams.y_src }
num_test = 20
all_test = rdr.next_minibatch(num_test, input_map=boston_input_map)
acc = mb_accuracy(all_test, X, Y, model, delta=3.00)
print("Prediction accuracy = %0.2f%%" % acc)
np.set_printoptions(precision = 2, suppress=True)
unknown = np.array([[0.09, 50.00, 4.5, 17.00, 350.00], dtype=np.float32)
print("\nPredicting median home value for feature/predictor values: ")
print(unknown[0])
pred_prob = model.eval({X: unknown)
print("\nPredicted value is: ")
print(“$%0.2f (x1000)” %pred_value[0,0])
if __name__== ”__main__”:
main()Wynik
Using CNTK version = 2.7
batch 0: mean squared error = 385.6727, accuracy = 0.00%
batch 300: mean squared error = 41.6229, accuracy = 20.00%
batch 600: mean squared error = 28.7667, accuracy = 40.00%
batch 900: mean squared error = 48.6435, accuracy = 40.00%
batch 1200: mean squared error = 77.9562, accuracy = 80.00%
batch 1500: mean squared error = 7.8342, accuracy = 60.00%
batch 1800: mean squared error = 47.7062, accuracy = 60.00%
batch 2100: mean squared error = 40.5068, accuracy = 40.00%
batch 2400: mean squared error = 46.5023, accuracy = 40.00%
batch 2700: mean squared error = 15.6235, accuracy = 60.00%
Evaluating test data
Prediction accuracy = 64.00%
Predicting median home value for feature/predictor values:
[0.09 50. 4.5 17. 350.]
Predicted value is:
$21.02(x1000)Zapisywanie wytrenowanego modelu
Ten zbiór danych dotyczących wartości Boston Home zawiera tylko 506 pozycji danych (spośród których pozyliśmy tylko 100). W związku z tym wytrenowanie modelu regresora NN zajęłoby tylko kilka sekund, ale uczenie na dużym zestawie danych zawierającym sto lub tysiące elementów danych może zająć godziny lub nawet dni.
Możemy zapisać nasz model, dzięki czemu nie będziemy musieli zachowywać go od nowa. Z pomocą śledzenia kodu Pythona możemy zapisać nasz wyszkolony NN -
nn_regressor = “.\\neuralregressor.model” #provide the name of the file
model.save(nn_regressor, format=C.ModelFormat.CNTKv2)Poniżej przedstawiono argumenty funkcji save () użytej powyżej -
Nazwa pliku jest pierwszym argumentem save()funkcjonować. Można go również zapisać wraz ze ścieżką do pliku.
Another parameter is the format parameter which has a default value C.ModelFormat.CNTKv2.
Loading the trained model
Once you saved the trained model, it’s very easy to load that model. We only need to use the load () function. Let’s check this in following example −
import numpy as np
import cntk as C
model = C.ops.functions.Function.load(“.\\neuralregressor.model”)
np.set_printoptions(precision = 2, suppress=True)
unknown = np.array([[0.09, 50.00, 4.5, 17.00, 350.00], dtype=np.float32)
print("\nPredicting area median home value for feature/predictor values: ")
print(unknown[0])
pred_prob = model.eval({X: unknown)
print("\nPredicted value is: ")
print(“$%0.2f (x1000)” %pred_value[0,0])The benefit of saved model is that once you load a saved model, it can be used exactly as if the model had just been trained.
CNTK - Classification Model
This chapter will help you to understand how to measure performance of classification model in CNTK. Let us begin with confusion matrix.
Confusion matrix
Confusion matrix - a table with the predicted output versus the expected output is the easiest way to measure the performance of a classification problem, where the output can be of two or more type of classes.
In order to understand how it works, we are going to create a confusion matrix for a binary classification model that predicts, whether a credit card transaction was normal or a fraud. It is shown as follows −
| Actual fraud | Actual normal | |
|---|---|---|
| Predicted fraud |
True positive |
False positive |
| Predicted normal |
False negative |
True negative |
As we can see, the above sample confusion matrix contains 2 columns, one for class fraud and other for class normal. In the same way we have 2 rows, one is added for class fraud and other is added for class normal. Following is the explanation of the terms associated with confusion matrix −
True Positives − When both actual class & predicted class of data point is 1.
True Negatives − When both actual class & predicted class of data point is 0.
False Positives − When actual class of data point is 0 & predicted class of data point is 1.
False Negatives − When actual class of data point is 1 & predicted class of data point is 0.
Let’s see, how we can calculate number of different things from the confusion matrix −
Accuracy − It is the number of correct predictions made by our ML classification model. It can be calculated with the help of following formula −
Precision −It tells us how many samples were correctly predicted out of all samples we predicted. It can be calculated with the help of following formula −
Recall or Sensitivity − Recall are the number of positives returned by our ML classification model. In other words, it tells us how many of the fraud cases in the dataset were actually detected by the model. It can be calculated with the help of following formula −
Specificity − Opposite to recall, it gives the number of negatives returned by our ML classification model. It can be calculated with the help of following formula −




F-measure
We can use F-measure as an alternative of Confusion matrix. The main reason behind this, we can’t maximize Recall and Precision at the same time. There is a very strong relationship between these metrics and that can be understood with the help of following example −
Suppose, we want to use a DL model to classify cell samples as cancerous or normal. Here, to reach maximum precision we need to reduce the number of predictions to 1. Although, this can give us reach around 100 percent precision, but recall will become really low.
On the other hand, if we would like to reach maximum recall, we need to make as many predictions as possible. Although, this can give us reach around 100 percent recall, but precision will become really low.
In practice, we need to find a way balancing between precision and recall. The F-measure metric allows us to do so, as it expresses a harmonic average between precision and recall.

This formula is called the F1-measure, where the extra term called B is set to 1 to get an equal ratio of precision and recall. In order to emphasize recall, we can set the factor B to 2. On the other hand, to emphasize precision, we can set the factor B to 0.5.
Using CNTK to measure classification performance
In previous section we have created a classification model using Iris flower dataset. Here, we will be measuring its performance by using confusion matrix and F-measure metric.
Creating Confusion matrix
We already created the model, so we can start the validating process, which includes confusion matrix, on the same. First, we are going to create confusion matrix with the help of the confusion_matrix function from scikit-learn. For this, we need the real labels for our test samples and the predicted labels for the same test samples.
Let’s calculate the confusion matrix by using following python code −
from sklearn.metrics import confusion_matrix
y_true = np.argmax(y_test, axis=1)
y_pred = np.argmax(z(X_test), axis=1)
matrix = confusion_matrix(y_true=y_true, y_pred=y_pred)
print(matrix)Output
[[10 0 0]
[ 0 1 9]
[ 0 0 10]]We can also use heatmap function to visualise a confusion matrix as follows −
import seaborn as sns
import matplotlib.pyplot as plt
g = sns.heatmap(matrix,
annot=True,
xticklabels=label_encoder.classes_.tolist(),
yticklabels=label_encoder.classes_.tolist(),
cmap='Blues')
g.set_yticklabels(g.get_yticklabels(), rotation=0)
plt.show()
We should also have a single performance number, that we can use to compare the model. For this, we need to calculate the classification error by using classification_error function, from the metrics package in CNTK as done while creating classification model.
Now to calculate the classification error, execute the test method on the loss function with a dataset. After that, CNTK will take the samples we provided as input for this function and make a prediction based on input features X_test.
loss.test([X_test, y_test])Output
{'metric': 0.36666666666, 'samples': 30}Implementing F-Measures
For implementing F-Measures, CNTK also includes function called fmeasures. We can use this function, while training the NN by replacing the cell cntk.metrics.classification_error, with a call to cntk.losses.fmeasure when defining the criterion factory function as follows −
import cntk
@cntk.Function
def criterion_factory(output, target):
loss = cntk.losses.cross_entropy_with_softmax(output, target)
metric = cntk.losses.fmeasure(output, target)
return loss, metricAfter using cntk.losses.fmeasure function, we will get different output for the loss.test method call given as follows −
loss.test([X_test, y_test])Output
{'metric': 0.83101488749, 'samples': 30}CNTK - Regression Model
Here, we will study about measuring performance with regards to a regression model.
Basics of validating a regression model
As we know that regression models are different than classification models, in the sense that, there is no binary measure of right or wrong for individuals’ samples. In regression models, we want to measure how close the prediction is to the actual value. The closer the prediction value is to the expected output, the better the model performs.
Here, we are going to measure the performance of NN used for regression using different error-rate functions.
Calculating error margin
As discussed earlier, while validating a regression model, we can’t say whether a prediction is right or wrong. We want our prediction to be as close as possible to the real value. But, a small error margin is acceptable here.
The formula for calculating the error margin is as follows −

Here,
Predicted value = indicated y by a hat
Real value = predicted by y
First, we need to calculate the distance between the predicted and the real value. Then, to get an overall error rate, we need to sum these squared distances and calculate the average. This is called the mean squared error function.
But, if we want performance figures that express an error margin, we need a formula that expresses the absolute error. The formula for mean absolute error function is as follows −

The above formula takes the absolute distance between the predicted and the real value.
Using CNTK to measure regression performance
Here, we will look at how to use the different metrics, we discussed in combination with CNTK. We will use a regression model, that predicts miles per gallon for cars using the steps given below.
Implementation steps−
Step 1 − First, we need to import the required components from cntk package as follows −
from cntk import default_option, input_variable
from cntk.layers import Dense, Sequential
from cntk.ops import reluStep 2 − Next, we need to define a default activation function using the default_options functions. Then, create a new Sequential layer set and provide two Dense layers with 64 neurons each. Then, we add an additional Dense layer (which will act as the output layer) to the Sequential layer set and give 1 neuron without an activation as follows −
with default_options(activation=relu):
model = Sequential([Dense(64),Dense(64),Dense(1,activation=None)])Step 3 − Once the network has been created, we need to create an input feature. We need to make sure that, it has the same shape as the features that we are going to be using for training.
features = input_variable(X.shape[1])Step 4 − Now, we need to create another input_variable with size 1. It will be used to store the expected value for NN.
target = input_variable(1)
z = model(features)Now, we need to train the model and in order to do so, we are going to split the dataset and perform preprocessing using the following implementation steps −
Step 5 −First, import StandardScaler from sklearn.preprocessing to get the values between -1 and +1. This will help us against exploding gradient problems in the NN.
from sklearn.preprocessing import StandardScalarStep 6 − Next, import train_test_split from sklearn.model_selection as follows−
from sklearn.model_selection import train_test_splitStep 7 − Drop the mpg column from the dataset by using the dropmethod. At last split the dataset into a training and validation set using the train_test_split function as follows −
x = df_cars.drop(columns=[‘mpg’]).values.astype(np.float32)
y=df_cars.iloc[: , 0].values.reshape(-1, 1).astype(np.float32)
scaler = StandardScaler()
X = scaler.fit_transform(x)
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2)Step 8 − Now, we need to create another input_variable with size 1. It will be used to store the expected value for NN.
target = input_variable(1)
z = model(features)We have split as well as preprocessed the data, now we need to train the NN. As did in previous sections while creating regression model, we need to define a combination of a loss and metric function to train the model.
import cntk
def absolute_error(output, target):
return cntk.ops.reduce_mean(cntk.ops.abs(output – target))
@ cntk.Function
def criterion_factory(output, target):
loss = squared_error(output, target)
metric = absolute_error(output, target)
return loss, metricNow, let’s have a look at how to use the trained model. For our model, we will use criterion_factory as the loss and metric combination.
from cntk.losses import squared_error
from cntk.learners import sgd
from cntk.logging import ProgressPrinter
progress_printer = ProgressPrinter(0)
loss = criterion_factory (z, target)
learner = sgd(z.parameters, 0.001)
training_summary=loss.train((x_train,y_train),parameter_learners=[learner],callbacks=[progress_printer],minibatch_size=16,max_epochs=10)Complete implementation example
from cntk import default_option, input_variable
from cntk.layers import Dense, Sequential
from cntk.ops import relu
with default_options(activation=relu):
model = Sequential([Dense(64),Dense(64),Dense(1,activation=None)])
features = input_variable(X.shape[1])
target = input_variable(1)
z = model(features)
from sklearn.preprocessing import StandardScalar
from sklearn.model_selection import train_test_split
x = df_cars.drop(columns=[‘mpg’]).values.astype(np.float32)
y=df_cars.iloc[: , 0].values.reshape(-1, 1).astype(np.float32)
scaler = StandardScaler()
X = scaler.fit_transform(x)
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2)
target = input_variable(1)
z = model(features)
import cntk
def absolute_error(output, target):
return cntk.ops.reduce_mean(cntk.ops.abs(output – target))
@ cntk.Function
def criterion_factory(output, target):
loss = squared_error(output, target)
metric = absolute_error(output, target)
return loss, metric
from cntk.losses import squared_error
from cntk.learners import sgd
from cntk.logging import ProgressPrinter
progress_printer = ProgressPrinter(0)
loss = criterion_factory (z, target)
learner = sgd(z.parameters, 0.001)
training_summary=loss.train((x_train,y_train),parameter_learners=[learner],callbacks=[progress_printer],minibatch_size=16,max_epochs=10)Output
-------------------------------------------------------------------
average since average since examples
loss last metric last
------------------------------------------------------
Learning rate per minibatch: 0.001
690 690 24.9 24.9 16
654 636 24.1 23.7 48
[………]In order to validate our regression model, we need to make sure that, the model handles new data just as well as it does with the training data. For this, we need to invoke the test method on loss and metric combination with test data as follows −
loss.test([X_test, y_test])Output−
{'metric': 1.89679785619, 'samples': 79}CNTK - Out-of-Memory Datasets
In this chapter, how to measure performance of out-of-memory datasets will be explained.
In previous sections, we have discussed about various methods to validate the performance of our NN, but the methods we have discussed, are ones that deals with the datasets that fit in the memory.
Here, the question arises what about out-of-memory datasets, because in production scenario, we need a lot of data to train NN. In this section, we are going to discuss how to measure performance when working with minibatch sources and manual minibatch loop.
Minibatch sources
While working with out-of-memory dataset, i.e. minibatch sources, we need slightly different setup for loss, as well as metric, than the setup we used while working with small datasets i.e. in-memory datasets. First, we will see how to set up a way to feed data to the trainer of NN model.
Following are the implementation steps−
Step 1 − First, from cntk.io module import the components for creating the minibatch source as follows−
from cntk.io import StreamDef, StreamDefs, MinibatchSource, CTFDeserializer,
INFINITY_REPEATStep 2 − Next, create a new function named say create_datasource. This function will have two parameters namely filename and limit, with a default value of INFINITELY_REPEAT.
def create_datasource(filename, limit =INFINITELY_REPEAT)Step 3 − Now, within the function, by using StreamDef class crate a stream definition for the labels that reads from the labels field that has three features. We also need to set is_sparse to False as follows−
labels_stream = StreamDef(field=’labels’, shape=3, is_sparse=False)Step 4 − Next, create to read the features filed from the input file, create another instance of StreamDef as follows.
feature_stream = StreamDef(field=’features’, shape=4, is_sparse=False)Step 5 − Now, initialise the CTFDeserializer instance class. Specify the filename and streams that we need to deserialize as follows −
deserializer = CTFDeserializer(filename, StreamDefs(labels=
label_stream, features=features_stream)Step 6 − Next, we need to create instance of minisourceBatch by using deserializer as follows −
Minibatch_source = MinibatchSource(deserializer, randomize=True, max_sweeps=limit)
return minibatch_sourceStep 7 − At last, we need to provide training and testing source, which we created in previous sections also. We are using iris flower dataset.
training_source = create_datasource(‘Iris_train.ctf’)
test_source = create_datasource(‘Iris_test.ctf’, limit=1)Once you create MinibatchSource instance, we need to train it. We can use the same training logic, as used when we worked with small in-memory datasets. Here, we will use MinibatchSource instance, as the input for the train method on loss function as follows −
Following are the implementation steps−
Step 1 − In order to log the output of the training session, first import the ProgressPrinter from cntk.logging module as follows −
from cntk.logging import ProgressPrinterStep 2 − Next, to set up the training session, import the trainer and training_session from cntk.train module as follows−
from cntk.train import Trainer, training_sessionStep 3 − Now, we need to define some set of constants like minibatch_size, samples_per_epoch and num_epochs as follows−
minbatch_size = 16
samples_per_epoch = 150
num_epochs = 30
max_samples = samples_per_epoch * num_epochsStep 4 − Next, in order to know how to read data during training in CNTK, we need to define a mapping between the input variable for the network and the streams in the minibatch source.
input_map = {
features: training_source.streams.features,
labels: training_source.streams.labels
}Step 5 − Next to log the output of the training process, initialize the progress_printer variable with a new ProgressPrinter instance. Also, initialize the trainer and provide it with the model as follows−
progress_writer = ProgressPrinter(0)
trainer: training_source.streams.labelsStep 6 − At last, to start the training process, we need to invoke the training_session function as follows −
session = training_session(trainer,
mb_source=training_source,
mb_size=minibatch_size,
model_inputs_to_streams=input_map,
max_samples=max_samples,
test_config=test_config)
session.train()Once we trained the model, we can add validation to this setup by using a TestConfig object and assign it to the test_config keyword argument of the train_session function.
Following are the implementation steps−
Step 1 − First, we need to import the TestConfig class from the module cntk.train as follows−
from cntk.train import TestConfigStep 2 − Now, we need to create a new instance of the TestConfig with the test_source as input−
Test_config = TestConfig(test_source)Complete Example
from cntk.io import StreamDef, StreamDefs, MinibatchSource, CTFDeserializer, INFINITY_REPEAT
def create_datasource(filename, limit =INFINITELY_REPEAT)
labels_stream = StreamDef(field=’labels’, shape=3, is_sparse=False)
feature_stream = StreamDef(field=’features’, shape=4, is_sparse=False)
deserializer = CTFDeserializer(filename, StreamDefs(labels=label_stream, features=features_stream)
Minibatch_source = MinibatchSource(deserializer, randomize=True, max_sweeps=limit)
return minibatch_source
training_source = create_datasource(‘Iris_train.ctf’)
test_source = create_datasource(‘Iris_test.ctf’, limit=1)
from cntk.logging import ProgressPrinter
from cntk.train import Trainer, training_session
minbatch_size = 16
samples_per_epoch = 150
num_epochs = 30
max_samples = samples_per_epoch * num_epochs
input_map = {
features: training_source.streams.features,
labels: training_source.streams.labels
}
progress_writer = ProgressPrinter(0)
trainer: training_source.streams.labels
session = training_session(trainer,
mb_source=training_source,
mb_size=minibatch_size,
model_inputs_to_streams=input_map,
max_samples=max_samples,
test_config=test_config)
session.train()
from cntk.train import TestConfig
Test_config = TestConfig(test_source)Output
-------------------------------------------------------------------
average since average since examples
loss last metric last
------------------------------------------------------
Learning rate per minibatch: 0.1
1.57 1.57 0.214 0.214 16
1.38 1.28 0.264 0.289 48
[………]
Finished Evaluation [1]: Minibatch[1-1]:metric = 69.65*30;Manual minibatch loop
As we see above, it is easy to measure the performance of our NN model during and after training, by using the metrics when training with regular APIs in CNTK. But, on the other side, things will not be that easy while working with a manual minibatch loop.
Here, we are using the model given below with 4 inputs and 3 outputs from Iris Flower dataset, created in previous sections too−
from cntk import default_options, input_variable
from cntk.layers import Dense, Sequential
from cntk.ops import log_softmax, relu, sigmoid
from cntk.learners import sgd
model = Sequential([
Dense(4, activation=sigmoid),
Dense(3, activation=log_softmax)
])
features = input_variable(4)
labels = input_variable(3)
z = model(features)Next, the loss for the model is defined as the combination of the cross-entropy loss function, and the F-measure metric as used in previous sections. We are going to use the criterion_factory utility, to create this as a CNTK function object as shown below−
import cntk
from cntk.losses import cross_entropy_with_softmax, fmeasure
@cntk.Function
def criterion_factory(outputs, targets):
loss = cross_entropy_with_softmax(outputs, targets)
metric = fmeasure(outputs, targets, beta=1)
return loss, metric
loss = criterion_factory(z, labels)
learner = sgd(z.parameters, 0.1)
label_mapping = {
'Iris-setosa': 0,
'Iris-versicolor': 1,
'Iris-virginica': 2
}Now, as we have defined the loss function, we will see how we can use it in the trainer, to set up a manual training session.
Following are the implementation steps −
Step 1 − First, we need to import the required packages like numpy and pandas to load and preprocess the data.
import pandas as pd
import numpy as npStep 2 − Next, in order to log information during training, import the ProgressPrinter class as follows−
from cntk.logging import ProgressPrinterStep 3 − Then, we need to import the trainer module from cntk.train module as follows −
from cntk.train import TrainerStep 4 − Next, create a new instance of ProgressPrinter as follows −
progress_writer = ProgressPrinter(0)Step 5 − Now, we need to initialise trainer with the parameters the loss, the learner and the progress_writer as follows −
trainer = Trainer(z, loss, learner, progress_writer)Step 6 −Next, in order to train the model, we will create a loop that will iterate over the dataset thirty times. This will be the outer training loop.
for _ in range(0,30):Step 7 − Now, we need to load the data from disk using pandas. Then, in order to load the dataset in mini-batches, set the chunksize keyword argument to 16.
input_data = pd.read_csv('iris.csv',
names=['sepal_length', 'sepal_width','petal_length','petal_width', 'species'],
index_col=False, chunksize=16)Step 8 − Now, create an inner training for loop to iterate over each of the mini-batches.
for df_batch in input_data:Step 9 − Now inside this loop, read the first four columns using the iloc indexer, as the features to train from and convert them to float32 −
feature_values = df_batch.iloc[:,:4].values
feature_values = feature_values.astype(np.float32)Step 10 − Now, read the last column as the labels to train from, as follows −
label_values = df_batch.iloc[:,-1]Step 11 − Next, we will use one-hot vectors to convert the label strings to their numeric presentation as follows −
label_values = label_values.map(lambda x: label_mapping[x])Step 12 − After that, take the numeric presentation of the labels. Next, convert them to a numpy array, so it is easier to work with them as follows −
label_values = label_values.valuesStep 13 − Now, we need to create a new numpy array that has the same number of rows as the label values that we have converted.
encoded_labels = np.zeros((label_values.shape[0], 3))Step 14 − Now, in order to create one-hot encoded labels, select the columns based on the numeric label values.
encoded_labels[np.arange(label_values.shape[0]), label_values] = 1.Step 15 − At last, we need to invoke the train_minibatch method on the trainer and provide the processed features and labels for the minibatch.
trainer.train_minibatch({features: feature_values, labels: encoded_labels})Complete Example
from cntk import default_options, input_variable
from cntk.layers import Dense, Sequential
from cntk.ops import log_softmax, relu, sigmoid
from cntk.learners import sgd
model = Sequential([
Dense(4, activation=sigmoid),
Dense(3, activation=log_softmax)
])
features = input_variable(4)
labels = input_variable(3)
z = model(features)
import cntk
from cntk.losses import cross_entropy_with_softmax, fmeasure
@cntk.Function
def criterion_factory(outputs, targets):
loss = cross_entropy_with_softmax(outputs, targets)
metric = fmeasure(outputs, targets, beta=1)
return loss, metric
loss = criterion_factory(z, labels)
learner = sgd(z.parameters, 0.1)
label_mapping = {
'Iris-setosa': 0,
'Iris-versicolor': 1,
'Iris-virginica': 2
}
import pandas as pd
import numpy as np
from cntk.logging import ProgressPrinter
from cntk.train import Trainer
progress_writer = ProgressPrinter(0)
trainer = Trainer(z, loss, learner, progress_writer)
for _ in range(0,30):
input_data = pd.read_csv('iris.csv',
names=['sepal_length', 'sepal_width','petal_length','petal_width', 'species'],
index_col=False, chunksize=16)
for df_batch in input_data:
feature_values = df_batch.iloc[:,:4].values
feature_values = feature_values.astype(np.float32)
label_values = df_batch.iloc[:,-1]
label_values = label_values.map(lambda x: label_mapping[x])
label_values = label_values.values
encoded_labels = np.zeros((label_values.shape[0], 3))
encoded_labels[np.arange(label_values.shape[0]),
label_values] = 1.
trainer.train_minibatch({features: feature_values, labels: encoded_labels})Output
-------------------------------------------------------------------
average since average since examples
loss last metric last
------------------------------------------------------
Learning rate per minibatch: 0.1
1.45 1.45 -0.189 -0.189 16
1.24 1.13 -0.0382 0.0371 48
[………]In the above output, we got both the output for the loss and the metric during training. It is because we combined a metric and loss in a function object and used a progress printer in the trainer configuration.
Now, in order to evaluate the model performance, we need to perform same task as with training the model, but this time, we need to use an Evaluator instance to test the model. It is shown in the following Python code−
from cntk import Evaluator
evaluator = Evaluator(loss.outputs[1], [progress_writer])
input_data = pd.read_csv('iris.csv',
names=['sepal_length', 'sepal_width','petal_length','petal_width', 'species'],
index_col=False, chunksize=16)
for df_batch in input_data:
feature_values = df_batch.iloc[:,:4].values
feature_values = feature_values.astype(np.float32)
label_values = df_batch.iloc[:,-1]
label_values = label_values.map(lambda x: label_mapping[x])
label_values = label_values.values
encoded_labels = np.zeros((label_values.shape[0], 3))
encoded_labels[np.arange(label_values.shape[0]), label_values] = 1.
evaluator.test_minibatch({ features: feature_values, labels:
encoded_labels})
evaluator.summarize_test_progress()Now, we will get the output something like the following−
Output
Finished Evaluation [1]: Minibatch[1-11]:metric = 74.62*143;CNTK - Monitoring the Model
In this chapter, we will understand how to monitor a model in CNTK.
Introduction
In previous sections, we have done some validation on our NN models. But, is it also necessary and possible to monitor our model during training?
Yes, already we have used ProgressWriter class to monitor our model and there are many more ways to do so. Before getting deep into the ways, first let’s have a look how monitoring in CNTK works and how we can use it to detect problems in our NN model.
Callbacks in CNTK
Actually, during training and validation, CNTK allows us to specify callbacks in several spots in the API. First, let’s take a closer look at when CNTK invokes callbacks.
When CNTK invoke callbacks?
CNTK will invoke the callbacks at the training and testing set moments when−
A minibatch is completed.
A full sweep over the dataset is completed during training.
A minibatch of testing is completed.
A full sweep over the dataset is completed during testing.
Specifying callbacks
While working with CNTK, we can specify callbacks in several spots in the API. For example−
When call train on a loss function?
Here, when we call train on a loss function, we can specify a set of callbacks through the callbacks argument as follows−
training_summary=loss.train((x_train,y_train),
parameter_learners=[learner],
callbacks=[progress_writer]),
minibatch_size=16, max_epochs=15)When working with minibatch sources or using a manual minibatch loop−
In this case, we can specify callbacks for monitoring purpose while creating the Trainer as follows−
from cntk.logging import ProgressPrinter
callbacks = [
ProgressPrinter(0)
]
Trainer = Trainer(z, (loss, metric), learner, [callbacks])Various monitoring tools
Let us study about different monitoring tools.
ProgressPrinter
While reading this tutorial, you will find ProgressPrinter as the most used monitoring tool. Some of the characteristics of ProgressPrinter monitoring tool are−
ProgressPrinter class implements basic console-based logging to monitor our model. It can log to disk we want it to.
Especially useful while working in a distributed training scenario.
It is also very useful while working in a scenario where we can’t log in on the console to see the output of our Python program.
With the help of following code, we can create an instance of ProgressPrinter−
ProgressPrinter(0, log_to_file=’test.txt’)We will get the output something that we have seen in the earlier sections−
Test.txt
CNTKCommandTrainInfo: train : 300
CNTKCommandTrainInfo: CNTKNoMoreCommands_Total : 300
CNTKCommandTrainBegin: train
-------------------------------------------------------------------
average since average since examples
loss last metric last
------------------------------------------------------
Learning rate per minibatch: 0.1
1.45 1.45 -0.189 -0.189 16
1.24 1.13 -0.0382 0.0371 48
[………]TensorBoard
One of the disadvantages of using ProgressPrinter is that, we can’t get a good view of how the loss and metric progress over time is hard. TensorBoardProgressWriter is a great alternative to the ProgressPrinter class in CNTK.
Before using it, we need to first install it with the help of following command −
pip install tensorboardNow, in order to use TensorBoard, we need to set up TensorBoardProgressWriter in our training code as follows−
import time
from cntk.logging import TensorBoardProgressWriter
tensorbrd_writer = TensorBoardProgressWriter(log_dir=’logs/{}’.format(time.time()),freq=1,model=z)It is a good practice to call the close method on TensorBoardProgressWriter instance after done with the training of NNmodel.
We can visualise the TensorBoard logging data with the help of following command −
Tensorboard –logdir logsCNTK - Convolutional Neural Network
In this chapter, let us study how to construct a Convolutional Neural Network (CNN) in CNTK.
Introduction
Convolutional neural networks (CNNs) are also made up of neurons, that have learnable weights and biases. That’s why in this manner, they are like ordinary neural networks (NNs).
If we recall the working of ordinary NNs, every neuron receives one or more inputs, takes a weighted sum and it passed through an activation function to produce the final output. Here, the question arises that if CNNs and ordinary NNs have so many similarities then what makes these two networks different to each other?
What makes them different is the treatment of input data and types of layers? The structure of input data is ignored in ordinary NN and all the data is converted into 1-D array before feeding it into the network.
But, Convolutional Neural Network architecture can consider the 2D structure of the images, process them and allow it to extract the properties that are specific to images. Moreover, CNNs have the advantage of having one or more Convolutional layers and pooling layer, which are the main building blocks of CNNs.
These layers are followed by one or more fully connected layers as in standard multilayer NNs. So, we can think of CNN, as a special case of fully connected networks.
Convolutional Neural Network (CNN) architecture
The architecture of CNN is basically a list of layers that transforms the 3-dimensional, i.e. width, height and depth of image volume into a 3-dimensional output volume. One important point to note here is that, every neuron in the current layer is connected to a small patch of the output from the previous layer, which is like overlaying a N*N filter on the input image.
It uses M filters, which are basically feature extractors that extract features like edges, corner and so on. Following are the layers [INPUT-CONV-RELU-POOL-FC] that are used to construct Convolutional neural networks (CNNs)−
INPUT− As the name implies, this layer holds the raw pixel values. Raw pixel values mean the data of the image as it is. Example, INPUT [64×64×3] is a 3-channeled RGB image of width-64, height-64 and depth-3.
CONV− This layer is one of the building blocks of CNNs as most of the computation is done in this layer. Example - if we use 6 filters on the above mentioned INPUT [64×64×3], this may result in the volume [64×64×6].
RELU−Also called rectified linear unit layer, that applies an activation function to the output of previous layer. In other manner, a non-linearity would be added to the network by RELU.
POOL− This layer, i.e. Pooling layer is one other building block of CNNs. The main task of this layer is down-sampling, which means it operates independently on every slice of the input and resizes it spatially.
FC− It is called Fully Connected layer or more specifically the output layer. It is used to compute output class score and the resulting output is volume of the size 1*1*L where L is the number corresponding to class score.
The diagram below represents the typical architecture of CNNs−

Creating CNN structure
We have seen the architecture and the basics of CNN, now we are going to building convolutional network using CNTK. Here, we will first see how to put together the structure of the CNN and then we will look at how to train the parameters of it.
At last we’ll see, how we can improve the neural network by changing its structure with various different layer setups. We are going to use MNIST image dataset.
So, first let’s create a CNN structure. Generally, when we build a CNN for recognizing patterns in images, we do the following−
We use a combination of convolution and pooling layers.
One or more hidden layer at the end of the network.
At last, we finish the network with a softmax layer for classification purpose.
With the help of following steps, we can build the network structure−
Step 1− First, we need to import the required layers for CNN.
from cntk.layers import Convolution2D, Sequential, Dense, MaxPoolingStep 2− Next, we need to import the activation functions for CNN.
from cntk.ops import log_softmax, reluStep 3− After that in order to initialize the convolutional layers later, we need to import the glorot_uniform_initializer as follows−
from cntk.initializer import glorot_uniformStep 4− Next, to create input variables import the input_variable function. And import default_option function, to make configuration of NN a bit easier.
from cntk import input_variable, default_optionsStep 5− Now to store the input images, create a new input_variable. It will contain three channels namely red, green and blue. It would have the size of 28 by 28 pixels.
features = input_variable((3,28,28))Step 6−Next, we need to create another input_variable to store the labels to predict.
labels = input_variable(10)Step 7− Now, we need to create the default_option for the NN. And, we need to use the glorot_uniform as the initialization function.
with default_options(initialization=glorot_uniform, activation=relu):Step 8− Next, in order to set the structure of the NN, we need to create a new Sequential layer set.
Step 9− Now we need to add a Convolutional2D layer with a filter_shape of 5 and a strides setting of 1, within the Sequential layer set. Also, enable padding, so that the image is padded to retain the original dimensions.
model = Sequential([
Convolution2D(filter_shape=(5,5), strides=(1,1), num_filters=8, pad=True),Step 10− Now it’s time to add a MaxPooling layer with filter_shape of 2, and a strides setting of 2 to compress the image by half.
MaxPooling(filter_shape=(2,2), strides=(2,2)),Step 11− Now, as we did in step 9, we need to add another Convolutional2D layer with a filter_shape of 5 and a strides setting of 1, use 16 filters. Also, enable padding, so that, the size of the image produced by the previous pooling layer should be retained.
Convolution2D(filter_shape=(5,5), strides=(1,1), num_filters=16, pad=True),Step 12− Now, as we did in step 10, add another MaxPooling layer with a filter_shape of 3 and a strides setting of 3 to reduce the image to a third.
MaxPooling(filter_shape=(3,3), strides=(3,3)),Step 13− At last, add a Dense layer with ten neurons for the 10 possible classes, the network can predict. In order to turn the network into a classification model, use a log_siftmax activation function.
Dense(10, activation=log_softmax)
])Complete Example for creating CNN structure
from cntk.layers import Convolution2D, Sequential, Dense, MaxPooling
from cntk.ops import log_softmax, relu
from cntk.initializer import glorot_uniform
from cntk import input_variable, default_options
features = input_variable((3,28,28))
labels = input_variable(10)
with default_options(initialization=glorot_uniform, activation=relu):
model = Sequential([
Convolution2D(filter_shape=(5,5), strides=(1,1), num_filters=8, pad=True),
MaxPooling(filter_shape=(2,2), strides=(2,2)),
Convolution2D(filter_shape=(5,5), strides=(1,1), num_filters=16, pad=True),
MaxPooling(filter_shape=(3,3), strides=(3,3)),
Dense(10, activation=log_softmax)
])
z = model(features)Training CNN with images
As we have created the structure of the network, it’s time to train the network. But before starting the training of our network, we need to set up minibatch sources, because training a NN that works with images requires more memory, than most computers have.
We have already created minibatch sources in previous sections. Following is the Python code to set up two minibatch sources −
As we have the create_datasource function, we can now create two separate data sources (training and testing one) to train the model.
train_datasource = create_datasource('mnist_train')
test_datasource = create_datasource('mnist_test', max_sweeps=1, train=False)Now, as we have prepared the images, we can start training of our NN. As we did in previous sections, we can use the train method on the loss function to kick off the training. Following is the code for this −
from cntk import Function
from cntk.losses import cross_entropy_with_softmax
from cntk.metrics import classification_error
from cntk.learners import sgd
@Function
def criterion_factory(output, targets):
loss = cross_entropy_with_softmax(output, targets)
metric = classification_error(output, targets)
return loss, metric
loss = criterion_factory(z, labels)
learner = sgd(z.parameters, lr=0.2)With the help of previous code, we have setup the loss and learner for the NN. The following code will train and validate the NN−
from cntk.logging import ProgressPrinter
from cntk.train import TestConfig
progress_writer = ProgressPrinter(0)
test_config = TestConfig(test_datasource)
input_map = {
features: train_datasource.streams.features,
labels: train_datasource.streams.labels
}
loss.train(train_datasource,
max_epochs=10,
minibatch_size=64,
epoch_size=60000,
parameter_learners=[learner],
model_inputs_to_streams=input_map,
callbacks=[progress_writer, test_config])Complete Implementation Example
from cntk.layers import Convolution2D, Sequential, Dense, MaxPooling
from cntk.ops import log_softmax, relu
from cntk.initializer import glorot_uniform
from cntk import input_variable, default_options
features = input_variable((3,28,28))
labels = input_variable(10)
with default_options(initialization=glorot_uniform, activation=relu):
model = Sequential([
Convolution2D(filter_shape=(5,5), strides=(1,1), num_filters=8, pad=True),
MaxPooling(filter_shape=(2,2), strides=(2,2)),
Convolution2D(filter_shape=(5,5), strides=(1,1), num_filters=16, pad=True),
MaxPooling(filter_shape=(3,3), strides=(3,3)),
Dense(10, activation=log_softmax)
])
z = model(features)
import os
from cntk.io import MinibatchSource, StreamDef, StreamDefs, ImageDeserializer, INFINITELY_REPEAT
import cntk.io.transforms as xforms
def create_datasource(folder, train=True, max_sweeps=INFINITELY_REPEAT):
mapping_file = os.path.join(folder, 'mapping.bin')
image_transforms = []
if train:
image_transforms += [
xforms.crop(crop_type='randomside', side_ratio=0.8),
xforms.scale(width=28, height=28, channels=3, interpolations='linear')
]
stream_definitions = StreamDefs(
features=StreamDef(field='image', transforms=image_transforms),
labels=StreamDef(field='label', shape=10)
)
deserializer = ImageDeserializer(mapping_file, stream_definitions)
return MinibatchSource(deserializer, max_sweeps=max_sweeps)
train_datasource = create_datasource('mnist_train')
test_datasource = create_datasource('mnist_test', max_sweeps=1, train=False)
from cntk import Function
from cntk.losses import cross_entropy_with_softmax
from cntk.metrics import classification_error
from cntk.learners import sgd
@Function
def criterion_factory(output, targets):
loss = cross_entropy_with_softmax(output, targets)
metric = classification_error(output, targets)
return loss, metric
loss = criterion_factory(z, labels)
learner = sgd(z.parameters, lr=0.2)
from cntk.logging import ProgressPrinter
from cntk.train import TestConfig
progress_writer = ProgressPrinter(0)
test_config = TestConfig(test_datasource)
input_map = {
features: train_datasource.streams.features,
labels: train_datasource.streams.labels
}
loss.train(train_datasource,
max_epochs=10,
minibatch_size=64,
epoch_size=60000,
parameter_learners=[learner],
model_inputs_to_streams=input_map,
callbacks=[progress_writer, test_config])Output
-------------------------------------------------------------------
average since average since examples
loss last metric last
------------------------------------------------------
Learning rate per minibatch: 0.2
142 142 0.922 0.922 64
1.35e+06 1.51e+07 0.896 0.883 192
[………]Image transformations
As we have seen, it’s difficult to train NN used for image recognition and, they require a lot of data to train also. One more issue is that, they tend to overfit on images used during training. Let us see with an example, when we have photos of faces in an upright position, our model will have a hard time recognizing faces that are rotated in another direction.
In order to overcome such problem, we can use image augmentation and CNTK supports specific transforms, when creating minibatch sources for images. We can use several transformations as follows−
We can randomly crop images used for training with just a few lines of code.
We can use a scale and color also.
Let’s see with the help of following Python code, how we can change the list of transformations by including a cropping transformation within the function used to create the minibatch source earlier.
import os
from cntk.io import MinibatchSource, StreamDef, StreamDefs, ImageDeserializer, INFINITELY_REPEAT
import cntk.io.transforms as xforms
def create_datasource(folder, train=True, max_sweeps=INFINITELY_REPEAT):
mapping_file = os.path.join(folder, 'mapping.bin')
image_transforms = []
if train:
image_transforms += [
xforms.crop(crop_type='randomside', side_ratio=0.8),
xforms.scale(width=28, height=28, channels=3, interpolations='linear')
]
stream_definitions = StreamDefs(
features=StreamDef(field='image', transforms=image_transforms),
labels=StreamDef(field='label', shape=10)
)
deserializer = ImageDeserializer(mapping_file, stream_definitions)
return MinibatchSource(deserializer, max_sweeps=max_sweeps)With the help of above code, we can enhance the function to include a set of image transforms, so that, when we will be training we can randomly crop the image, so we get more variations of the image.
CNTK - Recurrent Neural Network
Now, let us understand how to construct a Recurrent Neural Network (RNN) in CNTK.
Introduction
We learned how to classify images with a neural network, and it is one of the iconic jobs in deep learning. But, another area where neural network excels at and lot of research happening is Recurrent Neural Networks (RNN). Here, we are going to know what RNN is and how it can be used in scenarios where we need to deal with time-series data.
What is Recurrent Neural Network?
Recurrent neural networks (RNNs) may be defined as the special breed of NNs that are capable of reasoning over time. RNNs are mainly used in scenarios, where we need to deal with values that change over time, i.e. time-series data. In order to understand it in a better way, let’s have a small comparison between regular neural networks and recurrent neural networks −
As we know that, in a regular neural network, we can provide only one input. This limits it to results in only one prediction. To give you an example, we can do translating text job by using regular neural networks.
On the other hand, in recurrent neural networks, we can provide a sequence of samples that result in a single prediction. In other words, using RNNs we can predict an output sequence based on an input sequence. For example, there have been quite a few successful experiments with RNN in translation tasks.
Uses of Recurrent Neural Network
RNNs can be used in several ways. Some of them are as follows −
Predicting a single output
Before getting deep dive into the steps, that how RNN can predict a single output based on a sequence, let’s see how a basic RNN looks like−

As we can in the above diagram, RNN contains a loopback connection to the input and whenever, we feed a sequence of values it will process each element in the sequence as time steps.
Moreover, because of the loopback connection, RNN can combine the generated output with input for the next element in the sequence. In this way, RNN will build a memory over the whole sequence which can be used to make a prediction.
In order to make prediction with RNN, we can perform the following steps−
First, to create an initial hidden state, we need to feed the first element of the input sequence.
After that, to produce an updated hidden state, we need to take the initial hidden state and combine it with the second element in the input sequence.
At last, to produce the final hidden state and to predict the output for the RNN, we need to take the final element in the input sequence.
In this way, with the help of this loopback connection we can teach a RNN to recognize patterns that happen over time.
Predicting a sequence
The basic model, discussed above, of RNN can be extended to other use cases as well. For example, we can use it to predict a sequence of values based on a single input. In this scenario, order to make prediction with RNN we can perform the following steps −
First, to create an initial hidden state and predict the first element in the output sequence, we need to feed an input sample into the neural network.
After that, to produce an updated hidden state and the second element in the output sequence, we need to combine the initial hidden state with the same sample.
At last, to update the hidden state one more time and predict the final element in output sequence, we feed the sample another time.
Predicting sequences
As we have seen how to predict a single value based on a sequence and how to predict a sequence based on a single value. Now let’s see how we can predict sequences for sequences. In this scenario, order to make prediction with RNN we can perform the following steps −
First, to create an initial hidden state and predict the first element in the output sequence, we need to take the first element in the input sequence.
After that, to update the hidden state and predict the second element in the output sequence, we need to take the initial hidden state.
At last, to predict the final element in the output sequence, we need to take the updated hidden state and the final element in the input sequence.
Working of RNN
To understand the working of recurrent neural networks (RNNs) we need to first understand how recurrent layers in the network work. So first let’s discuss how e can predict the output with a standard recurrent layer.
Predicting output with standard RNN layer
As we discussed earlier also that a basic layer in RNN is quite different from a regular layer in a neural network. In previous section, we also demonstrated in the diagram the basic architecture of RNN. In order to update the hidden state for the first-time step-in sequence we can use the following formula −

In the above equation, we calculate the new hidden state by calculating the dot product between the initial hidden state and a set of weights.
Now for the next step, the hidden state for the current time step is used as the initial hidden state for the next time step in the sequence. That’s why, to update the hidden state for the second time step, we can repeat the calculations performed in the first-time step as follows −
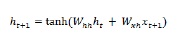
Next, we can repeat the process of updating the hidden state for the third and final step in the sequence as below −

And when we have processed all the above steps in the sequence, we can calculate the output as follows −

For the above formula, we have used a third set of weights and the hidden state from the final time step.
Advanced Recurrent Units
The main issue with basic recurrent layer is of vanishing gradient problem and due to this it is not very good at learning long-term correlations. In simple words basic recurrent layer does not handle long sequences very well. That’s the reason some other recurrent layer types that are much more suited for working with longer sequences are as follows −
Long-Short Term Memory (LSTM)

Long-short term memory (LSTMs) networks were introduced by Hochreiter & Schmidhuber. It solved the problem of getting a basic recurrent layer to remember things for a long time. The architecture of LSTM is given above in the diagram. As we can see it has input neurons, memory cells, and output neurons. In order to combat the vanishing gradient problem, Long-short term memory networks use an explicit memory cell (stores the previous values) and the following gates −
Forget gate− As name implies, it tells the memory cell to forget the previous values. The memory cell stores the values until the gate i.e. ‘forget gate’ tells it to forget them.
Input gate− As name implies, it adds new stuff to the cell.
Output gate− As name implies, output gate decides when to pass along the vectors from the cell to the next hidden state.
Gated Recurrent Units (GRUs)

Gradient recurrent units (GRUs) is a slight variation of LSTMs network. It has one less gate and are wired slightly different than LSTMs. Its architecture is shown in the above diagram. It has input neurons, gated memory cells, and output neurons. Gated Recurrent Units network has the following two gates −
Update gate− It determines the following two things−
What amount of the information should be kept from the last state?
What amount of the information should be let in from the previous layer?
Reset gate− The functionality of reset gate is much like that of forget gate of LSTMs network. The only difference is that it is located slightly differently.
In contrast to Long-short term memory network, Gated Recurrent Unit networks are slightly faster and easier to run.
Creating RNN structure
Before we can start, making prediction about the output from any of our data source, we need to first construct RNN and constructing RNN is quite same as we had build regular neural network in previous section. Following is the code to build one−
from cntk.losses import squared_error
from cntk.io import CTFDeserializer, MinibatchSource, INFINITELY_REPEAT, StreamDefs, StreamDef
from cntk.learners import adam
from cntk.logging import ProgressPrinter
from cntk.train import TestConfig
BATCH_SIZE = 14 * 10
EPOCH_SIZE = 12434
EPOCHS = 10Staking multiple layers
We can also stack multiple recurrent layers in CNTK. For example, we can use the following combination of layers−
from cntk import sequence, default_options, input_variable
from cntk.layers import Recurrence, LSTM, Dropout, Dense, Sequential, Fold
features = sequence.input_variable(1)
with default_options(initial_state = 0.1):
model = Sequential([
Fold(LSTM(15)),
Dense(1)
])(features)
target = input_variable(1, dynamic_axes=model.dynamic_axes)As we can see in the above code, we have the following two ways in which we can model RNN in CNTK −
First, if we only want the final output of a recurrent layer, we can use the Fold layer in combination with a recurrent layer, such as GRU, LSTM, or even RNNStep.
Second, as an alternative way, we can also use the Recurrence block.
Training RNN with time series data
Once we build the model, let’s see how we can train RNN in CNTK −
from cntk import Function
@Function
def criterion_factory(z, t):
loss = squared_error(z, t)
metric = squared_error(z, t)
return loss, metric
loss = criterion_factory(model, target)
learner = adam(model.parameters, lr=0.005, momentum=0.9)Now to load the data into the training process, we must have to deserialize sequences from a set of CTF files. Following code have the create_datasource function, which is a useful utility function to create both the training and test datasource.
target_stream = StreamDef(field='target', shape=1, is_sparse=False)
features_stream = StreamDef(field='features', shape=1, is_sparse=False)
deserializer = CTFDeserializer(filename, StreamDefs(features=features_stream, target=target_stream))
datasource = MinibatchSource(deserializer, randomize=True, max_sweeps=sweeps)
return datasource
train_datasource = create_datasource('Training data filename.ctf')#we need to provide the location of training file we created from our dataset.
test_datasource = create_datasource('Test filename.ctf', sweeps=1) #we need to provide the location of testing file we created from our dataset.Now, as we have setup the data sources, model and the loss function, we can start the training process. It is quite similar as we did in previous sections with basic neural networks.
progress_writer = ProgressPrinter(0)
test_config = TestConfig(test_datasource)
input_map = {
features: train_datasource.streams.features,
target: train_datasource.streams.target
}
history = loss.train(
train_datasource,
epoch_size=EPOCH_SIZE,
parameter_learners=[learner],
model_inputs_to_streams=input_map,
callbacks=[progress_writer, test_config],
minibatch_size=BATCH_SIZE,
max_epochs=EPOCHS
)We will get the output similar as follows −
Output−
average since average since examples
loss last metric last
------------------------------------------------------
Learning rate per minibatch: 0.005
0.4 0.4 0.4 0.4 19
0.4 0.4 0.4 0.4 59
0.452 0.495 0.452 0.495 129
[…]Validating the model
Actually redicting with a RNN is quite similar to making predictions with any other CNK model. The only difference is that, we need to provide sequences rather than single samples.
Now, as our RNN is finally done with training, we can validate the model by testing it using a few samples sequence as follows −
import pickle
with open('test_samples.pkl', 'rb') as test_file:
test_samples = pickle.load(test_file)
model(test_samples) * NORMALIZEOutput−
array([[ 8081.7905],
[16597.693 ],
[13335.17 ],
...,
[11275.804 ],
[15621.697 ],
[16875.555 ]], dtype=float32)