Big Data Analytics - Metodi statistici
Quando si analizzano i dati, è possibile avere un approccio statistico. Gli strumenti di base necessari per eseguire l'analisi di base sono:
- Analisi di correlazione
- Analisi della varianza
- Verifica di ipotesi
Quando si lavora con set di dati di grandi dimensioni, non comporta alcun problema in quanto questi metodi non sono intensivi dal punto di vista computazionale, ad eccezione dell'analisi di correlazione. In questo caso, è sempre possibile prelevare un campione ei risultati dovrebbero essere robusti.
Analisi di correlazione
L'analisi della correlazione cerca di trovare relazioni lineari tra variabili numeriche. Questo può essere utile in diverse circostanze. Un uso comune è l'analisi esplorativa dei dati, nella sezione 16.0.2 del libro c'è un esempio di base di questo approccio. Prima di tutto, la metrica di correlazione utilizzata nell'esempio citato si basa sulPearson coefficient. Esiste tuttavia un'altra metrica di correlazione interessante che non è influenzata dai valori anomali. Questa metrica è chiamata correlazione spearman.
Il spearman correlation la metrica è più robusta alla presenza di valori anomali rispetto al metodo Pearson e fornisce stime migliori delle relazioni lineari tra variabili numeriche quando i dati non sono distribuiti normalmente.
library(ggplot2)
# Select variables that are interesting to compare pearson and spearman
correlation methods.
x = diamonds[, c('x', 'y', 'z', 'price')]
# From the histograms we can expect differences in the correlations of both
metrics.
# In this case as the variables are clearly not normally distributed, the
spearman correlation
# is a better estimate of the linear relation among numeric variables.
par(mfrow = c(2,2))
colnm = names(x)
for(i in 1:4) {
hist(x[[i]], col = 'deepskyblue3', main = sprintf('Histogram of %s', colnm[i]))
}
par(mfrow = c(1,1))Dagli istogrammi nella figura seguente, possiamo aspettarci differenze nelle correlazioni di entrambe le metriche. In questo caso, poiché le variabili sono chiaramente non distribuite normalmente, la correlazione spearman è una stima migliore della relazione lineare tra variabili numeriche.
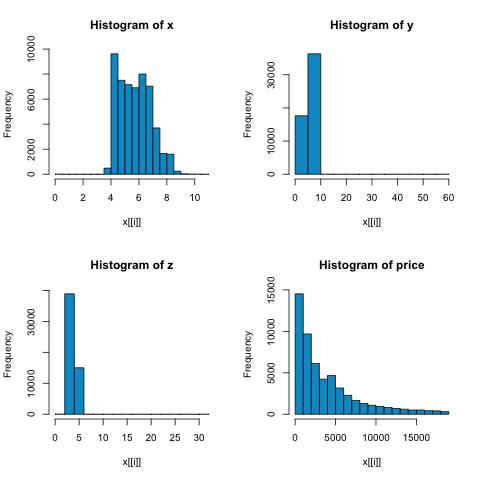
Per calcolare la correlazione in R, apri il file bda/part2/statistical_methods/correlation/correlation.R che ha questa sezione di codice.
## Correlation Matrix - Pearson and spearman
cor_pearson <- cor(x, method = 'pearson')
cor_spearman <- cor(x, method = 'spearman')
### Pearson Correlation
print(cor_pearson)
# x y z price
# x 1.0000000 0.9747015 0.9707718 0.8844352
# y 0.9747015 1.0000000 0.9520057 0.8654209
# z 0.9707718 0.9520057 1.0000000 0.8612494
# price 0.8844352 0.8654209 0.8612494 1.0000000
### Spearman Correlation
print(cor_spearman)
# x y z price
# x 1.0000000 0.9978949 0.9873553 0.9631961
# y 0.9978949 1.0000000 0.9870675 0.9627188
# z 0.9873553 0.9870675 1.0000000 0.9572323
# price 0.9631961 0.9627188 0.9572323 1.0000000Test chi quadrato
Il test del chi quadrato ci consente di verificare se due variabili casuali sono indipendenti. Ciò significa che la distribuzione di probabilità di ciascuna variabile non influenza l'altra. Per valutare il test in R dobbiamo prima creare una tabella di contingenza, quindi passare la tabella achisq.test R funzione.
Ad esempio, controlliamo se esiste un'associazione tra le variabili: taglio e colore dal dataset dei diamanti. Il test è formalmente definito come:
- H0: Il taglio variabile e il diamante sono indipendenti
- H1: Il taglio variabile e il diamante non sono indipendenti
Presumiamo che esista una relazione tra queste due variabili in base al loro nome, ma il test può fornire una "regola" oggettiva che indica quanto sia significativo questo risultato o meno.
Nel seguente frammento di codice, abbiamo scoperto che il valore p del test è 2.2e-16, questo è quasi zero in termini pratici. Quindi dopo aver eseguito il test facendo un fileMonte Carlo simulation, abbiamo scoperto che il valore p è 0,0004998, che è ancora abbastanza inferiore alla soglia 0,05. Questo risultato significa che rifiutiamo l'ipotesi nulla (H0), quindi crediamo alle variabilicut e color non sono indipendenti.
library(ggplot2)
# Use the table function to compute the contingency table
tbl = table(diamonds$cut, diamonds$color)
tbl
# D E F G H I J
# Fair 163 224 312 314 303 175 119
# Good 662 933 909 871 702 522 307
# Very Good 1513 2400 2164 2299 1824 1204 678
# Premium 1603 2337 2331 2924 2360 1428 808
# Ideal 2834 3903 3826 4884 3115 2093 896
# In order to run the test we just use the chisq.test function.
chisq.test(tbl)
# Pearson’s Chi-squared test
# data: tbl
# X-squared = 310.32, df = 24, p-value < 2.2e-16
# It is also possible to compute the p-values using a monte-carlo simulation
# It's needed to add the simulate.p.value = TRUE flag and the amount of
simulations
chisq.test(tbl, simulate.p.value = TRUE, B = 2000)
# Pearson’s Chi-squared test with simulated p-value (based on 2000 replicates)
# data: tbl
# X-squared = 310.32, df = NA, p-value = 0.0004998T-test
L'idea di t-testè valutare se ci sono differenze nella distribuzione di una variabile numerica # tra diversi gruppi di una variabile nominale. Per dimostrarlo, selezionerò i livelli dei livelli Giusto e Ideale del taglio della variabile fattoriale, quindi confronteremo i valori di una variabile numerica tra questi due gruppi.
data = diamonds[diamonds$cut %in% c('Fair', 'Ideal'), ]
data$cut = droplevels.factor(data$cut) # Drop levels that aren’t used from the
cut variable
df1 = data[, c('cut', 'price')]
# We can see the price means are different for each group
tapply(df1$price, df1$cut, mean)
# Fair Ideal
# 4358.758 3457.542I test t sono implementati in R con l'estensione t.testfunzione. L'interfaccia della formula a t.test è il modo più semplice per usarla, l'idea è che una variabile numerica sia spiegata da una variabile di gruppo.
Per esempio: t.test(numeric_variable ~ group_variable, data = data). Nell'esempio precedente, ilnumeric_variable è price e il group_variable è cut.
Da un punto di vista statistico, stiamo testando se ci sono differenze nelle distribuzioni della variabile numerica tra due gruppi. Formalmente il test di ipotesi è descritto con un'ipotesi nulla (H0) e un'ipotesi alternativa (H1).
H0: Non ci sono differenze nelle distribuzioni della variabile prezzo tra i gruppi Fair e Ideal
H1 Ci sono differenze nelle distribuzioni della variabile prezzo tra i gruppi Equo e Ideale
Quanto segue può essere implementato in R con il codice seguente:
t.test(price ~ cut, data = data)
# Welch Two Sample t-test
#
# data: price by cut
# t = 9.7484, df = 1894.8, p-value < 2.2e-16
# alternative hypothesis: true difference in means is not equal to 0
# 95 percent confidence interval:
# 719.9065 1082.5251
# sample estimates:
# mean in group Fair mean in group Ideal
# 4358.758 3457.542
# Another way to validate the previous results is to just plot the
distributions using a box-plot
plot(price ~ cut, data = data, ylim = c(0,12000),
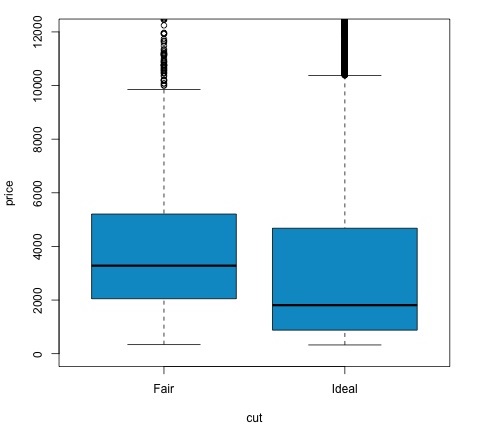
col = 'deepskyblue3')Possiamo analizzare il risultato del test controllando se il valore p è inferiore a 0,05. In tal caso, manteniamo l'ipotesi alternativa. Ciò significa che abbiamo riscontrato differenze di prezzo tra i due livelli del fattore di taglio. Dai nomi dei livelli ci saremmo aspettati questo risultato, ma non ci saremmo aspettati che il prezzo medio nel gruppo Fail sarebbe stato più alto che nel gruppo Ideal. Possiamo vederlo confrontando le medie di ciascun fattore.
Il plotIl comando produce un grafico che mostra la relazione tra il prezzo e la variabile di taglio. È un box-plot; abbiamo trattato questo grafico nella sezione 16.0.1 ma sostanzialmente mostra la distribuzione della variabile di prezzo per i due livelli di taglio che stiamo analizzando.
Analisi della varianza
L'analisi della varianza (ANOVA) è un modello statistico utilizzato per analizzare le differenze tra la distribuzione di gruppo confrontando la media e la varianza di ciascun gruppo, il modello è stato sviluppato da Ronald Fisher. ANOVA fornisce un test statistico per stabilire se le medie di più gruppi sono uguali e quindi generalizza il test t a più di due gruppi.
Gli ANOVA sono utili per confrontare tre o più gruppi per la significatività statistica perché l'esecuzione di più test t a due campioni comporterebbe una maggiore possibilità di commettere un errore statistico di tipo I.
In termini di fornire una spiegazione matematica, è necessario quanto segue per comprendere il test.
x ij = x + (x io - x) + (x ij - x)
Questo porta al seguente modello:
x ij = μ + α io + ∈ ij
dove μ è la media generale e α i è l'i esima media del gruppo. Si presume che il termine di errore ∈ ij sia iid da una distribuzione normale. L'ipotesi nulla del test è che:
α 1 = α 2 =… = α k
In termini di calcolo della statistica del test, dobbiamo calcolare due valori:
- Somma dei quadrati per differenza tra gruppi -
$$ SSD_B = \ sum_ {i} ^ {k} \ sum_ {j} ^ {n} (\ bar {x _ {\ bar {i}}} - \ bar {x}) ^ 2 $$
- Somma dei quadrati all'interno dei gruppi
$$ SSD_W = \ sum_ {i} ^ {k} \ sum_ {j} ^ {n} (\ bar {x _ {\ bar {ij}}} - \ bar {x _ {\ bar {i}}}) ^ 2 $$
dove SSD B ha un grado di libertà di k − 1 e SSD W ha un grado di libertà di N − k. Quindi possiamo definire le differenze quadratiche medie per ciascuna metrica.
MS B = SSD B / (k - 1)
MS w = SSD con (N - k)
Infine, la statistica del test in ANOVA è definita come il rapporto tra le due quantità di cui sopra
F = MS B / MS w
che segue una distribuzione F con k − 1 e N − k gradi di libertà. Se l'ipotesi nulla è vera, F sarebbe probabilmente vicino a 1. In caso contrario, è probabile che il quadrato medio MSB tra i gruppi sia grande, il che si traduce in un valore F elevato.
Fondamentalmente, ANOVA esamina le due fonti della varianza totale e vede quale parte contribuisce di più. Questo è il motivo per cui si chiama analisi della varianza sebbene l'intenzione sia quella di confrontare le medie di gruppo.
In termini di calcolo della statistica, in realtà è piuttosto semplice da fare in R. Il seguente esempio dimostrerà come è fatto e traccia i risultati.
library(ggplot2)
# We will be using the mtcars dataset
head(mtcars)
# mpg cyl disp hp drat wt qsec vs am gear carb
# Mazda RX4 21.0 6 160 110 3.90 2.620 16.46 0 1 4 4
# Mazda RX4 Wag 21.0 6 160 110 3.90 2.875 17.02 0 1 4 4
# Datsun 710 22.8 4 108 93 3.85 2.320 18.61 1 1 4 1
# Hornet 4 Drive 21.4 6 258 110 3.08 3.215 19.44 1 0 3 1
# Hornet Sportabout 18.7 8 360 175 3.15 3.440 17.02 0 0 3 2
# Valiant 18.1 6 225 105 2.76 3.460 20.22 1 0 3 1
# Let's see if there are differences between the groups of cyl in the mpg variable.
data = mtcars[, c('mpg', 'cyl')]
fit = lm(mpg ~ cyl, data = mtcars)
anova(fit)
# Analysis of Variance Table
# Response: mpg
# Df Sum Sq Mean Sq F value Pr(>F)
# cyl 1 817.71 817.71 79.561 6.113e-10 ***
# Residuals 30 308.33 10.28
# Signif. codes: 0 *** 0.001 ** 0.01 * 0.05 .
# Plot the distribution
plot(mpg ~ as.factor(cyl), data = mtcars, col = 'deepskyblue3')Il codice produrrà il seguente output:

Il valore p che otteniamo nell'esempio è significativamente inferiore a 0,05, quindi R restituisce il simbolo "***" per denotarlo. Significa che rifiutiamo l'ipotesi nulla e che troviamo differenze tra le medie mpg tra i diversi gruppi dicyl variabile.