AI with Python - Supervised Learning: Classification
W tym rozdziale skupimy się na wdrożeniu uczenia nadzorowanego - klasyfikacji.
Technika lub model klasyfikacji próbuje wyciągnąć wnioski z zaobserwowanych wartości. W przypadku problemu klasyfikacji mamy podzielone na kategorie wyniki, takie jak „Czarny” lub „biały” lub „Nauczanie” i „Nienauczanie”. Podczas budowania modelu klasyfikacji musimy mieć zbiór danych szkoleniowych zawierający punkty danych i odpowiadające im etykiety. Na przykład, jeśli chcemy sprawdzić, czy obraz przedstawia samochód, czy nie. Aby to sprawdzić, utworzymy zbiór danych szkoleniowych, zawierający dwie klasy związane z „samochodem” i „bez samochodu”. Następnie musimy wytrenować model przy użyciu próbek uczących. Modele klasyfikacyjne są używane głównie w rozpoznawaniu twarzy, identyfikacji spamu itp.
Kroki tworzenia klasyfikatora w Pythonie
Aby zbudować klasyfikator w Pythonie, użyjemy Pythona 3 i Scikit-learn, które jest narzędziem do uczenia maszynowego. Wykonaj następujące kroki, aby zbudować klasyfikator w Pythonie -
Krok 1 - Importuj Scikit-learn
Byłby to pierwszy krok do zbudowania klasyfikatora w Pythonie. W tym kroku zainstalujemy pakiet Pythona o nazwie Scikit-learn, który jest jednym z najlepszych modułów uczenia maszynowego w Pythonie. Następujące polecenie pomoże nam zaimportować pakiet -
Import SklearnKrok 2 - Importuj zestaw danych Scikit-learn
Na tym etapie możemy rozpocząć pracę ze zbiorem danych dla naszego modelu uczenia maszynowego. Tutaj będziemy używaćthe Breast Cancer Wisconsin Diagnostic Database. Zbiór danych zawiera różne informacje o guzach raka piersi, a także etykiety klasyfikacyjnemalignant lub benign. Zestaw danych zawiera 569 instancji lub dane dotyczące 569 guzów i zawiera informacje o 30 atrybutach lub cechach, takich jak promień guza, tekstura, gładkość i powierzchnia. Za pomocą następującego polecenia możemy zaimportować zestaw danych raka piersi Scikit-learn -
from sklearn.datasets import load_breast_cancerTeraz następujące polecenie załaduje zestaw danych.
data = load_breast_cancer()Poniżej znajduje się lista ważnych kluczy słownika -
- Nazwy etykiet klasyfikacyjnych (target_names)
- Rzeczywiste etykiety (cel)
- Nazwy atrybutów / cech (nazwy cech)
- Atrybut (dane)
Teraz za pomocą następującego polecenia możemy utworzyć nowe zmienne dla każdego ważnego zestawu informacji i przypisać dane. Innymi słowy, możemy uporządkować dane za pomocą następujących poleceń -
label_names = data['target_names']
labels = data['target']
feature_names = data['feature_names']
features = data['data']Teraz, aby było jaśniej, możemy wydrukować etykiety klas, etykietę pierwszej instancji danych, nasze nazwy funkcji i wartość funkcji za pomocą następujących poleceń -
print(label_names)Powyższe polecenie wypisze nazwy klas, które są odpowiednio złośliwe i łagodne. Jest to pokazane jako dane wyjściowe poniżej -
['malignant' 'benign']Teraz poniższe polecenie pokaże, że są one odwzorowane na wartości binarne 0 i 1. Tutaj 0 oznacza raka złośliwego, a 1 oznacza raka łagodnego. Otrzymasz następujący wynik -
print(labels[0])
0Dwa polecenia podane poniżej spowodują nazwy funkcji i ich wartości.
print(feature_names[0])
mean radius
print(features[0])
[ 1.79900000e+01 1.03800000e+01 1.22800000e+02 1.00100000e+03
1.18400000e-01 2.77600000e-01 3.00100000e-01 1.47100000e-01
2.41900000e-01 7.87100000e-02 1.09500000e+00 9.05300000e-01
8.58900000e+00 1.53400000e+02 6.39900000e-03 4.90400000e-02
5.37300000e-02 1.58700000e-02 3.00300000e-02 6.19300000e-03
2.53800000e+01 1.73300000e+01 1.84600000e+02 2.01900000e+03
1.62200000e-01 6.65600000e-01 7.11900000e-01 2.65400000e-01
4.60100000e-01 1.18900000e-01]Z powyższego wyniku widać, że pierwszą instancją danych jest nowotwór złośliwy, którego promień wynosi 1,7990000e + 01.
Krok 3 - Organizowanie danych w zestawy
W tym kroku podzielimy nasze dane na dwie części, a mianowicie zbiór uczący i zbiór testowy. Podział danych na te zbiory jest bardzo ważny, ponieważ musimy przetestować nasz model na niewidocznych danych. Aby podzielić dane na zestawy, sklearn ma funkcję o nazwietrain_test_split()funkcjonować. Za pomocą następujących poleceń możemy podzielić dane w tych zestawach -
from sklearn.model_selection import train_test_splitPowyższe polecenie zaimportuje plik train_test_splitfunction ze sklearn, a poniższe polecenie podzieli dane na dane treningowe i testowe. W poniższym przykładzie wykorzystujemy 40% danych do testowania, a pozostałe dane zostaną wykorzystane do uczenia modelu.
train, test, train_labels, test_labels = train_test_split(features,labels,test_size = 0.40, random_state = 42)Krok 4 - Budowa modelu
W tym kroku będziemy budować nasz model. Do budowy modelu użyjemy algorytmu Naïve Bayes. Do zbudowania modelu można użyć następujących poleceń -
from sklearn.naive_bayes import GaussianNBPowyższe polecenie zaimportuje moduł GaussianNB. Teraz poniższe polecenie pomoże Ci zainicjować model.
gnb = GaussianNB()Będziemy trenować model, dopasowując go do danych za pomocą gnb.fit ().
model = gnb.fit(train, train_labels)Krok 5 - Ocena modelu i jego dokładności
Na tym etapie ocenimy model, wykonując prognozy na naszych danych testowych. Wtedy też dowiemy się o jego dokładności. Do prognozowania użyjemy funkcji predykcji (). Poniższe polecenie pomoże ci to zrobić -
preds = gnb.predict(test)
print(preds)
[1 0 0 1 1 0 0 0 1 1 1 0 1 0 1 0 1 1 1 0 1 1 0 1 1 1 1 1 1
0 1 1 1 1 1 1 0 1 0 1 1 0 1 1 1 1 1 1 1 1 0 0 1 1 1 1 1 0
0 1 1 0 0 1 1 1 0 0 1 1 0 0 1 0 1 1 1 1 1 1 0 1 1 0 0 0 0
0 1 1 1 1 1 1 1 1 0 0 1 0 0 1 0 0 1 1 1 0 1 1 0 1 1 0 0 0
1 1 1 0 0 1 1 0 1 0 0 1 1 0 0 0 1 1 1 0 1 1 0 0 1 0 1 1 0
1 0 0 1 1 1 1 1 1 1 0 0 1 1 1 1 1 1 1 1 1 1 1 1 0 1 1 1 0
1 1 0 1 1 1 1 1 1 0 0 0 1 1 0 1 0 1 1 1 1 0 1 1 0 1 1 1 0
1 0 0 1 1 1 1 1 1 1 1 0 1 1 1 1 1 0 1 0 0 1 1 0 1]Powyższe serie 0 i 1 są wartościami przewidywanymi dla klas nowotworów - złośliwych i łagodnych.
Teraz, porównując dwie tablice, a mianowicie test_labels i predsmożemy sprawdzić dokładność naszego modelu. Będziemy używaćaccuracy_score()funkcję określania dokładności. Rozważ w tym celu następujące polecenie -
from sklearn.metrics import accuracy_score
print(accuracy_score(test_labels,preds))
0.951754385965Wynik pokazuje, że klasyfikator NaïveBayes jest dokładny w 95,17%.
W ten sposób za pomocą powyższych kroków możemy zbudować nasz klasyfikator w Pythonie.
Budowanie klasyfikatora w Pythonie
W tej sekcji dowiemy się, jak zbudować klasyfikator w Pythonie.
Naiwny klasyfikator Bayesa
Naiwny Bayes to technika klasyfikacji używana do budowania klasyfikatora przy użyciu twierdzenia Bayesa. Zakłada się, że predyktory są niezależne. W prostych słowach zakłada, że obecność określonej cechy w klasie nie ma związku z obecnością jakiejkolwiek innej cechy. Aby zbudować klasyfikator Naïve Bayes, musimy użyć biblioteki Pythona o nazwie scikit learn. Istnieją trzy typy nazwanych modeli Naïve BayesGaussian, Multinomial and Bernoulli pod pakietem scikit Learn.
Aby zbudować model klasyfikatora uczenia maszynowego Naïve Bayes, potrzebujemy znaku & minus
Zestaw danych
Zamierzamy wykorzystać zbiór danych o nazwie Breast Cancer Wisconsin Diagnostic Database. Zbiór danych zawiera różne informacje o guzach raka piersi, a także etykiety klasyfikacyjnemalignant lub benign. Zbiór danych ma 569 instancji lub dane dotyczące 569 guzów i zawiera informacje o 30 atrybutach lub cechach, takich jak promień guza, tekstura, gładkość i powierzchnia. Możemy zaimportować ten zestaw danych z pakietu sklearn.
Naiwny model Bayesa
Do zbudowania klasyfikatora Naïve Bayes potrzebujemy modelu Naïve Bayes. Jak wspomniano wcześniej, istnieją trzy typy nazwanych modeli Naïve BayesGaussian, Multinomial i Bernoullipod pakietem scikit Learn. Tutaj, w poniższym przykładzie, użyjemy modelu Gaussa Naïve Bayesa.
Korzystając z powyższego, zamierzamy zbudować model uczenia maszynowego Naiwego Bayesa, aby wykorzystać informacje o guzie do przewidywania, czy guz jest złośliwy, czy łagodny.
Na początek musimy zainstalować moduł sklearn. Można to zrobić za pomocą następującego polecenia -
Import SklearnTeraz musimy zaimportować zbiór danych o nazwie Breast Cancer Wisconsin Diagnostic Database.
from sklearn.datasets import load_breast_cancerTeraz następujące polecenie załaduje zestaw danych.
data = load_breast_cancer()Dane można uporządkować w następujący sposób -
label_names = data['target_names']
labels = data['target']
feature_names = data['feature_names']
features = data['data']Teraz, aby było jaśniej, możemy wydrukować etykiety klas, etykietę pierwszej instancji danych, nasze nazwy funkcji i wartość funkcji za pomocą następujących poleceń -
print(label_names)Powyższe polecenie wypisze nazwy klas, które są odpowiednio złośliwe i łagodne. Jest to pokazane jako dane wyjściowe poniżej -
['malignant' 'benign']Teraz polecenie podane poniżej pokaże, że są one odwzorowane na wartości binarne 0 i 1. Tutaj 0 oznacza złośliwego raka, a 1 oznacza łagodnego raka. Jest to pokazane jako dane wyjściowe poniżej -
print(labels[0])
0Następujące dwa polecenia utworzą nazwy i wartości elementów.
print(feature_names[0])
mean radius
print(features[0])
[ 1.79900000e+01 1.03800000e+01 1.22800000e+02 1.00100000e+03
1.18400000e-01 2.77600000e-01 3.00100000e-01 1.47100000e-01
2.41900000e-01 7.87100000e-02 1.09500000e+00 9.05300000e-01
8.58900000e+00 1.53400000e+02 6.39900000e-03 4.90400000e-02
5.37300000e-02 1.58700000e-02 3.00300000e-02 6.19300000e-03
2.53800000e+01 1.73300000e+01 1.84600000e+02 2.01900000e+03
1.62200000e-01 6.65600000e-01 7.11900000e-01 2.65400000e-01
4.60100000e-01 1.18900000e-01]Z powyższego wyniku widać, że pierwszą instancją danych jest nowotwór złośliwy, którego główny promień wynosi 1,7990000e + 01.
Aby przetestować nasz model na niewidocznych danych, musimy podzielić nasze dane na dane treningowe i testowe. Można to zrobić za pomocą następującego kodu -
from sklearn.model_selection import train_test_splitPowyższe polecenie zaimportuje plik train_test_splitfunction ze sklearn, a poniższe polecenie podzieli dane na dane treningowe i testowe. W poniższym przykładzie wykorzystujemy 40% danych do testowania, a pozostałe dane zostaną wykorzystane do szkolenia modelu.
train, test, train_labels, test_labels =
train_test_split(features,labels,test_size = 0.40, random_state = 42)Teraz budujemy model za pomocą następujących poleceń -
from sklearn.naive_bayes import GaussianNBPowyższe polecenie zaimportuje plik GaussianNBmoduł. Teraz za pomocą polecenia podanego poniżej musimy zainicjować model.
gnb = GaussianNB()Wytrenujemy model, dopasowując go do danych przy użyciu gnb.fit().
model = gnb.fit(train, train_labels)Teraz oceń model, dokonując prognozy na danych testowych i można to zrobić w następujący sposób -
preds = gnb.predict(test)
print(preds)
[1 0 0 1 1 0 0 0 1 1 1 0 1 0 1 0 1 1 1 0 1 1 0 1 1 1 1 1 1
0 1 1 1 1 1 1 0 1 0 1 1 0 1 1 1 1 1 1 1 1 0 0 1 1 1 1 1 0
0 1 1 0 0 1 1 1 0 0 1 1 0 0 1 0 1 1 1 1 1 1 0 1 1 0 0 0 0
0 1 1 1 1 1 1 1 1 0 0 1 0 0 1 0 0 1 1 1 0 1 1 0 1 1 0 0 0
1 1 1 0 0 1 1 0 1 0 0 1 1 0 0 0 1 1 1 0 1 1 0 0 1 0 1 1 0
1 0 0 1 1 1 1 1 1 1 0 0 1 1 1 1 1 1 1 1 1 1 1 1 0 1 1 1 0
1 1 0 1 1 1 1 1 1 0 0 0 1 1 0 1 0 1 1 1 1 0 1 1 0 1 1 1 0
1 0 0 1 1 1 1 1 1 1 1 0 1 1 1 1 1 0 1 0 0 1 1 0 1]Powyższe serie zer i 1 są wartościami przewidywanymi dla klas nowotworów, tj. Złośliwych i łagodnych.
Teraz, porównując dwie tablice, a mianowicie test_labels i predsmożemy sprawdzić dokładność naszego modelu. Będziemy używaćaccuracy_score()funkcję określania dokładności. Rozważ następujące polecenie -
from sklearn.metrics import accuracy_score
print(accuracy_score(test_labels,preds))
0.951754385965Wynik pokazuje, że klasyfikator NaïveBayes jest dokładny w 95,17%.
Był to klasyfikator uczenia maszynowego oparty na modelu Naïve Bayse Gaussian.
Pomocnicze maszyny wektorowe (SVM)
Zasadniczo maszyna wektorów nośnych (SVM) to nadzorowany algorytm uczenia maszynowego, którego można używać zarówno do regresji, jak i klasyfikacji. Główną koncepcją SVM jest wykreślenie każdego elementu danych jako punktu w przestrzeni n-wymiarowej, przy czym wartość każdej cechy jest wartością określonej współrzędnej. Tutaj n byłoby cechami, które mielibyśmy. Poniżej znajduje się prosta reprezentacja graficzna, aby zrozumieć koncepcję SVM -
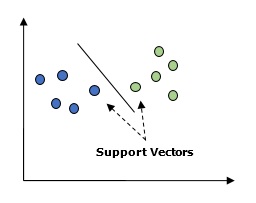
Na powyższym schemacie mamy dwie cechy. Dlatego najpierw musimy wykreślić te dwie zmienne w dwuwymiarowej przestrzeni, w której każdy punkt ma dwie współrzędne, zwane wektorami pomocniczymi. Linia dzieli dane na dwie różne sklasyfikowane grupy. Ta linia byłaby klasyfikatorem.
Tutaj mamy zamiar zbudować klasyfikator SVM przy użyciu zestawu danych scikit-learn i iris. Biblioteka Scikitlearn ma rozszerzeniesklearn.svmmodule i udostępnia sklearn.svm.svc do klasyfikacji. Klasyfikator SVM do przewidywania klasy rośliny tęczówki na podstawie 4 cech pokazano poniżej.
Zestaw danych
Użyjemy zbioru danych tęczówki, który zawiera 3 klasy po 50 instancji każda, gdzie każda klasa odnosi się do typu rośliny tęczówki. Każda instancja ma cztery cechy, a mianowicie długość działki, szerokość działki, długość i szerokość płatka. Poniżej przedstawiono klasyfikator SVM do przewidywania klasy rośliny tęczówki na podstawie 4 cech.
Jądro
Jest to technika używana przez SVM. Zasadniczo są to funkcje, które zajmują niskowymiarową przestrzeń wejściową i przekształcają ją w wyższą przestrzeń wymiarową. Przekształca nierozdzielny problem w możliwy do oddzielenia problem. Funkcją jądra może być dowolna spośród funkcji liniowej, wielomianowej, rbf i sigmoidalnej. W tym przykładzie użyjemy jądra liniowego.
Zaimportujmy teraz następujące pakiety -
import pandas as pd
import numpy as np
from sklearn import svm, datasets
import matplotlib.pyplot as pltTeraz załaduj dane wejściowe -
iris = datasets.load_iris()Bierzemy pierwsze dwie funkcje -
X = iris.data[:, :2]
y = iris.targetNa podstawie oryginalnych danych wykreślimy granice maszyny wektorów nośnych. Tworzymy siatkę do kreślenia.
x_min, x_max = X[:, 0].min() - 1, X[:, 0].max() + 1
y_min, y_max = X[:, 1].min() - 1, X[:, 1].max() + 1
h = (x_max / x_min)/100
xx, yy = np.meshgrid(np.arange(x_min, x_max, h),
np.arange(y_min, y_max, h))
X_plot = np.c_[xx.ravel(), yy.ravel()]Musimy podać wartość parametru regularyzacji.
C = 1.0Musimy stworzyć obiekt klasyfikatora SVM.
Svc_classifier = svm_classifier.SVC(kernel='linear',
C=C, decision_function_shape = 'ovr').fit(X, y)
Z = svc_classifier.predict(X_plot)
Z = Z.reshape(xx.shape)
plt.figure(figsize = (15, 5))
plt.subplot(121)
plt.contourf(xx, yy, Z, cmap = plt.cm.tab10, alpha = 0.3)
plt.scatter(X[:, 0], X[:, 1], c = y, cmap = plt.cm.Set1)
plt.xlabel('Sepal length')
plt.ylabel('Sepal width')
plt.xlim(xx.min(), xx.max())
plt.title('SVC with linear kernel')
Regresja logistyczna
Zasadniczo model regresji logistycznej jest jednym z członków rodziny algorytmów klasyfikacji nadzorowanej. Regresja logistyczna mierzy związek między zmiennymi zależnymi a zmiennymi niezależnymi, szacując prawdopodobieństwa za pomocą funkcji logistycznej.
Tutaj, jeśli mówimy o zmiennych zależnych i niezależnych, to zmienna zależna jest zmienną klasy docelowej, którą zamierzamy przewidzieć, a po drugiej stronie zmienne niezależne to cechy, których będziemy używać do przewidywania klasy docelowej.
W regresji logistycznej szacowanie prawdopodobieństw oznacza przewidywanie prawdopodobieństwa wystąpienia zdarzenia. Na przykład właściciel sklepu chciałby przewidzieć, że klient, który wszedł do sklepu, kupi (na przykład) Play Station lub nie. Byłoby wiele cech klienta - płeć, wiek itp., Które byłyby obserwowane przez sklepikarza, aby przewidzieć prawdopodobieństwo wystąpienia, tj. Zakup play station lub nie. Funkcja logistyczna to krzywa sigmoidalna, która służy do budowania funkcji o różnych parametrach.
Wymagania wstępne
Przed zbudowaniem klasyfikatora wykorzystującego regresję logistyczną musimy zainstalować pakiet Tkinter w naszym systemie. Można go zainstalować zhttps://docs.python.org/2/library/tkinter.html.
Teraz za pomocą kodu podanego poniżej możemy stworzyć klasyfikator wykorzystujący regresję logistyczną -
Najpierw zaimportujemy kilka paczek -
import numpy as np
from sklearn import linear_model
import matplotlib.pyplot as pltTeraz musimy zdefiniować przykładowe dane, które można wykonać w następujący sposób -
X = np.array([[2, 4.8], [2.9, 4.7], [2.5, 5], [3.2, 5.5], [6, 5], [7.6, 4],
[3.2, 0.9], [2.9, 1.9],[2.4, 3.5], [0.5, 3.4], [1, 4], [0.9, 5.9]])
y = np.array([0, 0, 0, 1, 1, 1, 2, 2, 2, 3, 3, 3])Następnie musimy utworzyć klasyfikator regresji logistycznej, który można wykonać w następujący sposób -
Classifier_LR = linear_model.LogisticRegression(solver = 'liblinear', C = 75)Wreszcie, musimy wyszkolić ten klasyfikator -
Classifier_LR.fit(X, y)Teraz, jak możemy wizualizować wynik? Można to zrobić, tworząc funkcję o nazwie Logistic_visualize () -
Def Logistic_visualize(Classifier_LR, X, y):
min_x, max_x = X[:, 0].min() - 1.0, X[:, 0].max() + 1.0
min_y, max_y = X[:, 1].min() - 1.0, X[:, 1].max() + 1.0W powyższym wierszu zdefiniowaliśmy minimalne i maksymalne wartości X i Y, które mają być użyte w siatce. Ponadto zdefiniujemy rozmiar kroku do wykreślenia siatki siatki.
mesh_step_size = 0.02Zdefiniujmy siatkę wartości X i Y w następujący sposób -
x_vals, y_vals = np.meshgrid(np.arange(min_x, max_x, mesh_step_size),
np.arange(min_y, max_y, mesh_step_size))Za pomocą poniższego kodu możemy uruchomić klasyfikator na siatce mesh -
output = classifier.predict(np.c_[x_vals.ravel(), y_vals.ravel()])
output = output.reshape(x_vals.shape)
plt.figure()
plt.pcolormesh(x_vals, y_vals, output, cmap = plt.cm.gray)
plt.scatter(X[:, 0], X[:, 1], c = y, s = 75, edgecolors = 'black',
linewidth=1, cmap = plt.cm.Paired)Poniższy wiersz kodu określi granice działki
plt.xlim(x_vals.min(), x_vals.max())
plt.ylim(y_vals.min(), y_vals.max())
plt.xticks((np.arange(int(X[:, 0].min() - 1), int(X[:, 0].max() + 1), 1.0)))
plt.yticks((np.arange(int(X[:, 1].min() - 1), int(X[:, 1].max() + 1), 1.0)))
plt.show()Teraz po uruchomieniu kodu otrzymamy następujący wynik, klasyfikator regresji logistycznej -

Klasyfikator drzewa decyzyjnego
Drzewo decyzyjne to w zasadzie schemat blokowy drzewa binarnego, w którym każdy węzeł dzieli grupę obserwacji zgodnie z pewną zmienną cech.
Tutaj tworzymy klasyfikator drzewa decyzyjnego do przewidywania płci męskiej lub żeńskiej. Weźmiemy bardzo mały zestaw danych zawierający 19 próbek. Próbki te miałyby dwie cechy - „wysokość” i „długość włosów”.
Warunek wstępny
Aby zbudować następujący klasyfikator, musimy zainstalować pydotplus i graphviz. Zasadniczo graphviz to narzędzie do rysowania grafiki za pomocą plików dot ipydotplusto moduł języka Dot Graphviz. Można go zainstalować za pomocą menedżera pakietów lub pip.
Teraz możemy zbudować klasyfikator drzewa decyzyjnego za pomocą następującego kodu Pythona -
Na początek zaimportujmy kilka ważnych bibliotek w następujący sposób -
import pydotplus
from sklearn import tree
from sklearn.datasets import load_iris
from sklearn.metrics import classification_report
from sklearn import cross_validation
import collectionsTeraz musimy dostarczyć zestaw danych w następujący sposób -
X = [[165,19],[175,32],[136,35],[174,65],[141,28],[176,15],[131,32],
[166,6],[128,32],[179,10],[136,34],[186,2],[126,25],[176,28],[112,38],
[169,9],[171,36],[116,25],[196,25]]
Y = ['Man','Woman','Woman','Man','Woman','Man','Woman','Man','Woman',
'Man','Woman','Man','Woman','Woman','Woman','Man','Woman','Woman','Man']
data_feature_names = ['height','length of hair']
X_train, X_test, Y_train, Y_test = cross_validation.train_test_split
(X, Y, test_size=0.40, random_state=5)Po dostarczeniu zbioru danych musimy dopasować model, co można zrobić w następujący sposób -
clf = tree.DecisionTreeClassifier()
clf = clf.fit(X,Y)Przewidywanie można wykonać za pomocą następującego kodu Pythona -
prediction = clf.predict([[133,37]])
print(prediction)Możemy zwizualizować drzewo decyzyjne za pomocą następującego kodu Pythona -
dot_data = tree.export_graphviz(clf,feature_names = data_feature_names,
out_file = None,filled = True,rounded = True)
graph = pydotplus.graph_from_dot_data(dot_data)
colors = ('orange', 'yellow')
edges = collections.defaultdict(list)
for edge in graph.get_edge_list():
edges[edge.get_source()].append(int(edge.get_destination()))
for edge in edges: edges[edge].sort()
for i in range(2):dest = graph.get_node(str(edges[edge][i]))[0]
dest.set_fillcolor(colors[i])
graph.write_png('Decisiontree16.png')Poda prognozę dla powyższego kodu jako [‘Woman’] i utwórz następujące drzewo decyzyjne -

Możemy zmienić wartości cech w prognozowaniu, aby to przetestować.
Losowy klasyfikator lasu
Jak wiemy, metody zespołowe to metody, które łączą modele uczenia maszynowego w potężniejszy model uczenia maszynowego. Jednym z nich jest Random Forest, zbiór drzew decyzyjnych. Jest lepsze niż pojedyncze drzewo decyzyjne, ponieważ zachowując moc predykcyjną, może zmniejszyć nadmierne dopasowanie poprzez uśrednienie wyników. Tutaj zamierzamy zaimplementować model losowego lasu na zbiorze danych SCIKIT Learn o rakach.
Importuj niezbędne pakiety -
from sklearn.ensemble import RandomForestClassifier
from sklearn.model_selection import train_test_split
from sklearn.datasets import load_breast_cancer
cancer = load_breast_cancer()
import matplotlib.pyplot as plt
import numpy as npTeraz musimy podać zestaw danych, który można wykonać w następujący sposób & minus
cancer = load_breast_cancer()
X_train, X_test, y_train,
y_test = train_test_split(cancer.data, cancer.target, random_state = 0)Po dostarczeniu zbioru danych musimy dopasować model, co można zrobić w następujący sposób -
forest = RandomForestClassifier(n_estimators = 50, random_state = 0)
forest.fit(X_train,y_train)Teraz uzyskaj dokładność zarówno na uczeniu, jak i testowaniu podzbioru: jeśli wtedy zwiększymy liczbę estymatorów, dokładność podzbioru testowego również wzrośnie.
print('Accuracy on the training subset:(:.3f)',format(forest.score(X_train,y_train)))
print('Accuracy on the training subset:(:.3f)',format(forest.score(X_test,y_test)))Wynik
Accuracy on the training subset:(:.3f) 1.0
Accuracy on the training subset:(:.3f) 0.965034965034965Teraz, podobnie jak drzewo decyzyjne, losowy las ma feature_importancemoduł, który zapewni lepszy widok wagi cech niż drzewo decyzyjne. Można go wykreślić i wizualizować w następujący sposób -
n_features = cancer.data.shape[1]
plt.barh(range(n_features),forest.feature_importances_, align='center')
plt.yticks(np.arange(n_features),cancer.feature_names)
plt.xlabel('Feature Importance')
plt.ylabel('Feature')
plt.show()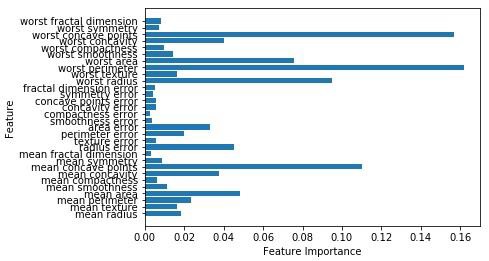
Wydajność klasyfikatora
Po wdrożeniu algorytmu uczenia maszynowego musimy dowiedzieć się, jak efektywny jest ten model. Kryteria pomiaru skuteczności mogą opierać się na zbiorach danych i miernikach. Do oceny różnych algorytmów uczenia maszynowego możemy użyć różnych wskaźników wydajności. Na przykład załóżmy, że jeśli klasyfikator jest używany do rozróżniania obrazów różnych obiektów, możemy użyć wskaźników wydajności klasyfikacji, takich jak średnia dokładność, AUC itp. W pewnym sensie metryka, którą wybieramy do oceny naszego modelu uczenia maszynowego, to bardzo ważne, ponieważ wybór metryk wpływa na sposób mierzenia i porównywania wydajności algorytmu uczenia maszynowego. Oto niektóre dane -
Macierz zamieszania
Zasadniczo jest używany do problemu klasyfikacji, w którym wynik może składać się z dwóch lub więcej typów klas. Jest to najłatwiejszy sposób pomiaru wydajności klasyfikatora. Macierz pomyłki to w zasadzie tabela z dwoma wymiarami: „Rzeczywiste” i „Przewidywane”. Oba wymiary mają „Prawdziwie pozytywne (TP)”, „Prawdziwie negatywne (TN)”, „Fałszywie dodatnie (FP)”, „Fałszywie negatywne (FN)”.
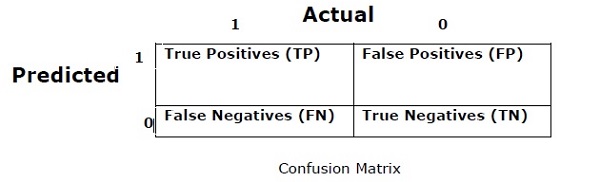
W powyższej macierzy nieporozumień 1 oznacza klasę pozytywną, a 0 - klasę negatywną.
Poniżej znajdują się terminy związane z macierzą nieporozumień -
True Positives − TP to przypadki, w których aktualna klasa punktu danych wynosiła 1, a przewidywana również 1.
True Negatives − TN to przypadki, w których aktualna klasa punktu danych wynosiła 0, a wartość przewidywana również wynosi 0.
False Positives − FP to przypadki, w których aktualna klasa punktu danych wynosiła 0, a przewidywana również 1.
False Negatives − FN to przypadki, w których aktualna klasa punktu danych wynosiła 1, a prognoza również wynosiła 0.
Precyzja
Sama macierz pomyłki nie jest miarą wydajności jako taka, ale prawie wszystkie macierze wydajności są oparte na macierzy pomyłki. Jednym z nich jest dokładność. W problemach klasyfikacyjnych można go zdefiniować jako liczbę poprawnych przewidywań wykonanych przez model dla wszystkich rodzajów prognoz. Wzór na obliczenie dokładności jest następujący -
$$ Dokładność = \ frac {TP + TN} {TP + FP + FN + TN} $$
Precyzja
Jest używany głównie do pobierania dokumentów. Można zdefiniować, ile zwróconych dokumentów jest poprawnych. Poniżej znajduje się wzór na obliczanie dokładności -
$$ Precision = \ frac {TP} {TP + FP} $$
Przypomnienie lub wrażliwość
Można to zdefiniować jako liczbę pozytywów zwracanych przez model. Poniżej znajduje się wzór na obliczanie częstotliwości odtwarzania / czułości modelu -
$$ Recall = \ frac {TP} {TP + FN} $$
Specyficzność
Można to zdefiniować jako liczbę negatywów zwracanych przez model. Przypominanie jest dokładnie odwrotne. Poniżej znajduje się wzór na obliczenie specyfiki modelu -
$$ Specyfika = \ frac {TN} {TN + FP} $$
Problem nierównowagi klas
Nierównowaga klas to scenariusz, w którym liczba obserwacji należących do jednej klasy jest znacznie niższa niż obserwacji należących do innych klas. Na przykład ten problem jest widoczny w scenariuszu, w którym musimy zidentyfikować rzadkie choroby, oszukańcze transakcje w banku itp.
Przykład niezrównoważonych klas
Rozważmy przykład zbioru danych dotyczących wykrywania oszustw, aby zrozumieć koncepcję niezrównoważonej klasy -
Total observations = 5000
Fraudulent Observations = 50
Non-Fraudulent Observations = 4950
Event Rate = 1%Rozwiązanie
Balancing the classes’działa jako rozwiązanie dla niezrównoważonych klas. Głównym celem zbilansowania klas jest zwiększenie częstotliwości klasy mniejszości lub zmniejszenie częstotliwości klasy większościowej. Poniżej przedstawiono podejścia do rozwiązania problemu klas nierównowagi -
Ponowne pobieranie próbek
Ponowne próbkowanie to seria metod używanych do rekonstrukcji przykładowych zbiorów danych - zarówno zbiorów uczących, jak i testowych. Ponowne próbkowanie ma na celu poprawę dokładności modelu. Oto kilka technik ponownego pobierania próbek -
Random Under-Sampling- Technika ta ma na celu zrównoważenie dystrybucji klas poprzez losowe eliminowanie przykładów klas większości. Dzieje się tak, dopóki instancje klas większości i mniejszości nie zostaną zrównoważone.
Total observations = 5000
Fraudulent Observations = 50
Non-Fraudulent Observations = 4950
Event Rate = 1%W tym przypadku pobieramy 10% próbek bez wymiany z przypadków, w których nie występuje oszustwo, a następnie łączymy je z przypadkami oszustwa -
Obserwacje niezwiązane z oszustwami po losowym doborze próby = 10% z 4950 = 495
Suma obserwacji po połączeniu ich z fałszywymi obserwacjami = 50 + 495 = 545
W związku z tym odsetek zdarzeń dla nowego zbioru danych po pobraniu próbki = 9%
Główną zaletą tej techniki jest to, że może skrócić czas działania i poprawić pamięć. Ale z drugiej strony może odrzucić przydatne informacje, jednocześnie zmniejszając liczbę próbek danych uczących.
Random Over-Sampling - Technika ta ma na celu zrównoważenie dystrybucji klas poprzez zwiększenie liczby instancji w klasie mniejszościowej poprzez ich replikację.
Total observations = 5000
Fraudulent Observations = 50
Non-Fraudulent Observations = 4950
Event Rate = 1%W przypadku, gdy replikujemy 50 fałszywych obserwacji 30 razy, to fałszywe obserwacje po replikowaniu obserwacji klasy mniejszości będą wynosić 1500. A następnie całkowita liczba obserwacji w nowych danych po nadpróbkowaniu wyniesie 4950 + 1500 = 6450. Stąd częstość zdarzeń dla nowego zestawu danych byłoby 1500/6450 = 23%.
Główną zaletą tej metody jest brak utraty przydatnych informacji. Ale z drugiej strony ma zwiększone szanse na przesadzenie, ponieważ powiela wydarzenia klas mniejszościowych.
Techniki zespołowe
Ta metodologia zasadniczo służy do modyfikowania istniejących algorytmów klasyfikacji, aby były odpowiednie dla niezrównoważonych zbiorów danych. W tym podejściu konstruujemy kilka dwustopniowych klasyfikatorów z oryginalnych danych, a następnie agregujemy ich przewidywania. Losowy klasyfikator lasu jest przykładem klasyfikatora opartego na zespołach.