R - Analisi di sopravvivenza
L'analisi di sopravvivenza si occupa di prevedere il momento in cui si verificherà un evento specifico. È anche noto come analisi del tempo di guasto o analisi del tempo fino alla morte. Ad esempio, prevedere il numero di giorni in cui una persona malata di cancro sopravviverà o prevedere il tempo in cui un sistema meccanico fallirà.
Il pacchetto R denominato survivalviene utilizzato per eseguire analisi di sopravvivenza. Questo pacchetto contiene la funzioneSurv()che prende i dati di input come una formula R e crea un oggetto di sopravvivenza tra le variabili scelte per l'analisi. Quindi usiamo la funzionesurvfit() per creare un grafico per l'analisi.
Installa pacchetto
install.packages("survival")Sintassi
La sintassi di base per creare analisi di sopravvivenza in R è:
Surv(time,event)
survfit(formula)Di seguito la descrizione dei parametri utilizzati:
time è il tempo di follow-up fino al verificarsi dell'evento.
event indica lo stato di accadimento dell'evento atteso.
formula è la relazione tra le variabili predittive.
Esempio
Considereremo il set di dati denominato "pbc" presente nei pacchetti di sopravvivenza installati sopra. Descrive i punti di dati di sopravvivenza sulle persone affette da cirrosi biliare primitiva (PBC) del fegato. Tra le tante colonne presenti nel data set ci occupiamo principalmente dei campi "time" e "status". Il tempo rappresenta il numero di giorni tra la registrazione del paziente e l'inizio dell'evento tra il paziente che riceve un trapianto di fegato o la morte del paziente.
# Load the library.
library("survival")
# Print first few rows.
print(head(pbc))Quando eseguiamo il codice sopra, produce il seguente risultato e grafico:
id time status trt age sex ascites hepato spiders edema bili chol
1 1 400 2 1 58.76523 f 1 1 1 1.0 14.5 261
2 2 4500 0 1 56.44627 f 0 1 1 0.0 1.1 302
3 3 1012 2 1 70.07255 m 0 0 0 0.5 1.4 176
4 4 1925 2 1 54.74059 f 0 1 1 0.5 1.8 244
5 5 1504 1 2 38.10541 f 0 1 1 0.0 3.4 279
6 6 2503 2 2 66.25873 f 0 1 0 0.0 0.8 248
albumin copper alk.phos ast trig platelet protime stage
1 2.60 156 1718.0 137.95 172 190 12.2 4
2 4.14 54 7394.8 113.52 88 221 10.6 3
3 3.48 210 516.0 96.10 55 151 12.0 4
4 2.54 64 6121.8 60.63 92 183 10.3 4
5 3.53 143 671.0 113.15 72 136 10.9 3
6 3.98 50 944.0 93.00 63 NA 11.0 3Dai dati di cui sopra stiamo considerando tempo e stato per la nostra analisi.
Applicazione delle funzioni Surv () e survfit ()
Ora procediamo ad applicare il Surv() funzione al set di dati sopra e creare un grafico che mostrerà la tendenza.
# Load the library.
library("survival")
# Create the survival object.
survfit(Surv(pbc$time,pbc$status == 2)~1)
# Give the chart file a name.
png(file = "survival.png")
# Plot the graph.
plot(survfit(Surv(pbc$time,pbc$status == 2)~1))
# Save the file.
dev.off()Quando eseguiamo il codice sopra, produce il seguente risultato e grafico:
Call: survfit(formula = Surv(pbc$time, pbc$status == 2) ~ 1)
n events median 0.95LCL 0.95UCL
418 161 3395 3090 3853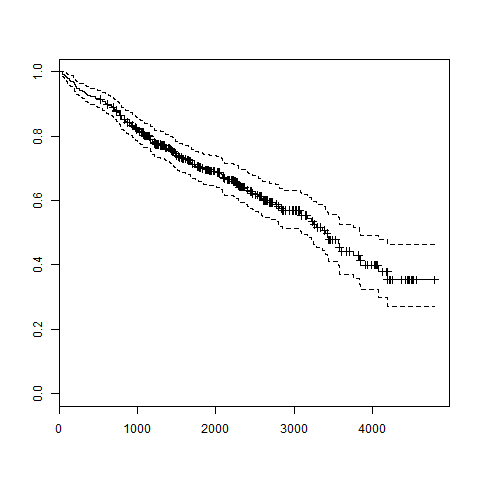
L'andamento nel grafico sopra ci aiuta a prevedere la probabilità di sopravvivenza alla fine di un certo numero di giorni.