Python을 사용한 AI –지도 학습 : 분류
이 장에서는지도 학습 (분류) 구현에 중점을 둘 것입니다.
분류 기술 또는 모델은 관찰 된 값에서 몇 가지 결론을 얻으려고합니다. 분류 문제에서 우리는“Black”또는“white”또는“Teaching”및“Non-Teaching”과 같은 분류 된 출력을 가지고 있습니다. 분류 모델을 구축하는 동안 데이터 포인트와 해당 레이블이 포함 된 훈련 데이터 세트가 필요합니다. 예를 들어 이미지가 자동차인지 여부를 확인하려는 경우입니다. 이를 확인하기 위해 "자동차"와 "자동차 없음"과 관련된 두 가지 클래스가있는 학습 데이터 세트를 작성합니다. 그런 다음 훈련 샘플을 사용하여 모델을 훈련해야합니다. 분류 모델은 주로 얼굴 인식, 스팸 식별 등에 사용됩니다.
Python에서 분류자를 빌드하는 단계
Python으로 분류기를 구축하기 위해 Python 3과 머신 러닝 도구 인 Scikit-learn을 사용할 것입니다. 다음 단계에 따라 Python으로 분류자를 작성하십시오.
1 단계-Scikit-learn 가져 오기
이것은 파이썬에서 분류자를 만드는 첫 단계가 될 것입니다. 이 단계에서는 Python에서 최고의 기계 학습 모듈 중 하나 인 Scikit-learn이라는 Python 패키지를 설치합니다. 다음 명령은 패키지를 가져 오는 데 도움이됩니다.
Import Sklearn2 단계-Scikit-learn의 데이터 세트 가져 오기
이 단계에서는 기계 학습 모델에 대한 데이터 세트 작업을 시작할 수 있습니다. 여기에서 우리는the 유방암 위스콘신 진단 데이터베이스. 데이터 세트에는 유방암 종양에 대한 다양한 정보와 분류 라벨이 포함됩니다.malignant 또는 benign. 데이터 세트에는 569 개의 종양에 대한 569 개의 인스턴스 또는 데이터가 있으며 종양의 반경, 질감, 부드러움 및 면적과 같은 30 가지 속성 또는 특징에 대한 정보가 포함됩니다. 다음 명령을 사용하여 Scikit-learn의 유방암 데이터 세트를 가져올 수 있습니다.
from sklearn.datasets import load_breast_cancer이제 다음 명령이 데이터 세트를로드합니다.
data = load_breast_cancer()다음은 중요한 사전 키 목록입니다.
- 분류 라벨 이름 (target_names)
- 실제 라벨 (대상)
- 속성 / 기능 이름 (feature_names)
- 속성 (데이터)
이제 다음 명령을 사용하여 중요한 각 정보 집합에 대한 새 변수를 만들고 데이터를 할당 할 수 있습니다. 즉, 다음 명령으로 데이터를 구성 할 수 있습니다.
label_names = data['target_names']
labels = data['target']
feature_names = data['feature_names']
features = data['data']이제 더 명확하게하기 위해 다음 명령을 사용하여 클래스 레이블, 첫 번째 데이터 인스턴스의 레이블, 기능 이름 및 기능 값을 인쇄 할 수 있습니다.
print(label_names)위의 명령은 각각 악성 및 양성인 클래스 이름을 인쇄합니다. 다음과 같이 출력됩니다.
['malignant' 'benign']이제 아래 명령은 이진 값 0과 1에 매핑되었음을 보여줍니다. 여기서 0은 악성 암을 나타내고 1은 양성 암을 나타냅니다. 다음과 같은 출력을 받게됩니다.
print(labels[0])
0아래에 주어진 두 명령은 기능 이름과 기능 값을 생성합니다.
print(feature_names[0])
mean radius
print(features[0])
[ 1.79900000e+01 1.03800000e+01 1.22800000e+02 1.00100000e+03
1.18400000e-01 2.77600000e-01 3.00100000e-01 1.47100000e-01
2.41900000e-01 7.87100000e-02 1.09500000e+00 9.05300000e-01
8.58900000e+00 1.53400000e+02 6.39900000e-03 4.90400000e-02
5.37300000e-02 1.58700000e-02 3.00300000e-02 6.19300000e-03
2.53800000e+01 1.73300000e+01 1.84600000e+02 2.01900000e+03
1.62200000e-01 6.65600000e-01 7.11900000e-01 2.65400000e-01
4.60100000e-01 1.18900000e-01]위의 출력에서 첫 번째 데이터 인스턴스는 반경이 1.7990000e + 01 인 악성 종양임을 알 수 있습니다.
3 단계-데이터를 세트로 구성
이 단계에서는 데이터를 훈련 세트와 테스트 세트라는 두 부분으로 나눕니다. 보이지 않는 데이터에 대해 모델을 테스트해야하기 때문에 데이터를 이러한 세트로 분할하는 것은 매우 중요합니다. 데이터를 세트로 분할하기 위해 sklearn에는train_test_split()함수. 다음 명령을 사용하여 이러한 세트의 데이터를 분할 할 수 있습니다.
from sklearn.model_selection import train_test_split위의 명령은 train_test_splitsklearn의 함수와 아래 명령은 데이터를 훈련 및 테스트 데이터로 분할합니다. 아래 주어진 예에서는 데이터의 40 %를 테스트에 사용하고 나머지 데이터는 모델 학습에 사용합니다.
train, test, train_labels, test_labels = train_test_split(features,labels,test_size = 0.40, random_state = 42)4 단계-모델 구축
이 단계에서는 모델을 빌드합니다. 모델 구축을 위해 Naïve Bayes 알고리즘을 사용할 것입니다. 다음 명령을 사용하여 모델을 구축 할 수 있습니다.
from sklearn.naive_bayes import GaussianNB위의 명령은 GaussianNB 모듈을 가져옵니다. 이제 다음 명령을 사용하여 모델을 초기화 할 수 있습니다.
gnb = GaussianNB()gnb.fit ()을 사용하여 모델을 데이터에 피팅하여 훈련시킬 것입니다.
model = gnb.fit(train, train_labels)5 단계-모델과 그 정확성 평가
이 단계에서는 테스트 데이터를 예측하여 모델을 평가할 것입니다. 그런 다음 그 정확성도 알아낼 것입니다. 예측을 위해 predict () 함수를 사용합니다. 다음 명령은이를 수행하는 데 도움이됩니다.
preds = gnb.predict(test)
print(preds)
[1 0 0 1 1 0 0 0 1 1 1 0 1 0 1 0 1 1 1 0 1 1 0 1 1 1 1 1 1
0 1 1 1 1 1 1 0 1 0 1 1 0 1 1 1 1 1 1 1 1 0 0 1 1 1 1 1 0
0 1 1 0 0 1 1 1 0 0 1 1 0 0 1 0 1 1 1 1 1 1 0 1 1 0 0 0 0
0 1 1 1 1 1 1 1 1 0 0 1 0 0 1 0 0 1 1 1 0 1 1 0 1 1 0 0 0
1 1 1 0 0 1 1 0 1 0 0 1 1 0 0 0 1 1 1 0 1 1 0 0 1 0 1 1 0
1 0 0 1 1 1 1 1 1 1 0 0 1 1 1 1 1 1 1 1 1 1 1 1 0 1 1 1 0
1 1 0 1 1 1 1 1 1 0 0 0 1 1 0 1 0 1 1 1 1 0 1 1 0 1 1 1 0
1 0 0 1 1 1 1 1 1 1 1 0 1 1 1 1 1 0 1 0 0 1 1 0 1]위의 일련의 0과 1은 종양 등급 (악성 및 양성)에 대한 예측 값입니다.
이제 두 배열을 비교하여 test_labels 과 preds, 모델의 정확성을 확인할 수 있습니다. 우리는accuracy_score()정확도를 결정하는 기능. 이에 대한 다음 명령을 고려하십시오-
from sklearn.metrics import accuracy_score
print(accuracy_score(test_labels,preds))
0.951754385965결과는 NaïveBayes 분류 기가 95.17 % 정확하다는 것을 보여줍니다.
이런 식으로 위의 단계를 통해 Python으로 분류자를 빌드 할 수 있습니다.
파이썬으로 분류기 만들기
이 섹션에서는 Python으로 분류자를 빌드하는 방법을 배웁니다.
나이브 베이 즈 분류기
Naïve Bayes는 Bayes 정리를 사용하여 분류기를 구축하는 데 사용되는 분류 기술입니다. 예측 변수가 독립적이라는 가정이 있습니다. 간단히 말해서, 클래스에있는 특정 기능의 존재가 다른 기능의 존재와 관련이 없다고 가정합니다. Naïve Bayes 분류기를 빌드하려면 scikit learn이라는 Python 라이브러리를 사용해야합니다. Naïve Bayes 모델에는 다음과 같은 세 가지 유형이 있습니다.Gaussian, Multinomial and Bernoulli scikit 학습 패키지에서.
Naïve Bayes 기계 학습 분류기 모델을 구축하려면 다음이 필요합니다.
데이터 세트
Breast Cancer Wisconsin Diagnostic Database 라는 데이터 세트를 사용할 것 입니다. 데이터 세트에는 유방암 종양에 대한 다양한 정보와 분류 라벨이 포함됩니다.malignant 또는 benign. 데이터 세트에는 569 개의 종양에 대한 569 개의 인스턴스 또는 데이터가 있으며 종양의 반경, 질감, 부드러움 및 면적과 같은 30 가지 속성 또는 특징에 대한 정보가 포함됩니다. sklearn 패키지에서이 데이터 세트를 가져올 수 있습니다.
나이브 베이 즈 모델
Naïve Bayes 분류기를 구축하려면 Naïve Bayes 모델이 필요합니다. 앞서 말했듯이 이름이 지정된 Naïve Bayes 모델에는 세 가지 유형이 있습니다.Gaussian, Multinomial 과 Bernoulliscikit 학습 패키지에서. 다음 예제에서는 Gaussian Naïve Bayes 모델을 사용합니다.
위의 내용을 사용하여 종양 정보를 사용하여 종양이 악성인지 양성인지 예측하는 Naïve Bayes 기계 학습 모델을 구축 할 것입니다.
우선 sklearn 모듈을 설치해야합니다. 다음 명령을 사용하여 수행 할 수 있습니다.
Import Sklearn이제 Breast Cancer Wisconsin Diagnostic Database라는 데이터 세트를 가져와야합니다.
from sklearn.datasets import load_breast_cancer이제 다음 명령이 데이터 세트를로드합니다.
data = load_breast_cancer()데이터는 다음과 같이 구성 할 수 있습니다.
label_names = data['target_names']
labels = data['target']
feature_names = data['feature_names']
features = data['data']이제 더 명확하게하기 위해 다음 명령을 사용하여 클래스 레이블, 첫 번째 데이터 인스턴스의 레이블, 기능 이름 및 기능 값을 인쇄 할 수 있습니다.
print(label_names)위의 명령은 각각 악성 및 양성인 클래스 이름을 인쇄합니다. 다음과 같이 출력됩니다.
['malignant' 'benign']이제 아래의 명령은 이진 값 0과 1에 매핑되어 있음을 보여줍니다. 여기서 0은 악성 암을 나타내고 1은 양성 암을 나타냅니다. 다음과 같이 출력됩니다.
print(labels[0])
0다음 두 명령은 기능 이름과 기능 값을 생성합니다.
print(feature_names[0])
mean radius
print(features[0])
[ 1.79900000e+01 1.03800000e+01 1.22800000e+02 1.00100000e+03
1.18400000e-01 2.77600000e-01 3.00100000e-01 1.47100000e-01
2.41900000e-01 7.87100000e-02 1.09500000e+00 9.05300000e-01
8.58900000e+00 1.53400000e+02 6.39900000e-03 4.90400000e-02
5.37300000e-02 1.58700000e-02 3.00300000e-02 6.19300000e-03
2.53800000e+01 1.73300000e+01 1.84600000e+02 2.01900000e+03
1.62200000e-01 6.65600000e-01 7.11900000e-01 2.65400000e-01
4.60100000e-01 1.18900000e-01]위의 출력에서 첫 번째 데이터 인스턴스가 주요 반경이 1.7990000e + 01 인 악성 종양임을 알 수 있습니다.
보이지 않는 데이터에서 모델을 테스트하려면 데이터를 훈련 및 테스트 데이터로 분할해야합니다. 다음 코드를 사용하여 수행 할 수 있습니다.
from sklearn.model_selection import train_test_split위의 명령은 train_test_splitsklearn의 함수와 아래 명령은 데이터를 훈련 및 테스트 데이터로 분할합니다. 아래 예에서는 데이터의 40 %를 테스트에 사용하고 리 마이닝 데이터는 모델 학습에 사용됩니다.
train, test, train_labels, test_labels =
train_test_split(features,labels,test_size = 0.40, random_state = 42)이제 다음 명령으로 모델을 구축하고 있습니다.
from sklearn.naive_bayes import GaussianNB위의 명령은 GaussianNB기준 치수. 이제 아래의 명령으로 모델을 초기화해야합니다.
gnb = GaussianNB()다음을 사용하여 모델을 데이터에 피팅하여 훈련시킬 것입니다. gnb.fit().
model = gnb.fit(train, train_labels)이제 테스트 데이터를 예측하여 모델을 평가하면 다음과 같이 수행 할 수 있습니다.
preds = gnb.predict(test)
print(preds)
[1 0 0 1 1 0 0 0 1 1 1 0 1 0 1 0 1 1 1 0 1 1 0 1 1 1 1 1 1
0 1 1 1 1 1 1 0 1 0 1 1 0 1 1 1 1 1 1 1 1 0 0 1 1 1 1 1 0
0 1 1 0 0 1 1 1 0 0 1 1 0 0 1 0 1 1 1 1 1 1 0 1 1 0 0 0 0
0 1 1 1 1 1 1 1 1 0 0 1 0 0 1 0 0 1 1 1 0 1 1 0 1 1 0 0 0
1 1 1 0 0 1 1 0 1 0 0 1 1 0 0 0 1 1 1 0 1 1 0 0 1 0 1 1 0
1 0 0 1 1 1 1 1 1 1 0 0 1 1 1 1 1 1 1 1 1 1 1 1 0 1 1 1 0
1 1 0 1 1 1 1 1 1 0 0 0 1 1 0 1 0 1 1 1 1 0 1 1 0 1 1 1 0
1 0 0 1 1 1 1 1 1 1 1 0 1 1 1 1 1 0 1 0 0 1 1 0 1]위의 일련의 0과 1은 종양 클래스 즉 악성 및 양성에 대한 예측 값입니다.
이제 두 배열을 비교하여 test_labels 과 preds, 모델의 정확성을 확인할 수 있습니다. 우리는accuracy_score()정확도를 결정하는 기능. 다음 명령을 고려하십시오-
from sklearn.metrics import accuracy_score
print(accuracy_score(test_labels,preds))
0.951754385965결과는 NaïveBayes 분류 기가 95.17 % 정확하다는 것을 보여줍니다.
그것은 Naïve Bayse Gaussian 모델을 기반으로 한 기계 학습 분류기였습니다.
서포트 벡터 머신 (SVM)
기본적으로 SVM (Support Vector Machine)은 회귀 및 분류 모두에 사용할 수있는 감독되는 기계 학습 알고리즘입니다. SVM의 주요 개념은 각 데이터 항목을 특정 좌표의 값이되는 각 특성의 값을 사용하여 n 차원 공간의 점으로 플로팅하는 것입니다. 여기서 n은 우리가 가질 수있는 기능입니다. 다음은 SVM의 개념을 이해하기위한 간단한 그래픽 표현입니다.
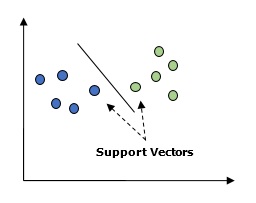
위의 다이어그램에는 두 가지 기능이 있습니다. 따라서 먼저이 두 변수를 각 점에 지원 벡터라고하는 두 개의 좌표가있는 2 차원 공간에 플로팅해야합니다. 선은 데이터를 두 개의 다른 분류 그룹으로 분할합니다. 이 라인이 분류 자입니다.
여기에서는 scikit-learn 및 iris 데이터 세트를 사용하여 SVM 분류기를 구축 할 것입니다. Scikitlearn 라이브러리에는sklearn.svm모듈 및 분류를 위해 sklearn.svm.svc를 제공합니다. 다음은 4 가지 특징을 기반으로 붓꽃의 등급을 예측하는 SVM 분류기입니다.
데이터 세트
각각 50 개 인스턴스의 3 개 클래스가 포함 된 홍채 데이터 세트를 사용합니다. 여기서 각 클래스는 붓꽃 종류를 나타냅니다. 각 인스턴스에는 꽃받침 길이, 꽃받침 너비, 꽃잎 길이 및 꽃잎 너비의 네 가지 기능이 있습니다. 다음은 4 가지 특징을 기반으로 붓꽃의 등급을 예측하는 SVM 분류기입니다.
핵심
SVM에서 사용하는 기술입니다. 기본적으로 이것들은 저 차원 입력 공간을 가져다가 고차원 공간으로 변환하는 기능입니다. 분리 불가능한 문제를 분리 가능한 문제로 변환합니다. 커널 함수는 선형, 다항식, rbf 및 시그 모이 드 중 하나 일 수 있습니다. 이 예에서는 선형 커널을 사용합니다.
이제 다음 패키지를 가져 오겠습니다.
import pandas as pd
import numpy as np
from sklearn import svm, datasets
import matplotlib.pyplot as plt이제 입력 데이터를로드합니다.
iris = datasets.load_iris()우리는 처음 두 가지 기능을 사용합니다.
X = iris.data[:, :2]
y = iris.target원본 데이터로 서포트 벡터 머신 경계를 플로팅합니다. 플롯 할 메시를 만들고 있습니다.
x_min, x_max = X[:, 0].min() - 1, X[:, 0].max() + 1
y_min, y_max = X[:, 1].min() - 1, X[:, 1].max() + 1
h = (x_max / x_min)/100
xx, yy = np.meshgrid(np.arange(x_min, x_max, h),
np.arange(y_min, y_max, h))
X_plot = np.c_[xx.ravel(), yy.ravel()]정규화 매개 변수의 값을 제공해야합니다.
C = 1.0SVM 분류기 객체를 만들어야합니다.
Svc_classifier = svm_classifier.SVC(kernel='linear',
C=C, decision_function_shape = 'ovr').fit(X, y)
Z = svc_classifier.predict(X_plot)
Z = Z.reshape(xx.shape)
plt.figure(figsize = (15, 5))
plt.subplot(121)
plt.contourf(xx, yy, Z, cmap = plt.cm.tab10, alpha = 0.3)
plt.scatter(X[:, 0], X[:, 1], c = y, cmap = plt.cm.Set1)
plt.xlabel('Sepal length')
plt.ylabel('Sepal width')
plt.xlim(xx.min(), xx.max())
plt.title('SVC with linear kernel')
로지스틱 회귀
기본적으로 로지스틱 회귀 모델은 감독 분류 알고리즘 제품군의 구성원 중 하나입니다. 로지스틱 회귀는 로지스틱 함수를 사용하여 확률을 추정하여 종속 변수와 독립 변수 간의 관계를 측정합니다.
여기서 종속 변수와 독립 변수에 대해 이야기하면 종속 변수는 우리가 예측할 대상 클래스 변수이고 다른 쪽에서 독립 변수는 대상 클래스를 예측하는 데 사용할 기능입니다.
로지스틱 회귀에서 확률을 추정하는 것은 이벤트의 발생 가능성을 예측하는 것을 의미합니다. 예를 들어, 상점 주인은 상점에 들어간 고객이 플레이 스테이션 (예 :)을 구매할지 여부를 예측하려고합니다. 고객의 많은 특징이있을 것입니다. 성별, 나이 등 상점 주인은 가능성 발생 가능성을 예측하기 위해 관찰 할 것입니다 (예 : 플레이 스테이션 구매 여부). 로지스틱 함수는 다양한 매개 변수를 사용하여 함수를 생성하는 데 사용되는 시그 모이 드 곡선입니다.
전제 조건
로지스틱 회귀를 사용하여 분류기를 빌드하기 전에 시스템에 Tkinter 패키지를 설치해야합니다. 다음에서 설치할 수 있습니다.https://docs.python.org/2/library/tkinter.html.
이제 아래 주어진 코드의 도움으로 로지스틱 회귀를 사용하여 분류기를 만들 수 있습니다.
먼저 몇 가지 패키지를 가져옵니다.
import numpy as np
from sklearn import linear_model
import matplotlib.pyplot as plt이제 다음과 같이 수행 할 수있는 샘플 데이터를 정의해야합니다.
X = np.array([[2, 4.8], [2.9, 4.7], [2.5, 5], [3.2, 5.5], [6, 5], [7.6, 4],
[3.2, 0.9], [2.9, 1.9],[2.4, 3.5], [0.5, 3.4], [1, 4], [0.9, 5.9]])
y = np.array([0, 0, 0, 1, 1, 1, 2, 2, 2, 3, 3, 3])다음으로 로지스틱 회귀 분류기를 생성해야합니다. 다음과 같이 수행 할 수 있습니다.
Classifier_LR = linear_model.LogisticRegression(solver = 'liblinear', C = 75)마지막으로이 분류기를 훈련시켜야합니다.
Classifier_LR.fit(X, y)이제 출력을 어떻게 시각화 할 수 있습니까? Logistic_visualize ()라는 함수를 생성하여 수행 할 수 있습니다.
Def Logistic_visualize(Classifier_LR, X, y):
min_x, max_x = X[:, 0].min() - 1.0, X[:, 0].max() + 1.0
min_y, max_y = X[:, 1].min() - 1.0, X[:, 1].max() + 1.0위의 라인에서 우리는 메쉬 그리드에서 사용할 최소값과 최대 값 X와 Y를 정의했습니다. 또한 메쉬 그리드를 플로팅하기위한 단계 크기를 정의합니다.
mesh_step_size = 0.02다음과 같이 X 및 Y 값의 메쉬 그리드를 정의하겠습니다.
x_vals, y_vals = np.meshgrid(np.arange(min_x, max_x, mesh_step_size),
np.arange(min_y, max_y, mesh_step_size))다음 코드의 도움으로 메쉬 그리드에서 분류기를 실행할 수 있습니다.
output = classifier.predict(np.c_[x_vals.ravel(), y_vals.ravel()])
output = output.reshape(x_vals.shape)
plt.figure()
plt.pcolormesh(x_vals, y_vals, output, cmap = plt.cm.gray)
plt.scatter(X[:, 0], X[:, 1], c = y, s = 75, edgecolors = 'black',
linewidth=1, cmap = plt.cm.Paired)다음 코드 줄은 플롯의 경계를 지정합니다.
plt.xlim(x_vals.min(), x_vals.max())
plt.ylim(y_vals.min(), y_vals.max())
plt.xticks((np.arange(int(X[:, 0].min() - 1), int(X[:, 0].max() + 1), 1.0)))
plt.yticks((np.arange(int(X[:, 1].min() - 1), int(X[:, 1].max() + 1), 1.0)))
plt.show()이제 코드를 실행하면 다음과 같은 로지스틱 회귀 분류 기가 출력됩니다.

의사 결정 트리 분류기
의사 결정 트리는 기본적으로 각 노드가 일부 기능 변수에 따라 관측 그룹을 분할하는 이진 트리 순서도입니다.
여기에서는 남성 또는 여성을 예측하기위한 의사 결정 트리 분류기를 구축하고 있습니다. 19 개의 샘플이있는 매우 작은 데이터 세트를 가져옵니다. 이 샘플은 '높이'와 '머리카락 길이'의 두 가지 특징으로 구성됩니다.
전제 조건
다음 분류기를 작성하려면 다음을 설치해야합니다. pydotplus 과 graphviz. 기본적으로 graphviz는 도트 파일을 사용하여 그래픽을 그리는 도구입니다.pydotplusGraphviz의 Dot 언어에 대한 모듈입니다. 패키지 관리자 또는 pip로 설치할 수 있습니다.
이제 다음 Python 코드를 사용하여 결정 트리 분류기를 만들 수 있습니다.
먼저 다음과 같이 중요한 라이브러리를 가져 오겠습니다.
import pydotplus
from sklearn import tree
from sklearn.datasets import load_iris
from sklearn.metrics import classification_report
from sklearn import cross_validation
import collections이제 다음과 같이 데이터 세트를 제공해야합니다.
X = [[165,19],[175,32],[136,35],[174,65],[141,28],[176,15],[131,32],
[166,6],[128,32],[179,10],[136,34],[186,2],[126,25],[176,28],[112,38],
[169,9],[171,36],[116,25],[196,25]]
Y = ['Man','Woman','Woman','Man','Woman','Man','Woman','Man','Woman',
'Man','Woman','Man','Woman','Woman','Woman','Man','Woman','Woman','Man']
data_feature_names = ['height','length of hair']
X_train, X_test, Y_train, Y_test = cross_validation.train_test_split
(X, Y, test_size=0.40, random_state=5)데이터 세트를 제공 한 후 다음과 같이 수행 할 수있는 모델을 적합해야합니다.
clf = tree.DecisionTreeClassifier()
clf = clf.fit(X,Y)다음 Python 코드를 사용하여 예측할 수 있습니다.
prediction = clf.predict([[133,37]])
print(prediction)다음 Python 코드를 사용하여 의사 결정 트리를 시각화 할 수 있습니다.
dot_data = tree.export_graphviz(clf,feature_names = data_feature_names,
out_file = None,filled = True,rounded = True)
graph = pydotplus.graph_from_dot_data(dot_data)
colors = ('orange', 'yellow')
edges = collections.defaultdict(list)
for edge in graph.get_edge_list():
edges[edge.get_source()].append(int(edge.get_destination()))
for edge in edges: edges[edge].sort()
for i in range(2):dest = graph.get_node(str(edges[edge][i]))[0]
dest.set_fillcolor(colors[i])
graph.write_png('Decisiontree16.png')위 코드에 대한 예측을 다음과 같이 제공합니다. [‘Woman’] 다음 결정 트리를 만듭니다.

예측에서 특성 값을 변경하여 테스트 할 수 있습니다.
랜덤 포레스트 분류기
앙상블 방법은 기계 학습 모델을 더 강력한 기계 학습 모델로 결합하는 방법입니다. 의사 결정 트리 모음 인 Random Forest도 그중 하나입니다. 예측력을 유지하면서 결과를 평균화하여 과적 합을 줄일 수 있기 때문에 단일 의사 결정 트리보다 낫습니다. 여기에서는 scikit learn cancer 데이터 셋에 랜덤 포레스트 모델을 구현할 것입니다.
필요한 패키지 가져 오기-
from sklearn.ensemble import RandomForestClassifier
from sklearn.model_selection import train_test_split
from sklearn.datasets import load_breast_cancer
cancer = load_breast_cancer()
import matplotlib.pyplot as plt
import numpy as np이제 다음과 같이 할 수있는 데이터 셋을 제공해야합니다.
cancer = load_breast_cancer()
X_train, X_test, y_train,
y_test = train_test_split(cancer.data, cancer.target, random_state = 0)데이터 세트를 제공 한 후 다음과 같이 수행 할 수있는 모델을 적합해야합니다.
forest = RandomForestClassifier(n_estimators = 50, random_state = 0)
forest.fit(X_train,y_train)이제 훈련 및 테스트 하위 집합에 대한 정확성을 얻습니다. 추정기 수를 늘리면 테스트 하위 집합의 정확도도 증가합니다.
print('Accuracy on the training subset:(:.3f)',format(forest.score(X_train,y_train)))
print('Accuracy on the training subset:(:.3f)',format(forest.score(X_test,y_test)))산출
Accuracy on the training subset:(:.3f) 1.0
Accuracy on the training subset:(:.3f) 0.965034965034965이제 의사 결정 트리처럼 임의의 포리스트에는 feature_importance결정 트리보다 기능 가중치에 대한 더 나은보기를 제공하는 모듈입니다. 다음과 같이 플롯하고 시각화 할 수 있습니다.
n_features = cancer.data.shape[1]
plt.barh(range(n_features),forest.feature_importances_, align='center')
plt.yticks(np.arange(n_features),cancer.feature_names)
plt.xlabel('Feature Importance')
plt.ylabel('Feature')
plt.show()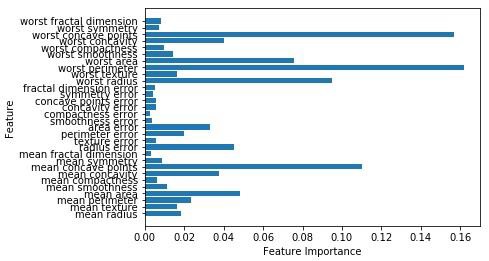
분류기의 성능
기계 학습 알고리즘을 구현 한 후 모델이 얼마나 효과적인지 알아 내야합니다. 효과를 측정하기위한 기준은 데이터 세트와 메트릭을 기반으로 할 수 있습니다. 다른 기계 학습 알고리즘을 평가하기 위해 다른 성능 메트릭을 사용할 수 있습니다. 예를 들어 분류기를 사용하여 서로 다른 객체의 이미지를 구분한다고 가정하면 평균 정확도, AUC 등과 같은 분류 성능 메트릭을 사용할 수 있습니다. 하나 또는 다른 의미에서 기계 학습 모델을 평가하기 위해 선택한 메트릭은 다음과 같습니다. 메트릭 선택이 기계 학습 알고리즘의 성능을 측정하고 비교하는 방법에 영향을 미치기 때문에 매우 중요합니다. 다음은 몇 가지 메트릭입니다.
혼동 매트릭스
기본적으로 출력이 둘 이상의 클래스 유형이 될 수있는 분류 문제에 사용됩니다. 분류기의 성능을 측정하는 가장 쉬운 방법입니다. 혼동 행렬은 기본적으로 "실제"와 "예측"이라는 두 차원이있는 테이블입니다. 두 차원 모두“참 양성 (TP)”,“참 음성 (TN)”,“거짓 양성 (FP)”,“거짓 음성 (FN)”을 갖습니다.
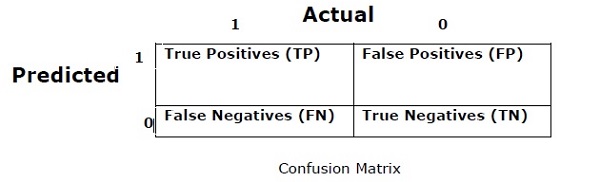
위의 혼동 행렬에서 1은 포지티브 클래스이고 0은 네거티브 클래스입니다.
다음은 혼동 행렬과 관련된 용어입니다.
True Positives − TP는 데이터 포인트의 실제 클래스가 1이고 예측도 1 인 경우입니다.
True Negatives − TN은 데이터 포인트의 실제 클래스가 0이고 예측도 0 인 경우입니다.
False Positives − FP는 데이터 포인트의 실제 클래스가 0이고 예측도 1 인 경우입니다.
False Negatives − FN은 데이터 포인트의 실제 클래스가 1이고 예측 값도 0 인 경우입니다.
정확성
혼동 행렬 자체는 성능 측정이 아니지만 거의 모든 성능 행렬이 혼동 행렬을 기반으로합니다. 그중 하나는 정확성입니다. 분류 문제에서는 모든 종류의 예측에 대해 모델이 만든 올바른 예측의 수로 정의 할 수 있습니다. 정확도를 계산하는 공식은 다음과 같습니다.
$$ Accuracy = \ frac {TP + TN} {TP + FP + FN + TN} $$
정도
주로 문서 검색에 사용됩니다. 반환 된 문서 중 올바른 수로 정의 할 수 있습니다. 다음은 정밀도를 계산하는 공식입니다-
$$ 정밀도 = \ frac {TP} {TP + FP} $$
회상 또는 감도
모델이 반환하는 긍정의 수로 정의 할 수 있습니다. 다음은 모델의 재현율 / 민감도를 계산하는 공식입니다.
$$ Recall = \ frac {TP} {TP + FN} $$
특성
모델이 반환하는 네거티브의 수로 정의 할 수 있습니다. 회상과는 정반대입니다. 다음은 모델의 특이성을 계산하는 공식입니다.
$$ Specificity = \ frac {TN} {TN + FP} $$
클래스 불균형 문제
클래스 불균형은 한 클래스에 속하는 관측치 수가 다른 클래스에 속하는 관측치보다 현저히 낮은 시나리오입니다. 예를 들어,이 문제는 희귀 질환, 은행의 사기 거래 등을 식별해야하는 시나리오에서 두드러집니다.
불균형 클래스의 예
불균형 클래스의 개념을 이해하기 위해 사기 탐지 데이터 세트의 예를 살펴 보겠습니다.
Total observations = 5000
Fraudulent Observations = 50
Non-Fraudulent Observations = 4950
Event Rate = 1%해결책
Balancing the classes’불균형 클래스에 대한 해결책으로 작용합니다. 계급 균형의 주된 목적은 소수 계급의 빈도를 늘리거나 대다수 계급의 빈도를 줄이는 것입니다. 다음은 불균형 클래스 문제를 해결하는 방법입니다.
재 샘플링
재 샘플링은 학습 세트와 테스트 세트 모두를 포함하여 샘플 데이터 세트를 재구성하는 데 사용되는 일련의 방법입니다. 모델의 정확도를 높이기 위해 리샘플링이 수행됩니다. 다음은 몇 가지 재 샘플링 기술입니다.
Random Under-Sampling−이 기법은 다수의 클래스 예를 무작위로 제거하여 클래스 분포의 균형을 맞추는 것을 목표로합니다. 이는 다수 및 소수 클래스 인스턴스가 균형을 이룰 때까지 수행됩니다.
Total observations = 5000
Fraudulent Observations = 50
Non-Fraudulent Observations = 4950
Event Rate = 1%이 경우, 우리는 사기가 아닌 인스턴스에서 교체하지 않고 10 % 샘플을 채취 한 다음 사기 인스턴스와 결합합니다.
무작위 언더 샘플링 후 사기가 아닌 관찰 = 4950의 10 % = 495
거짓 관찰과 결합한 후의 총 관찰 = 50 + 495 = 545
따라서 이제 언더 샘플링 후 새 데이터 세트의 이벤트 비율 = 9 %
이 기술의 주요 장점은 런타임을 줄이고 스토리지를 향상시킬 수 있다는 것입니다. 그러나 다른 한편으로는 유용한 정보를 버리고 훈련 데이터 샘플의 수를 줄일 수 있습니다.
Random Over-Sampling −이 기법은 복제를 통해 소수 클래스의 인스턴스 수를 늘려 클래스 분포의 균형을 맞추는 것을 목표로합니다.
Total observations = 5000
Fraudulent Observations = 50
Non-Fraudulent Observations = 4950
Event Rate = 1%50 개의 사기 관찰을 30 번 복제하는 경우 소수 클래스 관찰을 복제 한 후의 부정 관찰은 1500이됩니다. 그러면 오버 샘플링 후 새 데이터의 총 관찰은 4950 + 1500 = 6450이됩니다. 따라서 새 데이터 세트의 이벤트 비율은 1500/6450 = 23 %가됩니다.
이 방법의 가장 큰 장점은 유용한 정보가 손실되지 않는다는 것입니다. 그러나 반면에 소수 계급 이벤트를 복제하기 때문에 과적 합 가능성이 증가합니다.
앙상블 기법
이 방법론은 기본적으로 기존 분류 알고리즘을 수정하여 불균형 데이터 세트에 적합하게 만드는 데 사용됩니다. 이 접근 방식에서는 원본 데이터에서 여러 개의 2 단계 분류기를 구성한 다음 예측을 집계합니다. 랜덤 포레스트 분류기는 앙상블 기반 분류기의 예입니다.