빅 데이터 분석-통계적 방법
데이터를 분석 할 때 통계적 접근이 가능합니다. 기본 분석을 수행하는 데 필요한 기본 도구는 다음과 같습니다.
- 상관 분석
- 분산 분석
- 가설 검증
대규모 데이터 세트로 작업 할 때 상관 분석을 제외하고는 이러한 방법이 계산 집약적이지 않으므로 문제가 발생하지 않습니다. 이 경우 항상 샘플을 채취 할 수 있으며 결과는 견고해야합니다.
상관 분석
상관 관계 분석은 숫자 변수 간의 선형 관계를 찾습니다. 이것은 다른 상황에서 사용될 수 있습니다. 한 가지 일반적인 용도는 탐색 적 데이터 분석이며,이 책의 섹션 16.0.2에는이 접근 방식의 기본 예가 있습니다. 우선, 언급 된 예에서 사용 된 상관 관계 측정 항목은Pearson coefficient. 그러나 특이 치의 영향을받지 않는 또 다른 흥미로운 상관 관계 지표가 있습니다. 이 측정 항목을 spearman 상관 관계라고합니다.
그만큼 spearman correlation 메트릭은 Pearson 방법보다 특이 치의 존재에 더 강력하며 데이터가 정규 분포를 따르지 않을 때 숫자 변수 간의 선형 관계에 대한 더 나은 추정치를 제공합니다.
library(ggplot2)
# Select variables that are interesting to compare pearson and spearman
correlation methods.
x = diamonds[, c('x', 'y', 'z', 'price')]
# From the histograms we can expect differences in the correlations of both
metrics.
# In this case as the variables are clearly not normally distributed, the
spearman correlation
# is a better estimate of the linear relation among numeric variables.
par(mfrow = c(2,2))
colnm = names(x)
for(i in 1:4) {
hist(x[[i]], col = 'deepskyblue3', main = sprintf('Histogram of %s', colnm[i]))
}
par(mfrow = c(1,1))다음 그림의 히스토그램에서 두 메트릭의 상관 관계에서 차이를 예상 할 수 있습니다. 이 경우 변수가 명확하게 정규 분포를 따르지 않기 때문에 spearman 상관 관계는 숫자 변수 간의 선형 관계를 더 잘 추정합니다.
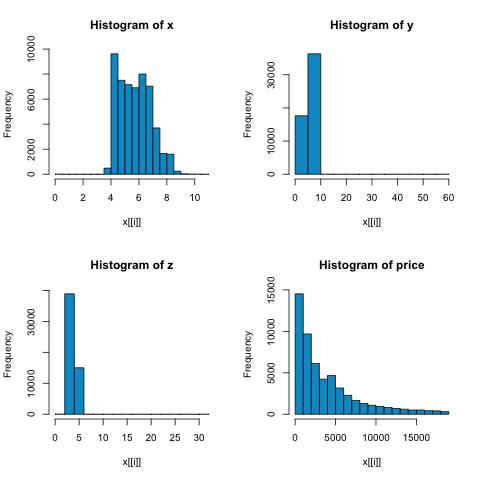
R의 상관 관계를 계산하려면 파일을 엽니 다. bda/part2/statistical_methods/correlation/correlation.R 이 코드 섹션이 있습니다.
## Correlation Matrix - Pearson and spearman
cor_pearson <- cor(x, method = 'pearson')
cor_spearman <- cor(x, method = 'spearman')
### Pearson Correlation
print(cor_pearson)
# x y z price
# x 1.0000000 0.9747015 0.9707718 0.8844352
# y 0.9747015 1.0000000 0.9520057 0.8654209
# z 0.9707718 0.9520057 1.0000000 0.8612494
# price 0.8844352 0.8654209 0.8612494 1.0000000
### Spearman Correlation
print(cor_spearman)
# x y z price
# x 1.0000000 0.9978949 0.9873553 0.9631961
# y 0.9978949 1.0000000 0.9870675 0.9627188
# z 0.9873553 0.9870675 1.0000000 0.9572323
# price 0.9631961 0.9627188 0.9572323 1.0000000카이 제곱 검정
카이 제곱 테스트를 사용하면 두 개의 랜덤 변수가 독립적인지 테스트 할 수 있습니다. 이는 각 변수의 확률 분포가 다른 변수에 영향을 미치지 않음을 의미합니다. R에서 테스트를 평가하려면 먼저 분할 테이블을 만든 다음 테이블을chisq.test R 함수.
예를 들어, 다이아몬드 데이터 셋의 cut과 color 변수 사이에 연관성이 있는지 확인해 보겠습니다. 테스트는 공식적으로 다음과 같이 정의됩니다.
- H0 : 가변 컷과 다이아몬드는 독립적입니다.
- H1 : 가변 컷과 다이아몬드는 독립적이지 않습니다.
이름으로이 두 변수간에 관계가 있다고 가정하지만 테스트는이 결과가 얼마나 중요한지 여부를 나타내는 객관적인 "규칙"을 제공 할 수 있습니다.
다음 코드 스 니펫에서 테스트의 p- 값은 2.2e-16이고 실제적으로는 거의 0입니다. 그런 다음 테스트를 실행 한 후Monte Carlo simulation, 우리는 p- 값이 0.0004998로 여전히 임계 값 0.05보다 훨씬 낮다는 것을 발견했습니다. 이 결과는 귀무 가설 (H0)을 기각한다는 것을 의미하므로 변수가cut 과 color 독립적이지 않습니다.
library(ggplot2)
# Use the table function to compute the contingency table
tbl = table(diamonds$cut, diamonds$color)
tbl
# D E F G H I J
# Fair 163 224 312 314 303 175 119
# Good 662 933 909 871 702 522 307
# Very Good 1513 2400 2164 2299 1824 1204 678
# Premium 1603 2337 2331 2924 2360 1428 808
# Ideal 2834 3903 3826 4884 3115 2093 896
# In order to run the test we just use the chisq.test function.
chisq.test(tbl)
# Pearson’s Chi-squared test
# data: tbl
# X-squared = 310.32, df = 24, p-value < 2.2e-16
# It is also possible to compute the p-values using a monte-carlo simulation
# It's needed to add the simulate.p.value = TRUE flag and the amount of
simulations
chisq.test(tbl, simulate.p.value = TRUE, B = 2000)
# Pearson’s Chi-squared test with simulated p-value (based on 2000 replicates)
# data: tbl
# X-squared = 310.32, df = NA, p-value = 0.0004998T- 테스트
아이디어 t-test명목 변수의 서로 다른 그룹간에 숫자 변수 분포에 차이가 있는지 평가하는 것입니다. 이를 입증하기 위해 요인 변수 컷의 공정 및 이상 수준 수준을 선택한 다음 두 그룹간에 숫자 변수 값을 비교합니다.
data = diamonds[diamonds$cut %in% c('Fair', 'Ideal'), ]
data$cut = droplevels.factor(data$cut) # Drop levels that aren’t used from the
cut variable
df1 = data[, c('cut', 'price')]
# We can see the price means are different for each group
tapply(df1$price, df1$cut, mean)
# Fair Ideal
# 4358.758 3457.542t- 검정은 R에서 t.test함수. t.test에 대한 공식 인터페이스는 그것을 사용하는 가장 간단한 방법이며, 아이디어는 숫자 변수가 그룹 변수로 설명된다는 것입니다.
예를 들면 : t.test(numeric_variable ~ group_variable, data = data). 이전 예에서numeric_variable 이다 price 그리고 group_variable 이다 cut.
통계적 관점에서 두 그룹 간의 숫자 변수 분포에 차이가 있는지 테스트하고 있습니다. 공식적으로 가설 검정은 귀무 (H0) 가설과 대립 가설 (H1)로 설명됩니다.
H0 : 공정한 그룹과 이상적인 그룹 간의 가격 변수 분포에는 차이가 없습니다.
H1 공정한 그룹과 이상적인 그룹 간의 가격 변수 분포에 차이가 있습니다.
다음은 다음 코드로 R에서 구현할 수 있습니다.
t.test(price ~ cut, data = data)
# Welch Two Sample t-test
#
# data: price by cut
# t = 9.7484, df = 1894.8, p-value < 2.2e-16
# alternative hypothesis: true difference in means is not equal to 0
# 95 percent confidence interval:
# 719.9065 1082.5251
# sample estimates:
# mean in group Fair mean in group Ideal
# 4358.758 3457.542
# Another way to validate the previous results is to just plot the
distributions using a box-plot
plot(price ~ cut, data = data, ylim = c(0,12000),
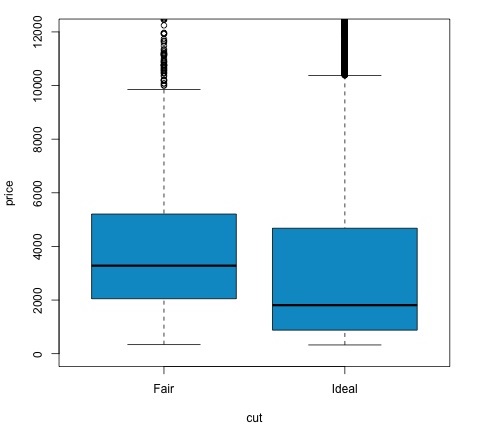
col = 'deepskyblue3')p- 값이 0.05 미만인지 확인하여 검정 결과를 분석 할 수 있습니다. 이 경우 대립 가설을 유지합니다. 이는 컷 팩터의 두 수준간에 가격 차이를 발견했음을 의미합니다. 레벨의 이름으로 우리는이 결과를 예상했을 것이지만 실패 그룹의 평균 가격이 이상적인 그룹보다 높을 것이라고는 예상하지 못했을 것입니다. 각 요인의 평균을 비교하여이를 알 수 있습니다.
그만큼 plot명령은 가격과 컷 변수 사이의 관계를 보여주는 그래프를 생성합니다. 이것은 상자 그림입니다. 섹션 16.0.1에서이 그림을 다루었지만 기본적으로 분석중인 두 가지 수준의 인하에 대한 가격 변수의 분포를 보여줍니다.
분산 분석
분산 분석 (ANOVA)은 각 그룹의 평균과 분산을 비교하여 그룹 분포 간의 차이를 분석하는 데 사용되는 통계 모델이며, 모델은 Ronald Fisher가 개발했습니다. ANOVA는 여러 그룹의 평균이 같은지 여부에 대한 통계 테스트를 제공하므로 t- 테스트를 세 개 이상의 그룹으로 일반화합니다.
ANOVA는 통계적 유의성에 대해 세 개 이상의 그룹을 비교하는 데 유용합니다. 여러 개의 2- 표본 t- 검정을 수행하면 통계 제 1 종 오류를 범할 가능성이 증가하기 때문입니다.
수학적 설명을 제공하는 측면에서 테스트를 이해하려면 다음이 필요합니다.
x ij = x + (x i − x) + (x ij − x)
이것은 다음 모델로 이어집니다-
x ij = μ + α i + ∈ ij
여기서 μ는 총 평균이고 α i 는 i 번째 그룹 평균입니다. 오차항 ∈ ij 는 정규 분포의 iid라고 가정합니다. 검정의 귀무 가설은 다음과 같습니다.
α 1 = α 2 =… = α k
테스트 통계를 계산할 때 두 값을 계산해야합니다.
- 그룹 차이에 대한 제곱합 −
$$ SSD_B = \ sum_ {i} ^ {k} \ sum_ {j} ^ {n} (\ bar {x _ {\ bar {i}}}-\ bar {x}) ^ 2 $$
- 그룹 내 제곱의 합
$$ SSD_W = \ sum_ {i} ^ {k} \ sum_ {j} ^ {n} (\ bar {x _ {\ bar {ij}}}-\ bar {x _ {\ bar {i}}}) ^ 2 $$
여기서 SSD B 의 자유도는 k-1이고 SSD W 의 자유도는 N-k입니다. 그런 다음 각 메트릭에 대한 평균 제곱 차이를 정의 할 수 있습니다.
MS B = SSD B / (k-1)
MS w = SSD w / (N-k)
마지막으로 ANOVA의 검정 통계량은 위의 두 양의 비율로 정의됩니다.
F = MS B / MS w
k-1 및 N-k 자유도를 갖는 F- 분포를 따릅니다 . 귀무 가설이 참이면 F는 1에 가까울 것입니다. 그렇지 않으면 그룹 간 평균 제곱 MSB가 클 가능성이 높아 F 값이 커집니다.
기본적으로 ANOVA는 총 분산의 두 소스를 조사하고 어떤 부분이 더 많은 기여를하는지 확인합니다. 이것이 그룹 평균을 비교하려는 의도이지만 분산 분석이라고하는 이유입니다.
통계 계산 측면에서 실제로 R에서 수행하는 것은 다소 간단합니다. 다음 예제는 수행 방법을 보여주고 결과를 플로팅합니다.
library(ggplot2)
# We will be using the mtcars dataset
head(mtcars)
# mpg cyl disp hp drat wt qsec vs am gear carb
# Mazda RX4 21.0 6 160 110 3.90 2.620 16.46 0 1 4 4
# Mazda RX4 Wag 21.0 6 160 110 3.90 2.875 17.02 0 1 4 4
# Datsun 710 22.8 4 108 93 3.85 2.320 18.61 1 1 4 1
# Hornet 4 Drive 21.4 6 258 110 3.08 3.215 19.44 1 0 3 1
# Hornet Sportabout 18.7 8 360 175 3.15 3.440 17.02 0 0 3 2
# Valiant 18.1 6 225 105 2.76 3.460 20.22 1 0 3 1
# Let's see if there are differences between the groups of cyl in the mpg variable.
data = mtcars[, c('mpg', 'cyl')]
fit = lm(mpg ~ cyl, data = mtcars)
anova(fit)
# Analysis of Variance Table
# Response: mpg
# Df Sum Sq Mean Sq F value Pr(>F)
# cyl 1 817.71 817.71 79.561 6.113e-10 ***
# Residuals 30 308.33 10.28
# Signif. codes: 0 *** 0.001 ** 0.01 * 0.05 .
# Plot the distribution
plot(mpg ~ as.factor(cyl), data = mtcars, col = 'deepskyblue3')코드는 다음 출력을 생성합니다.

예제에서 얻은 p- 값은 0.05보다 훨씬 작으므로 R은이를 나타내는 기호 '***'를 반환합니다. 그것은 우리가 귀무 가설을 기각하고 다른 그룹의 mpg 평균 사이의 차이를 발견한다는 것을 의미합니다.cyl 변하기 쉬운.