Análise de Big Data - Métodos Estatísticos
Ao analisar os dados, é possível ter uma abordagem estatística. As ferramentas básicas necessárias para realizar a análise básica são -
- Análise de correlação
- Análise de variação
- Testando hipóteses
Ao trabalhar com grandes conjuntos de dados, isso não envolve um problema, pois esses métodos não são computacionalmente intensivos, com exceção da análise de correlação. Nesse caso, sempre é possível tirar uma amostra e os resultados devem ser robustos.
Análise de correlação
A Análise de Correlação procura encontrar relações lineares entre variáveis numéricas. Isso pode ser útil em diferentes circunstâncias. Um uso comum é a análise exploratória de dados, na seção 16.0.2 do livro há um exemplo básico dessa abordagem. Em primeiro lugar, a métrica de correlação usada no exemplo mencionado é baseada naPearson coefficient. No entanto, há outra métrica interessante de correlação que não é afetada por outliers. Essa métrica é chamada de correlação spearman.
o spearman correlation métrica é mais robusta à presença de outliers do que o método de Pearson e fornece melhores estimativas das relações lineares entre as variáveis numéricas quando os dados não são normalmente distribuídos.
library(ggplot2)
# Select variables that are interesting to compare pearson and spearman
correlation methods.
x = diamonds[, c('x', 'y', 'z', 'price')]
# From the histograms we can expect differences in the correlations of both
metrics.
# In this case as the variables are clearly not normally distributed, the
spearman correlation
# is a better estimate of the linear relation among numeric variables.
par(mfrow = c(2,2))
colnm = names(x)
for(i in 1:4) {
hist(x[[i]], col = 'deepskyblue3', main = sprintf('Histogram of %s', colnm[i]))
}
par(mfrow = c(1,1))A partir dos histogramas da figura a seguir, podemos esperar diferenças nas correlações de ambas as métricas. Nesse caso, como as variáveis claramente não têm distribuição normal, a correlação de Spearman é uma estimativa melhor da relação linear entre as variáveis numéricas.
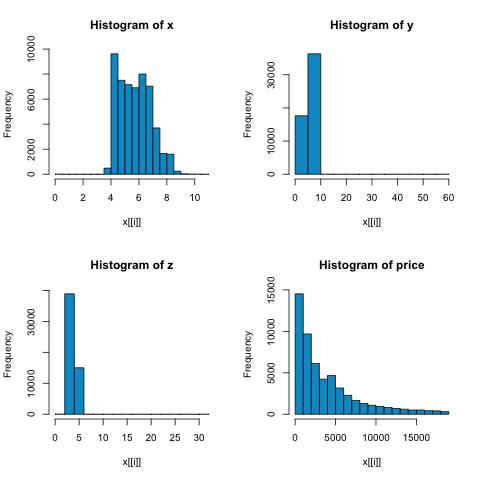
Para calcular a correlação em R, abra o arquivo bda/part2/statistical_methods/correlation/correlation.R que tem esta seção de código.
## Correlation Matrix - Pearson and spearman
cor_pearson <- cor(x, method = 'pearson')
cor_spearman <- cor(x, method = 'spearman')
### Pearson Correlation
print(cor_pearson)
# x y z price
# x 1.0000000 0.9747015 0.9707718 0.8844352
# y 0.9747015 1.0000000 0.9520057 0.8654209
# z 0.9707718 0.9520057 1.0000000 0.8612494
# price 0.8844352 0.8654209 0.8612494 1.0000000
### Spearman Correlation
print(cor_spearman)
# x y z price
# x 1.0000000 0.9978949 0.9873553 0.9631961
# y 0.9978949 1.0000000 0.9870675 0.9627188
# z 0.9873553 0.9870675 1.0000000 0.9572323
# price 0.9631961 0.9627188 0.9572323 1.0000000Teste Qui-quadrado
O teste qui-quadrado nos permite testar se duas variáveis aleatórias são independentes. Isso significa que a distribuição de probabilidade de cada variável não influencia a outra. Para avaliar o teste em R, precisamos primeiro criar uma tabela de contingência e, em seguida, passá-la para ochisq.test R função.
Por exemplo, vamos verificar se existe uma associação entre as variáveis: corte e cor do conjunto de dados de diamantes. O teste é formalmente definido como -
- H0: O corte variável e o diamante são independentes
- H1: O corte variável e o diamante não são independentes
Suporíamos que existe uma relação entre essas duas variáveis por seus nomes, mas o teste pode fornecer uma "regra" objetiva dizendo o quão significativo é esse resultado ou não.
No fragmento de código a seguir, descobrimos que o valor p do teste é 2,2e-16, isso é quase zero em termos práticos. Então, depois de executar o teste, fazer umMonte Carlo simulation, descobrimos que o valor p é 0,0004998, que ainda é bastante inferior ao limite 0,05. Este resultado significa que rejeitamos a hipótese nula (H0), então acreditamos nas variáveiscut e color não são independentes.
library(ggplot2)
# Use the table function to compute the contingency table
tbl = table(diamonds$cut, diamonds$color)
tbl
# D E F G H I J
# Fair 163 224 312 314 303 175 119
# Good 662 933 909 871 702 522 307
# Very Good 1513 2400 2164 2299 1824 1204 678
# Premium 1603 2337 2331 2924 2360 1428 808
# Ideal 2834 3903 3826 4884 3115 2093 896
# In order to run the test we just use the chisq.test function.
chisq.test(tbl)
# Pearson’s Chi-squared test
# data: tbl
# X-squared = 310.32, df = 24, p-value < 2.2e-16
# It is also possible to compute the p-values using a monte-carlo simulation
# It's needed to add the simulate.p.value = TRUE flag and the amount of
simulations
chisq.test(tbl, simulate.p.value = TRUE, B = 2000)
# Pearson’s Chi-squared test with simulated p-value (based on 2000 replicates)
# data: tbl
# X-squared = 310.32, df = NA, p-value = 0.0004998Teste t
A ideia de t-testé avaliar se existem diferenças na distribuição de uma variável numérica # entre diferentes grupos de uma variável nominal. Para demonstrar isso, selecionarei os níveis dos níveis Justo e Ideal do fator de corte variável, em seguida, compararemos os valores de uma variável numérica entre esses dois grupos.
data = diamonds[diamonds$cut %in% c('Fair', 'Ideal'), ]
data$cut = droplevels.factor(data$cut) # Drop levels that aren’t used from the
cut variable
df1 = data[, c('cut', 'price')]
# We can see the price means are different for each group
tapply(df1$price, df1$cut, mean)
# Fair Ideal
# 4358.758 3457.542Os testes t são implementados em R com o t.testfunção. A interface da fórmula para t.test é a maneira mais simples de usá-la, a ideia é que uma variável numérica é explicada por uma variável de grupo.
Por exemplo: t.test(numeric_variable ~ group_variable, data = data). No exemplo anterior, onumeric_variable é price e a group_variable é cut.
Do ponto de vista estatístico, estamos testando se há diferenças nas distribuições da variável numérica entre dois grupos. Formalmente, o teste de hipótese é descrito com uma hipótese nula (H0) e uma hipótese alternativa (H1).
H0: Não há diferenças nas distribuições da variável de preço entre os grupos Justo e Ideal
H1 Existem diferenças nas distribuições da variável preço entre os grupos Justo e Ideal
O seguinte pode ser implementado em R com o seguinte código -
t.test(price ~ cut, data = data)
# Welch Two Sample t-test
#
# data: price by cut
# t = 9.7484, df = 1894.8, p-value < 2.2e-16
# alternative hypothesis: true difference in means is not equal to 0
# 95 percent confidence interval:
# 719.9065 1082.5251
# sample estimates:
# mean in group Fair mean in group Ideal
# 4358.758 3457.542
# Another way to validate the previous results is to just plot the
distributions using a box-plot
plot(price ~ cut, data = data, ylim = c(0,12000),
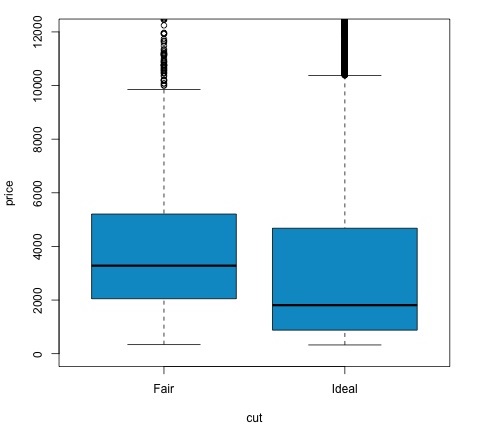
col = 'deepskyblue3')Podemos analisar o resultado do teste verificando se o valor p é menor que 0,05. Se for esse o caso, mantemos a hipótese alternativa. Isso significa que encontramos diferenças de preço entre os dois níveis do fator de corte. Pelos nomes dos níveis, esperaríamos esse resultado, mas não esperaríamos que o preço médio no grupo Fail fosse maior do que no grupo Ideal. Podemos ver isso comparando as médias de cada fator.
o plotcomando produz um gráfico que mostra a relação entre o preço e a variável de corte. É um enredo de caixa; cobrimos esse gráfico na seção 16.0.1, mas basicamente mostra a distribuição da variável de preço para os dois níveis de corte que estamos analisando.
Análise de variação
A Análise de Variância (ANOVA) é um modelo estatístico utilizado para analisar as diferenças entre a distribuição dos grupos, comparando a média e a variância de cada grupo, o modelo foi desenvolvido por Ronald Fisher. A ANOVA fornece um teste estatístico para determinar se as médias de vários grupos são iguais ou não e, portanto, generaliza o teste t para mais de dois grupos.
ANOVAs são úteis para comparar três ou mais grupos para significância estatística porque fazer vários testes t de duas amostras resultaria em uma chance maior de cometer um erro estatístico tipo I.
Em termos de fornecer uma explicação matemática, o seguinte é necessário para entender o teste.
x ij = x + (x i - x) + (x ij - x)
Isso leva ao seguinte modelo -
x ij = μ + α i + ∈ ij
onde μ é a grande média e α i é a i-ésima média do grupo. O termo de erro ∈ ij é assumido como sendo iid de uma distribuição normal. A hipótese nula do teste é que -
α 1 = α 2 =… = α k
Em termos de cálculo da estatística de teste, precisamos calcular dois valores -
- Soma dos quadrados para diferença entre os grupos -
$$ SSD_B = \ sum_ {i} ^ {k} \ sum_ {j} ^ {n} (\ bar {x _ {\ bar {i}}} - \ bar {x}) ^ 2 $$
- Soma dos quadrados dentro dos grupos
$$ SSD_W = \ sum_ {i} ^ {k} \ sum_ {j} ^ {n} (\ bar {x _ {\ bar {ij}}} - \ bar {x _ {\ bar {i}}}) ^ 2 $$
onde SSD B tem um grau de liberdade de k − 1 e SSD W tem um grau de liberdade de N − k. Em seguida, podemos definir as diferenças médias quadradas para cada métrica.
MS B = SSD B / (k - 1)
MS w = SSD w / (N - k)
Finalmente, a estatística de teste em ANOVA é definida como a razão das duas quantidades acima
F = MS B / MS w
que segue uma distribuição F com k − 1 e N − k graus de liberdade. Se a hipótese nula for verdadeira, F provavelmente seria próximo de 1. Caso contrário, o MSB do quadrado médio entre os grupos provavelmente será grande, o que resulta em um grande valor de F.
Basicamente, ANOVA examina as duas fontes da variância total e vê qual parte contribui mais. É por isso que é chamada de análise de variância, embora a intenção seja comparar as médias dos grupos.
Em termos de cálculo da estatística, é realmente bastante simples de fazer em R. O exemplo a seguir demonstrará como isso é feito e representará graficamente os resultados.
library(ggplot2)
# We will be using the mtcars dataset
head(mtcars)
# mpg cyl disp hp drat wt qsec vs am gear carb
# Mazda RX4 21.0 6 160 110 3.90 2.620 16.46 0 1 4 4
# Mazda RX4 Wag 21.0 6 160 110 3.90 2.875 17.02 0 1 4 4
# Datsun 710 22.8 4 108 93 3.85 2.320 18.61 1 1 4 1
# Hornet 4 Drive 21.4 6 258 110 3.08 3.215 19.44 1 0 3 1
# Hornet Sportabout 18.7 8 360 175 3.15 3.440 17.02 0 0 3 2
# Valiant 18.1 6 225 105 2.76 3.460 20.22 1 0 3 1
# Let's see if there are differences between the groups of cyl in the mpg variable.
data = mtcars[, c('mpg', 'cyl')]
fit = lm(mpg ~ cyl, data = mtcars)
anova(fit)
# Analysis of Variance Table
# Response: mpg
# Df Sum Sq Mean Sq F value Pr(>F)
# cyl 1 817.71 817.71 79.561 6.113e-10 ***
# Residuals 30 308.33 10.28
# Signif. codes: 0 *** 0.001 ** 0.01 * 0.05 .
# Plot the distribution
plot(mpg ~ as.factor(cyl), data = mtcars, col = 'deepskyblue3')O código produzirá a seguinte saída -

O valor de p obtido no exemplo é significativamente menor do que 0,05, então R retorna o símbolo '***' para denotar isso. Isso significa que rejeitamos a hipótese nula e que encontramos diferenças entre as médias de mpg entre os diferentes grupos docyl variável.