R - Nieliniowy najmniejszy kwadrat
Podczas modelowania rzeczywistych danych do analizy regresji obserwujemy, że rzadko zdarza się, że równanie modelu jest równaniem liniowym dającym liniowy wykres. W większości przypadków równanie modelu danych ze świata rzeczywistego obejmuje funkcje matematyczne wyższego stopnia, takie jak wykładnik 3 lub funkcja grzechu. W takim scenariuszu wykres modelu przedstawia krzywą, a nie linię. Celem zarówno regresji liniowej, jak i nieliniowej jest dostosowanie wartości parametrów modelu w celu znalezienia linii lub krzywej, która jest najbliższa danym. Po znalezieniu tych wartości będziemy w stanie oszacować zmienną odpowiedzi z dobrą dokładnością.
W regresji metodą najmniejszych kwadratów ustalamy model regresji, w którym suma kwadratów odległości pionowych różnych punktów z krzywej regresji jest zminimalizowana. Zwykle zaczynamy od zdefiniowanego modelu i przyjmujemy pewne wartości współczynników. Następnie stosujemynls() funkcji R, aby uzyskać dokładniejsze wartości wraz z przedziałami ufności.
Składnia
Podstawowa składnia tworzenia nieliniowego testu najmniejszych kwadratów w R to -
nls(formula, data, start)Poniżej znajduje się opis użytych parametrów -
formula jest nieliniową formułą modelu zawierającą zmienne i parametry.
data to ramka danych używana do oceny zmiennych w formule.
start jest nazwaną listą lub nazwanym wektorem liczbowym początkowych oszacowań.
Przykład
Rozważymy model nieliniowy przy założeniu początkowych wartości jego współczynników. Następnie zobaczymy, jakie są przedziały ufności tych założonych wartości, abyśmy mogli ocenić, jak dobrze te wartości pasują do modelu.
Rozważmy więc poniższe równanie w tym celu -
a = b1*x^2+b2Załóżmy, że początkowe współczynniki wynoszą 1 i 3 i dopasujmy te wartości do funkcji nls ().
xvalues <- c(1.6,2.1,2,2.23,3.71,3.25,3.4,3.86,1.19,2.21)
yvalues <- c(5.19,7.43,6.94,8.11,18.75,14.88,16.06,19.12,3.21,7.58)
# Give the chart file a name.
png(file = "nls.png")
# Plot these values.
plot(xvalues,yvalues)
# Take the assumed values and fit into the model.
model <- nls(yvalues ~ b1*xvalues^2+b2,start = list(b1 = 1,b2 = 3))
# Plot the chart with new data by fitting it to a prediction from 100 data points.
new.data <- data.frame(xvalues = seq(min(xvalues),max(xvalues),len = 100))
lines(new.data$xvalues,predict(model,newdata = new.data))
# Save the file.
dev.off()
# Get the sum of the squared residuals.
print(sum(resid(model)^2))
# Get the confidence intervals on the chosen values of the coefficients.
print(confint(model))Kiedy wykonujemy powyższy kod, daje on następujący wynik -
[1] 1.081935
Waiting for profiling to be done...
2.5% 97.5%
b1 1.137708 1.253135
b2 1.497364 2.496484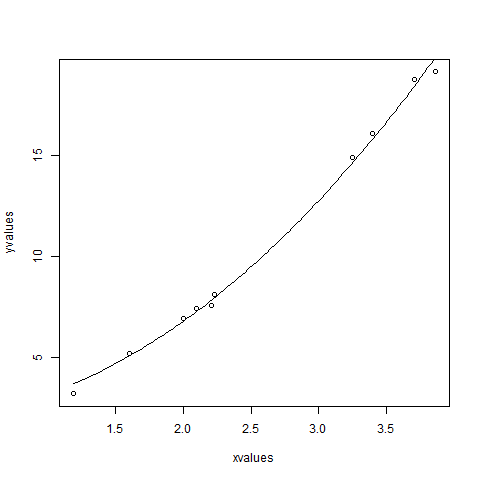
Możemy wywnioskować, że wartość b1 jest bliższa 1, podczas gdy wartość b2 jest bliższa 2, a nie 3.